Scilab 6.0.1での結果です。
FIFFファイルの扱い
Fiff2MatFileを使ってFIFFファイルを読む
Scilab 6.0.0ではMATLABのMATファイルがかなり読めるようになりましたが、
1)単精度浮動小数点singleが読めない
2)文字列のセル構造が大きいと読めない
3)文字列の空白が0x20でなく、0x00だと読み込みを中止する
といった問題があり、そのままでは読み込めません
そこでFIFFファイルをMATファイルに変換するFiff2MatFileにfor Scilabという項目を設け、
1)単精度浮動小数点singleは倍精度浮動小数点doubleに
2)文字列のセルはセルをやめてコロンをつけて文字列に
3)0x00の空白文字は削除する
機能を設けました。ここをクリックすればScilabのloadmatfile関数でMATファイルに変換したFIFFファイルを読むことができます。
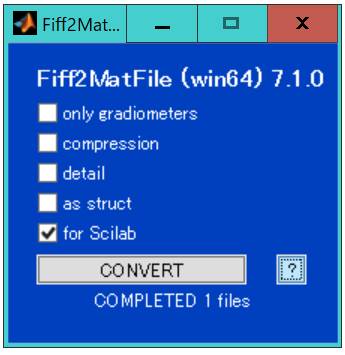
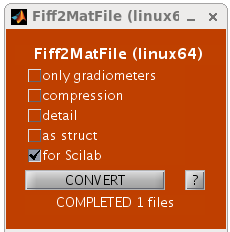
64bit
Windowsと64bit Linuxのみに対応しています。
64bit
Windows版はhttp://meg.aalip.jp/freeware/resource/Fiff2MatFile5/Fiff2MatFile_win64_pkg.exe
64bit
Linux版はhttp://meg.aalip.jp/freeware/resource/Fiff2MatFile5/Fiff2MatFile_lnx64_pkg.zip
からdownloadしてください。
また別途MATLAB Compiler Runtime v81 (R2013a版)のインストールが必要となります。
64bit
Linux版ではFiff2MatFile_lnx64のあるフォルダに移動しterminal上で
./run_Fiff2MatFile_lnx64.sh
/usr/local/MATLAB/MATLAB_Compiler_Runtime/v81
などとする必要があります。
体性感覚誘発磁場データ
右正中神経電気刺激の体性感覚誘発波形のFIFFファイルを読み込みます。そこそこ読み込みに時間がかかります。
[filename,pathname]=uigetfile('*.mat');
loadfile=pathname+'\'+filename;
// MATLABだとloadfile=[pathname,filename];
loadmatfile(loadfile);
変数ブラウザに変数が読み込まれます。
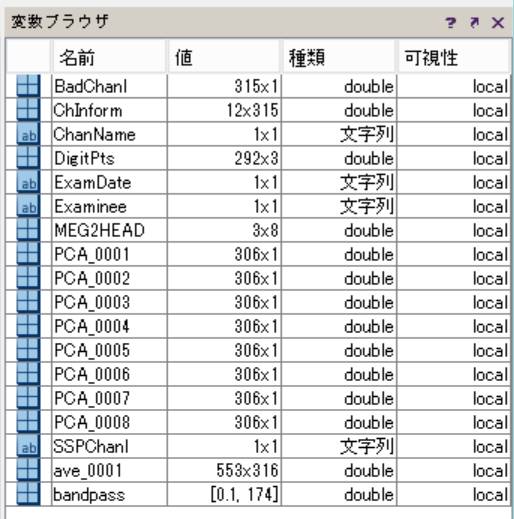
チャンネル名を表示させます。
-->
ChanName
ChanName =
MEG 0113:MEG 0112:MEG 0111:MEG 0122:MEG
0123:MEG 0121:MEG 0132:MEG 0133:MEG 0131:
MEG 0143:MEG 0142:MEG 0141:MEG 0213:MEG
0212:MEG 0211:MEG 0222:MEG 0223:MEG 0221:
MEG 0232:MEG 0233:MEG 0231:MEG 0243:MEG
0242:MEG 0241:MEG 0313:MEG 0312:MEG 0311:
MEG 0322:MEG 0323:MEG 0321:MEG 0333:MEG
0332:MEG 0331:MEG 0343:MEG 0342:MEG 0341:
中略
MEG 2442:MEG 2443:MEG 2441:MEG 2512:MEG
2513:MEG 2511:MEG 2522:MEG 2523:MEG 2521:
MEG 2533:MEG 2532:MEG 2531:MEG 2543:MEG
2542:MEG 2541:MEG 2612:MEG 2613:MEG 2611:
MEG 2623:MEG 2622:MEG 2621:MEG 2633:MEG
2632:MEG 2631:MEG 2642:MEG 2643:MEG 2641:
STI 001:STI 002:STI 003:STI 004:STI
005:STI 006:STI 014:STI 015:STI 016
loadmatfileはMATファイルで保存されている長い文字列を読むのに時間がかかるようです。セル構造に変換します。
st=':'+ChanName+':';
k=strindex(st,':');
n=length(k)-1;
name=cell(1,n);
for m=1:n
name{1,m}=part(st,(k(m)+1):(k(m+1)-1));//文字列の切り出しはpart関数を使う
end
ChanName=name;
SSPChanlも同様にセル構造に変換します。
st=':'+SSPChanl+':';
k=strindex(st,':');
n=length(k)-1;
name=cell(1,n);
for m=1:n
name{1,m}=part(st,(k(m)+1):(k(m+1)-1));//文字列の切り出しはpart関数を使う
end
SSPChanl=name;
ScilabのDATファイルとして保存します。MATファイルからDATファイルにすると文字列の読み込みは格段に高速化されるようです。
save('SEF_median_ave.dat','BadChanl','ChInform','ChanName','DigitPts','ExamDate','Examinee',...
'MEG2HEAD','PCA_0001','PCA_0002','PCA_0003','PCA_0004','PCA_0005','PCA_0006','PCA_0007',...
'PCA_0008','SSPChanl','ave_0001','bandpass');
clear();
load('SEF_median_ave.dat');//MATファイルと違って読み込みは速いです。
ChanNameからチャンネルの分別をします。
GRA=[];MAG=[];STI=[];
for n=1:length(ChanName)
if
strindex(ChanName{n},'MEG')==1
if part(ChanName{n},$)=='1'//最後が1だとmagnetometer
MAG=[MAG,n];
else
GRA=[GRA,n];
end
elseif
strindex(ChanName{n},'STI')==1
STI=[STI,n];
end
end
planar
gradiometerのみ波形を表示します。
hf=figure('figure_name','SEF of right median nerve');
hf.backgroundcolor=[1,1,1];
time=ave_0001(:,$);
gra=ave_0001(:,GRA);
plot(time,gra);//h=plotとは出力できず
p=get("hdl");//MATLABだとp=gco ただしpolylineの集合体も1つのchildrenとして扱う
for n=1:length(p.children)
set(p.children(n),'foreground',2);//青に
end
set(gca(),'tight_limits',["on","off","off"]);//x軸のみtight
title('planar gradiometers');
xlabel('msec');
ylabel('fT/cm')
xgrid([0,5,-1]);//数位はindex colorの色の順番
結果です。

そもそも色の番号はどうなってるのか?
編集→図のプロパティを選択します。
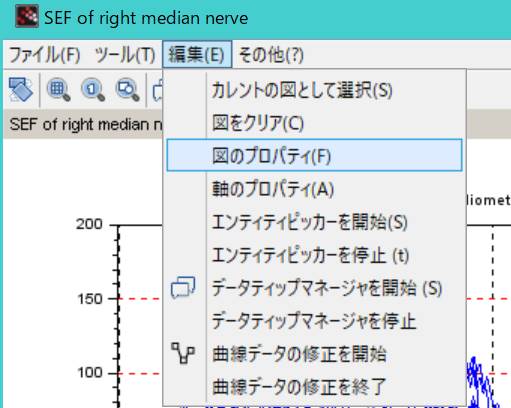
Colormapを選択すると、1〜38番の色が指定してあります。

マグネトメータをセンサ座標から頭座標に変換させ、Polhemusでプロットした点と一緒に表示させます。
plot3dは曲面、param3dは曲線を描くのに使うようです。
hf=figure('backgroundcolor',[1,1,1]);
ch=ChInform(:,MAG);
w=0.014;//センサの幅
P=MEG2HEAD(1:3,1:3)*ch(1:3,:)+MEG2HEAD(1:3,4)*ones(1,length(MAG));
X=w*MEG2HEAD(1:3,1:3)*ch(4:6,:);
Y=w*MEG2HEAD(1:3,1:3)*ch(7:9,:);
Q=zeros(3,102,4);
Q(:,:,1)=P+X+Y;
Q(:,:,2)=P+X-Y;
Q(:,:,3)=P-X-Y;
Q(:,:,4)=P-X+Y;
Qx=squeeze(Q(1,:,:));
Qy=squeeze(Q(2,:,:));
Qz=squeeze(Q(3,:,:));
plot3d(Qx',Qy',Qz');
h=get('hdl');//typeはFac3dらしい get(h,'type')
set(h,'color_mode',4,'hiddencolor',2);
param3d(DigitPts(:,1),DigitPts(:,2),DigitPts(:,3));
h=get('hdl');
set(h,'line_mode','off','mark_size',2,'mark_style',6,'mark_foreground',5,'mark_background',6);
set(gca(),'tight_limits',["on","on","on"],'isoview','on');
set(gca(),'rotation_angles',[60,30],'box','off','grid',[0,0,0]);
結果です。右ドラッグで任意の角度に絵を回転することができます。
透明度の設定はなさそうです。MATLABのopenGLでなくzbufferのような感じです。
またMATLABのCameraViewAngleに相当する機能はなさそうです。
編集→図のプロパティを選択します。
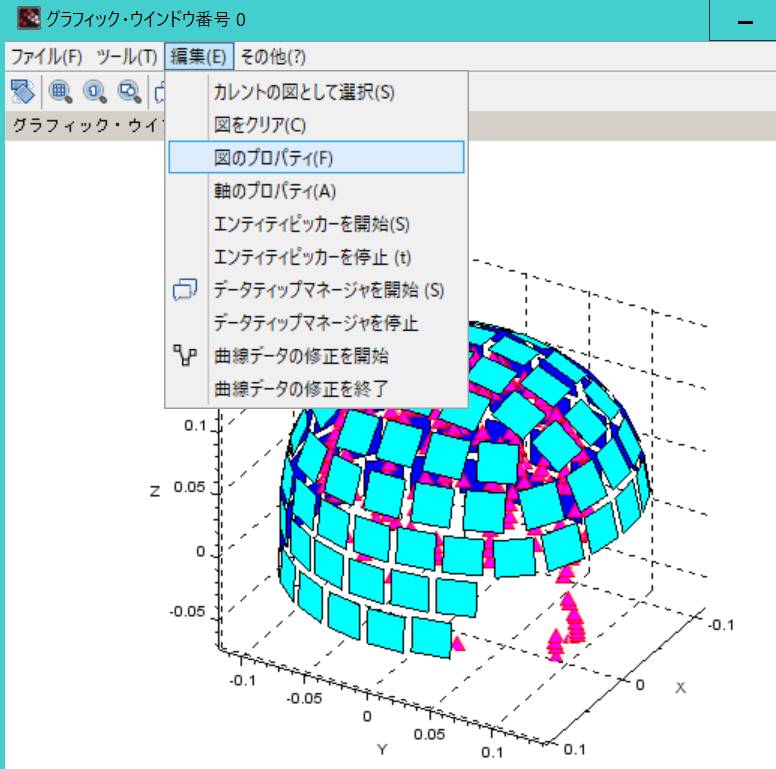
MATLABのinspect関数みたいな感じで、いろいろいじれるようです。
MRI三次元データ
***3D.fifファイルをFiff2MatFileでMATファイル化したファイルを読み込みます。長い文字列がないので読み込みはsmoothです。
clear();
loadfile=uigetfile('*.mat');
loadmatfile(loadfile);
読み込んだ結果です。読み込みが十分高速なのでDATファイルにする必要はないと思います。
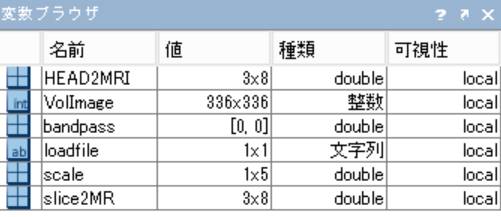
scaleの内容は以下の通りです。
-->
scale
scale =
256. 256. 0.24 0.24 86.
X軸のピクセル数、Y軸のピクセル数、X軸の長さ、Y軸の長さ、Z軸のピクセル数です。
Z軸の長さがありませんので、slice2MRから計算します。
SliceGap=norm(slice2MR(1:3,4,$)-slice2MR(1:3,4,1))/(size(Volume3D,3)-1);
scale(6)=SliceGap*size(Volume3D,3)
Z軸のピクセル長は0.002
m=2 mmでした。
scale(6)にZ軸の長さを入れておきます。
x=gsort(Volume3D,'g','i');//昇順 MATLABだとsort(Volume3D(:),'ascend')
N=length(x);
clim=x(round([0.1,0.9]*N));// 10〜90%とする
clim=double(clim);
hf=figure();//'backgroundcolor',[1,1,1]);
どうも反映されません
hf.color_map=graycolormap(256);//白黒表示
for m=1:3
subplot(2,2,m);
select
m //MATLABだとswitch
case
1 then
Q=squeeze(Volume3D(128,:,:));
Q=Q';
case
2 then
Q=squeeze(Volume3D(:,128,:));
Q=Q';
case
3 then Q=squeeze(Volume3D(:,:,43));
end
Q=double(Q);//整数のままだと単純な白黒2値のみ
Q=(Q-clim(1))/(clim(2)-clim(1))*255*0.8;//0.8かけたくらいが見やすい
Q(Q<0)=0;
Q(Q>255)=255;
Matplot(Q);
set(gca(),'isoview','on');
end
set(hf,'background',255);
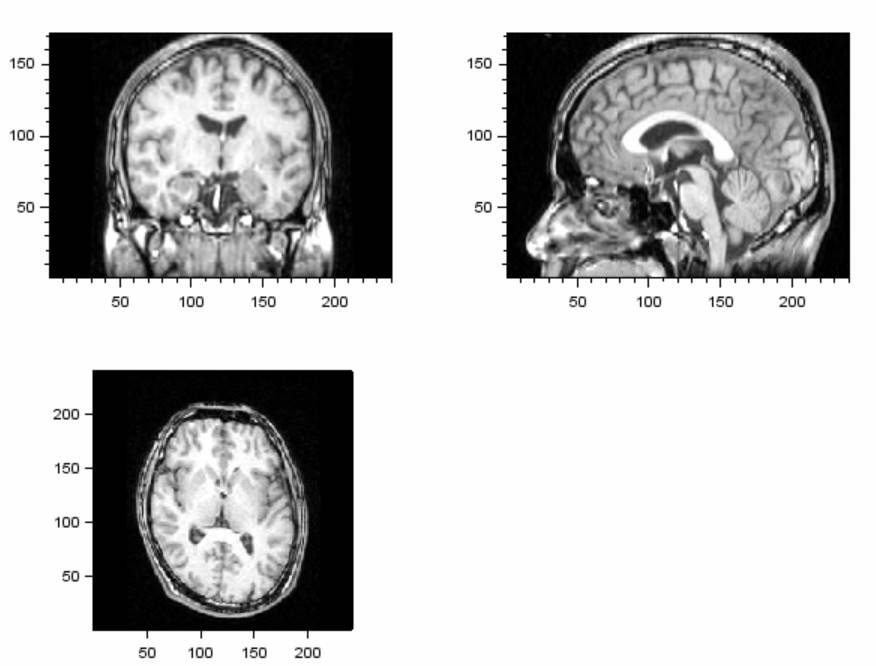
結果です。
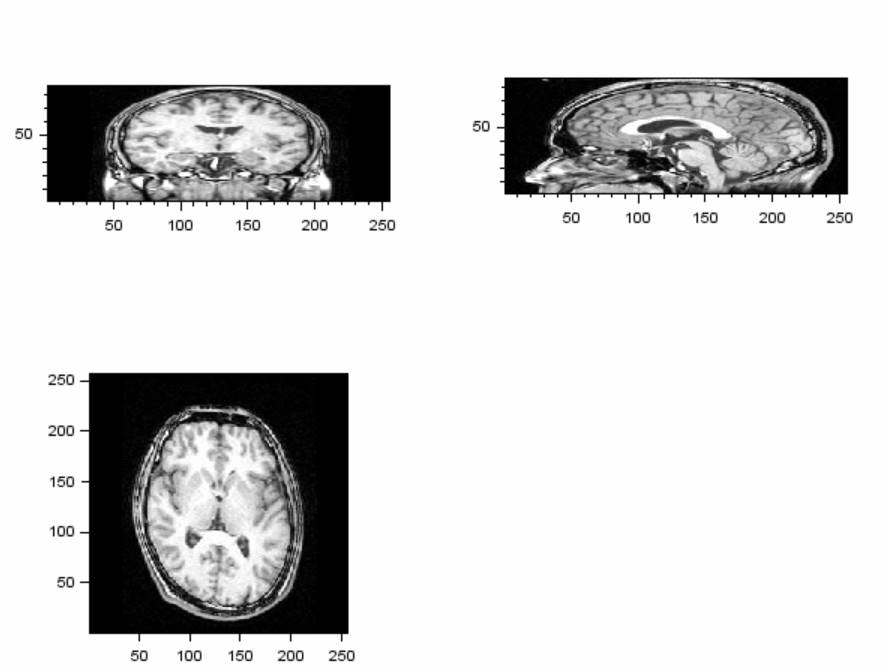
MATLABのaxesのclimで輝度を調整する方法は使えません。
画像の直接を0〜255の間になるように計算しています。
MATLABのdaspect機能に相当するものはないようです。
このままだと縦横比が合っていません
こちらで補間する必要があるようです。
linear_interpn関数を使います。MATLABのinterp2関数に相当しますが、引数が、linear_interpn(行列、行列、ベクトル、ベクトル、補間される行列)となっていて、interp2関数とはかなり異なっているので注意が必要です。
x=gsort(Volume3D,'g','i');//昇順 MATLABだとsort(Volume3D(:),'ascend')
N=length(x);
clim=x(round([0.1,0.9]*N));// 10〜90%とする
clim=double(clim);
hf=figure();//'backgroundcolor',[1,1,1]);
どうも反映されません
hf.color_map=graycolormap(256);//白黒表示
x0=1:scale(1);
y0=1:scale(2);
z0=1:scale(5);
x1=linspace(1,scale(1),round(scale(3)*1000));//scale(3)*1000を整数にするため
y1=linspace(1,scale(2),round(scale(4)*1000));
z1=linspace(1,scale(5),round(scale(6)*1000));
for m=1:3
subplot(2,2,m);
select
m //MATLABだとswitch
case
1 then
Q=squeeze(Volume3D(128,:,:));
[Z1,Y1]=meshgrid(z1,y1);
Q=linear_interpn(Y1,Z1,y0,z0,double(Q));//Qがint16のままだと実行できず
Q=Q';
case
2 then
Q=squeeze(Volume3D(:,128,:));
[Z1,X1]=meshgrid(z1,x1);
Q=linear_interpn(X1,Z1,x0,z0,double(Q));
Q=Q';
case
3 then
Q=squeeze(Volume3D(:,:,43));
[Y1,X1]=meshgrid(y1,x1);
Q=linear_interpn(X1,Y1,x0,y0,double(Q));
end
Q=double(Q);//整数のままだと単純な白黒2値のみ
Q=(Q-clim(1))/(clim(2)-clim(1))*255*0.8;//0.8かけたくらいが見やすい
Q(Q<0)=0;
Q(Q>255)=255;
Matplot(Q);
set(gca(),'isoview','on');
end
set(hf,'background',255);
結果です。
1pixelが1mmの正方形になるようにして、縦横比を合わせています。
脳表画像データ
*.scenes.fifファイルをFiff2MatFileでMATファイル化したファイルを読み込みます。こちらも読み込みは高速です。
clear();
loadfile=uigetfile('*.mat');
loadmatfile(loadfile);
読み込んだ結果です。
Neuromagは電流双極子を三次元脳表画像に重畳するのに、volume
renderingのアプリを用意していません。予め決まった角度に切り出した脳表画像を作成して、その上に載せる、という方針をとっていました。その名残です。こちらは縦横比は一定なので補間せずに、適当な角度の脳表画像を表示してみます。
hf=figure();
nn=round(rand(1,6)*size(VolImage,3));
for n=1:length(nn)
subplot(2,3,n);
Matplot(squeeze(VolImage(:,:,nn(n))));
set(gca(),'isoview','on');
end
hf.background=8;
結果です。
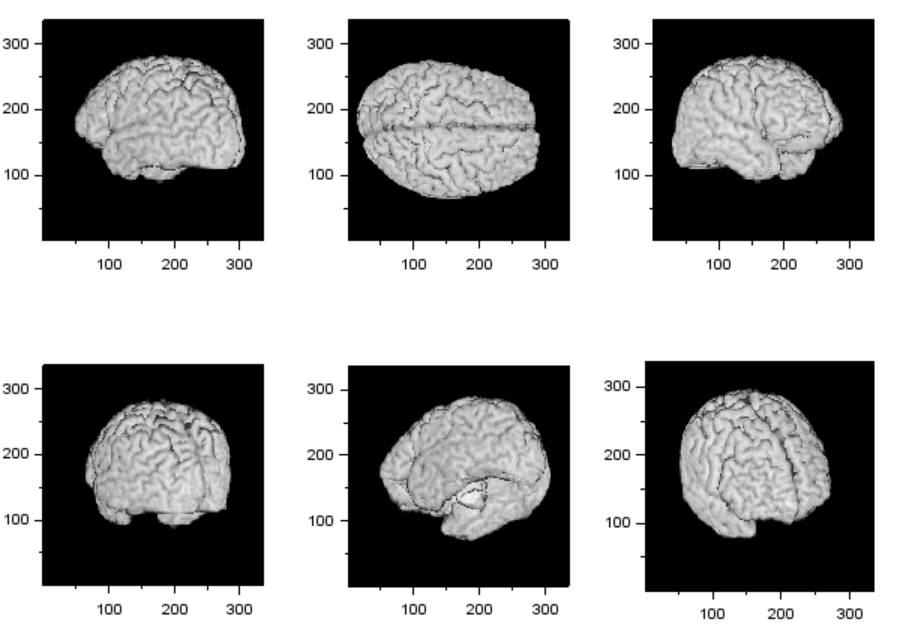
ここではfigureのcolor_mapの設定はしていませんが,ちゃんと白黒表示されています。。
Matplotは0〜255の整数だとcolormapを反映せず、浮動小数点doubleだとcolormapを反映する、といった特徴があるようです。