内容
MEG-Pythonの以下の部分を試みました。

導入〜パワースペクトラム密度表示まで
spyderを立ち上げ、Python consoleを使います。
MNE-Pythonを導入します
import numpy as np
import mne
from mne.datasets import sample
ファイルを読みます。
raw_fname='C:\\Users\\akira\\mne_data\\MNE-sample-data\\MEG\\sample\\sample_audvis_raw.fif'
proj_faname='C:\\Users\\akira\\mne_data\\MEG-sample-data\\MEG\\sample\\sample_audvis_eog_proj.fif'
tmin,tmax=0,20 # 最初の20秒を使う
raw=mne.io.read_raw_fif(raw_fname,add_eeg_ref=False)
raw.crop(tmin,tmax).load_data() #データの切り出し
raw.info['bads']=['MEG 2443','EEG 053'] # bad channel
0〜20秒のデータで、左側頭部の脳磁図センサの2〜300Hzのパワースペクトラム密度を表示します。
fmin,fmax=2,300 # 2〜300Hzが対象
n_fft=2048
selection=mne.read_selection('Left-temporal') #側頭部のセンサを選択
picks=mne.pick_types(raw.info,meg='mag',eeg=False,eog=False,stim=False,exclude='bads',selection=selection)
print(picks) # 左側頭部の脳磁図magnetometerだけが選択される
raw.plot_psd(area_mode='range',tmax=10.0,picks=picks)
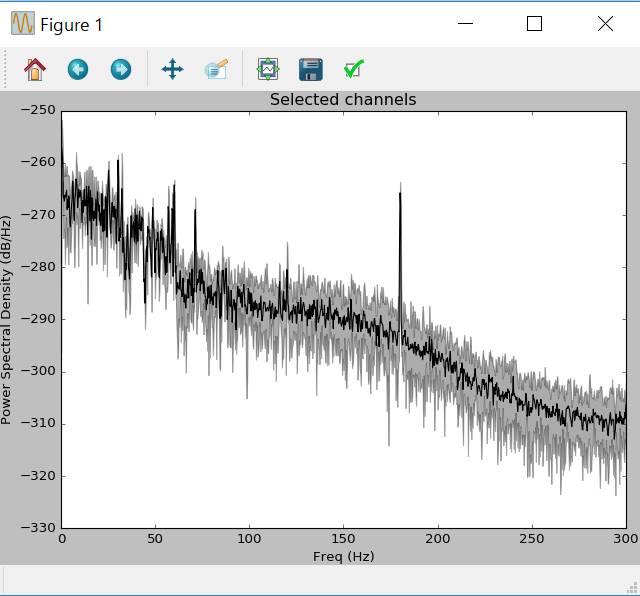
60、120、180Hzにやや上に凸の波形が出ています。
Notchフィルタの適応
notchフィルタをかけます。
raw.notch_filter(np.arange(60,241,60),picks=picks,filter_length='auto',phase='zero')
fir_windowはMNE-Python
0.13ではhann窓が使われていたけど、0.14でhamming窓に変更されてますと表示されますが、無視します。
ノッチフィルタ処理後のパワースペクトラム密度を見てみます。
raw.plot_psd(area_mode='range',tmax=10.0,picks=picks)
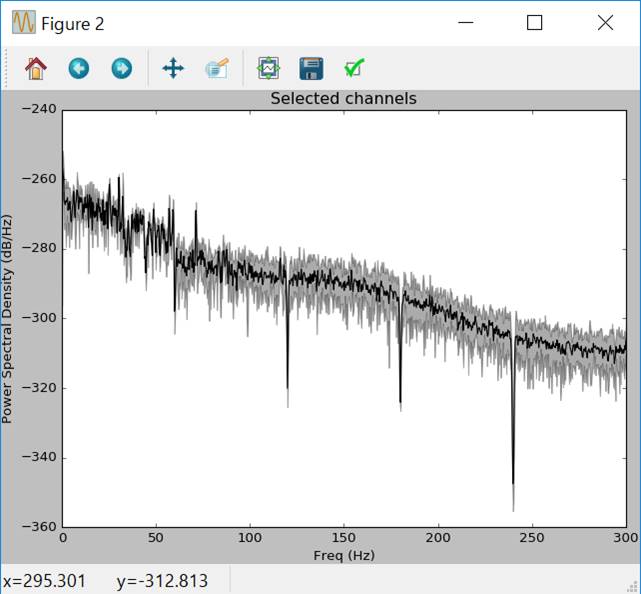
たぶん、60,120,180,240Hzの帯域が除去され下に尖った波形が出ています。
商用電源以下のローパスフィルタをかける
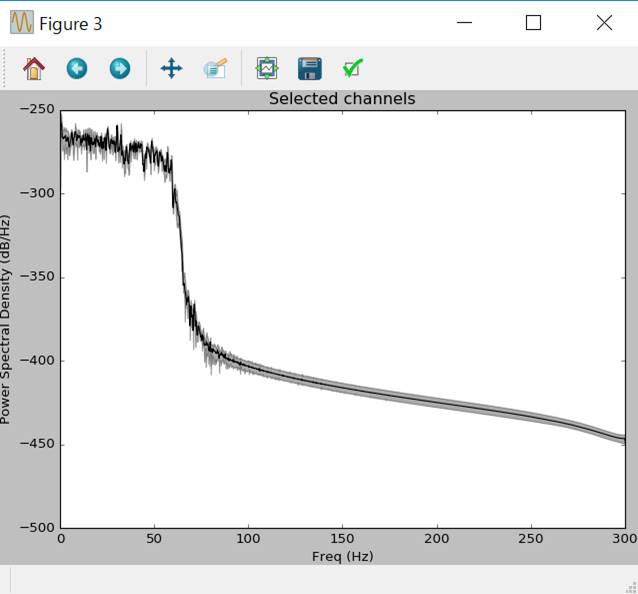
単に60Hzのローパスフィルタにすれば商用電源由来の雑音power-line noiseは取れます。
raw.filter(None,50.,h_trans_bandwidth='auto',filter_length='auto',phase='zero')
raw.plot_psd(area_mode='range',tmax=10.0,picks=picks)
低周波の揺らぎをハイパスフィルタでとる
低周波の揺らぎslow driftをハイパスフィルタで除去します。1Hz以下をカットします。
ただし0.1Hz以下の場合は慎重にしてください。
raw.filter(1.,None,l_trans_bandwidth='auto',filter_length='auto',phase='zero')
raw.plot_psd(area_mode='range',tmax=10.0,picks=picks)
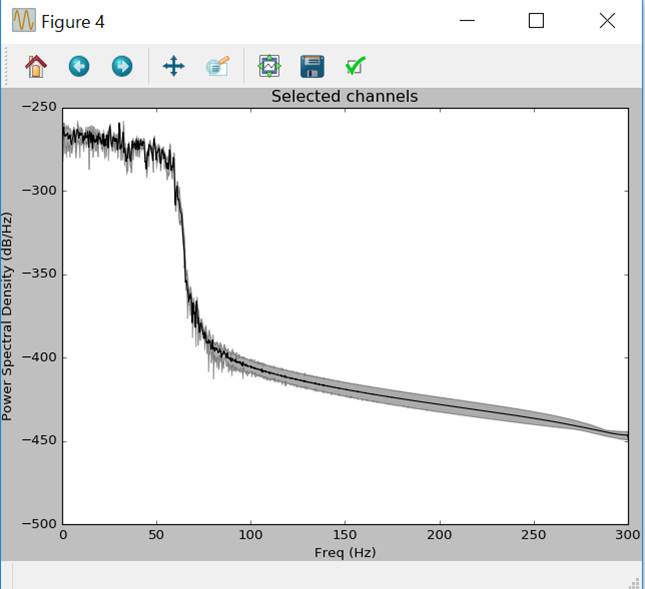
変わってないじゃん!
周波数帯域の表示を拡大します。
raw.plot_psd(area_mode='range',tmax=10.0,fmax=10.0,picks=picks)
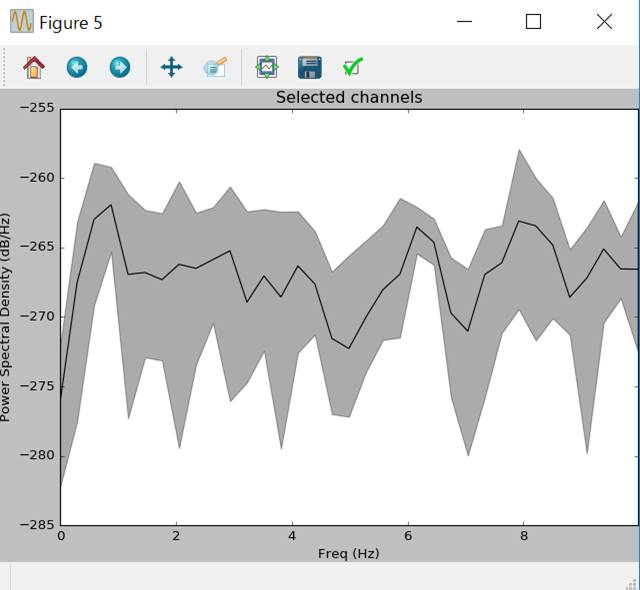
一応ハイパスフィルタかかってはいるんだ。
ローパス・ハイパスを同時にかける
1〜50Hzの周波数帯域通過フィルタを作成してかけてみます。
raw=mne.io.read_raw_fif(raw_fname,add_eeg_ref=False)
raw.crop(tmin,tmax).load_data()
raw.filter(1.,50,l_trans_bandwidth='auto',filter_length='auto',phase='zero')
raw.plot_psd(area_mode='range',tmax=10.0,fmax=60.,picks=picks)
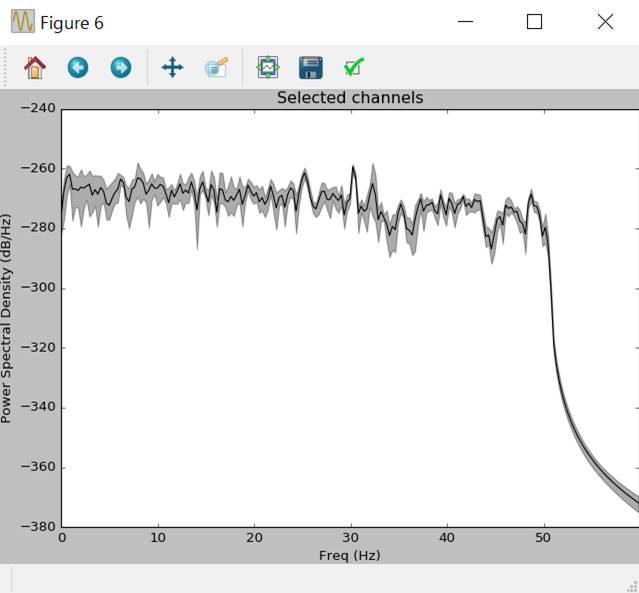
0〜60Hzで表示しています。
標本化周波数を減らしデータを間引く
標本化周波数が高いとデータ処理に時間がかかります。高周波成分や時間分解能は低下しますが、データ処理が速くなります。
標本化周波数を1000Hzから100Hzと1/10にします。
raw=mne.io.read_raw_fif(raw_fname,add_eeg_ref=False)
raw.crop(tmin,tmax).load_data()
raw.resample(100,npad='auto')
raw.plot_psd(area_mode='range',tmax=10.0,picks=picks)
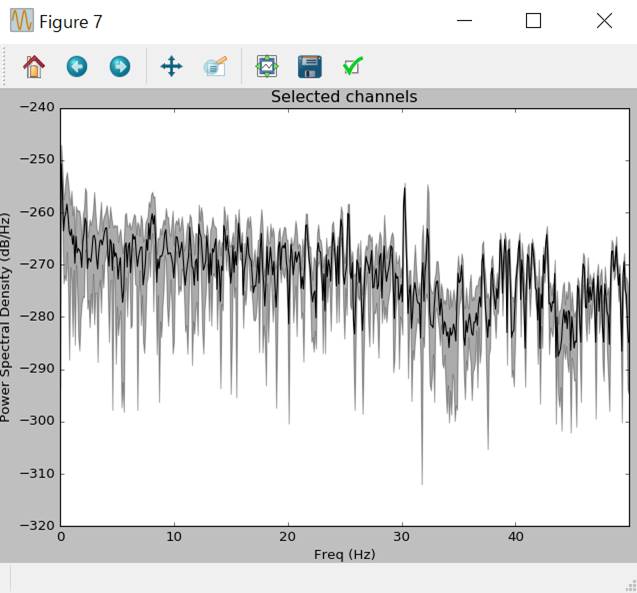
標本化周波数が100Hzなので0〜50Hzの表示になっています。
Aliasing予防のためローパスフィルタ後にデータを間引てdecimationいます。
詳細はscipy.signal.resample()とscipy.signal.resample_poly()を参照してください。
ウインドウを全部閉じます。
import
matplotlib.pyplot as plt
plt.close('all')