内容
MNE-Pythonのtutorialの以下の部分を試してみました。
L2ノルム推定
準備
import
numpy as np
import
matplotlib.pyplot as plt
import
mne
from
mne.datasets import sample
from
mne.minimum_norm import (make_inverse_operator,apply_inverse,write_inverse_operator)
脳磁図データ処理
脳磁図の生データを読み込んでエポックの切り出しをします。
data_path=sample.data_path()
raw_fname=data_path+'\\MEG\\sample\\sample_audvis_filt-0-40_raw.fif'
raw=mne.io.read_raw_fif(raw_fname,add_eeg_ref=False)
raw.set_eeg_reference() # EEGの平均をreferenceとする
events=mne.find_events(raw,stim_channel='STI
014')
event_id=dict(aud_r=1) # event triggerと条件
tmin=-0.2
tmax=0.5
raw.info['bads']=['MEG 2443','EEG 053']
picks=mne.pick_types(raw.info,meg=True,eeg=False,eog=True,exclude='bads')
baseline=(None,0)
reject=dict(grad=4000e-13,mag=4e-12,eog=150e-6)
epochs=mne.Epochs(raw,events,event_id,tmin,tmax,proj=True,picks=picks,baseline=baseline,reject=reject,add_eeg_ref=False)
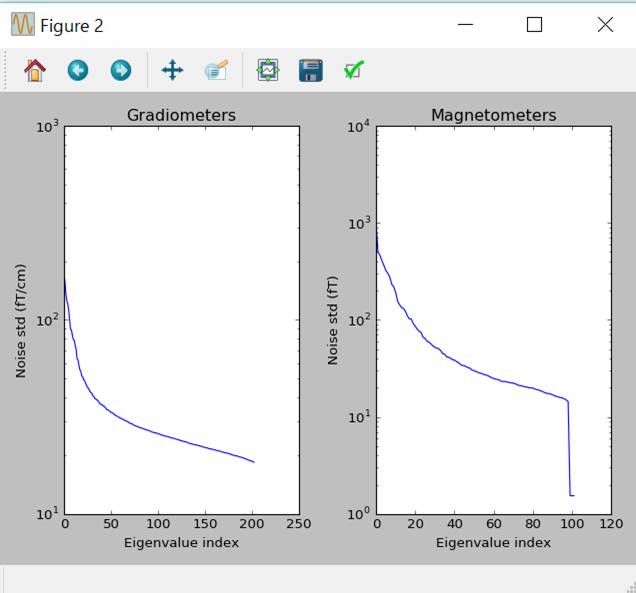
分散行列の規則化
noise_cov=mne.compute_covariance(epochs,tmax=0.,method=['shrunk','empirical'])
fig_cov,fig_spectra=mne.viz.plot_cov(noise_cov,raw.info)
加算平均波形の計算
evoked=epochs.average()
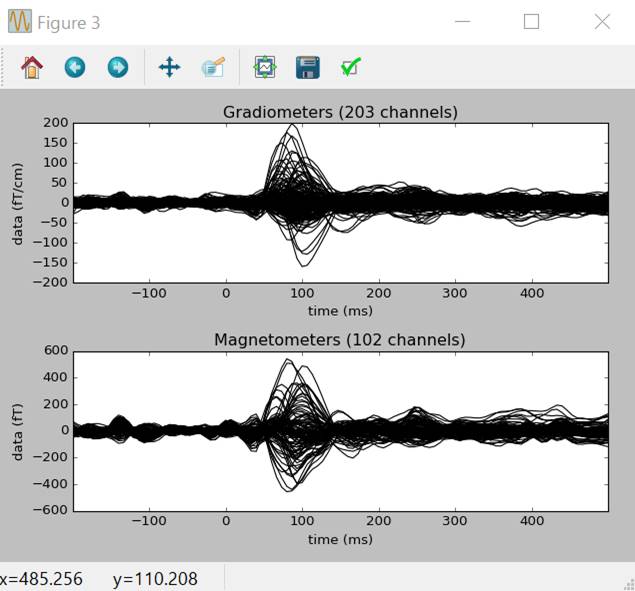
evoked.plot()
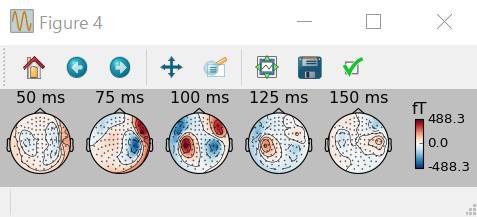
evoked.plot_topomap(times=np.linspace(0.05,0.15,5),ch_type='mag')
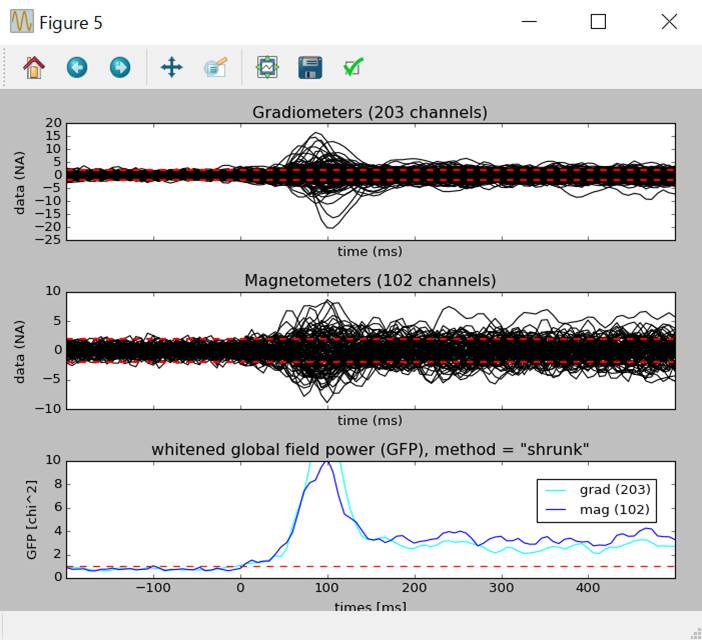
evoked.plot_white(noise_cov)
逆問題を解く準備
#
導体モデルの読み込み
fname_fwd=data_path+'\\MEG\\sample\\sample_audvis-meg-oct-6-fwd.fif'
fwd=mne.read_forward_solution(fname_fwd,surf_ori=True)
#
導体モデルを脳磁図だけにする
fwd=mne.pick_types_forward(fwd,meg=True,eeg=False)
#
逆問題を解く
info=evoked.info
inverse_operator=make_inverse_operator(info,fwd,noise_cov,loose=0.2,depth=0.8)
write_inverse_operator('sample_audvis-meg-oct-6-inv.fif',inverse_operator)
電流源推定
method='dSPM'
snr=3.
lambda2=1./snr**2
stc=apply_inverse(evoked,inverse_operator,lambda2,method=method,pick_ori=None)
#
del fwd,inverse_operator,epochs # メモリ節約
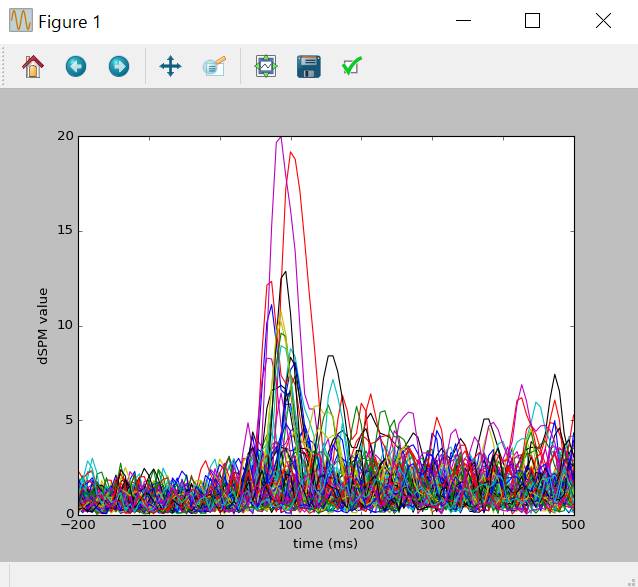
結果表示(3Dは実行不可)
plt.close('all') # ウインドウは全部閉じる
plt.plot(1e3*stc.times,stc.data[::100,:].T)
plt.xlabel('time (ms)')
plt.ylabel('%s value' % method)
plt.show()
三次元表示です。実行は・・・できませんでした。
vertno_max,time_max=stc.get_peak(hemi='rh')
subjects_dir=data_path+'\\subjects'
brain=stc.plot(surface='inflated',hemi='rh',subjects_dir=subjects_dir,clim=dict(kind='value',lims=[8,12,15]),initial_time=time_max,time_unit='s')
brain.add_foci(vertno_max,coords_as_verts=True,hemi='rh',color='blue',scale_factor=0.6)
brain.show_view('lateral')
File
"C:\Users\akira\Anaconda3\lib\site-packages\mne\source_estimate.py",
line 1334, in plot
time_unit=time_unit)
File "C:\Users\akira\Anaconda3\lib\site-packages\mne\viz\_3d.py",
line 704, in plot_source_estimates
Traceback (most recent call last):
File "<stdin>", line 1, in <module>
import
surfer
ImportError: No module named 'surfer'
module
surferの導入を試みます。
Microsoft Windows [Version 10.0.14393]
(c) 2016 Microsoft Corporation. All rights reserved.
C:\Users\akira>pip install surfer
Collecting surfer
Could not find a version that satisfies
the requirement surfer (from versions: )
No matching
distribution found for surfer
C:\Users\akira>
導入できませんでした。
以下もmodule surferがないため実行できませんでした。
fs_vertices=[np.arange(10242)]*2
morph_mat=mne.compute_morph_matrix('sample','fsaverage',stc.vertices,fs_vertices,smooth=None,subjects_dir=subjects_dir)
stc_fsaverage=stc.morph_precomputed('fsaverage',fs_vertices,morph_mat)
brain_fsaverage=stc_fsaverage.plot(surface='inflated',hemi='rh',subjects_dir=subjects_dir,clim=dict(kind='value',lims=[8,12,15]),initial_time=time_max,
time_unit='s')
brain_fsaverage.show_view('lateral')
単一電流双極子推定
準備と各ファイル読み込み
from
os import path as op
import
numpy as np
import
matplotlib.pyplot as plt
plt.close('all')
import
mne
from
mne.forward import make_forward_dipole
from
mne.evoked import combine_evoked
from
mne.simulation import simulate_evoked
data_path=mne.datasets.sample.data_path()
subjects_dir=op.join(data_path,'subjects')
fname_ave=op.join(data_path,'MEG','sample','sample_audvis-ave.fif')
fname_cov=op.join(data_path,'MEG','sample','sample_audvis-cov.fif')
fname_bem=op.join(subjects_dir,'sample','bem','sample-5120-bem-sol.fif')
fname_trans=op.join(data_path,'MEG','sample','sample_audvis_raw-trans.fif')
fname_surf_lh=op.join(subjects_dir,'sample','surf','lh.white')
単一電流双極子推定
脳磁図データだけを使って単一電流双極子推定を行います。
evoked=mne.read_evokeds(fname_ave,condition='Right
Auditory',baseline=(None,0))
evoked.pick_types(meg=True,eeg=False)
evoked_full=evoked.copy()
evoked.crop(0.07,0.08)
dip=mne.fit_dipole(evoked,fname_cov,fname_bem,fname_trans)[0]
結果表示(実行不可)
結果の表示ですが、できません。module mayaviのPython 3対応待ちです。
dip.plot_locations(fname_trans,'sample',subjects_dir)
計測データと計算データの比較
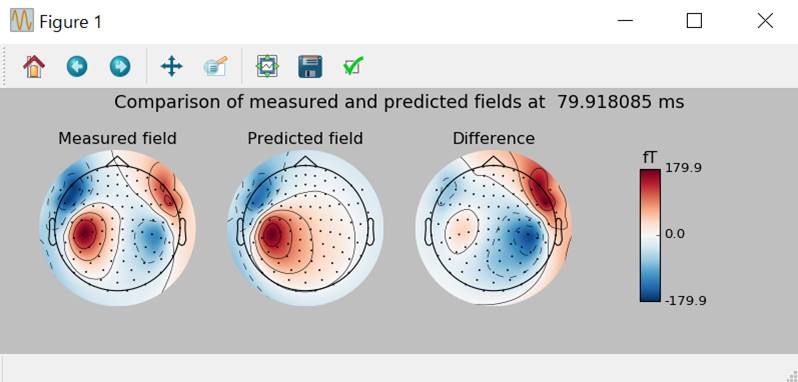
GOF最高時点の等磁場線図の比較をします。
fwd,stc=make_forward_dipole(dip,fname_bem,evoked.info,fname_trans)
pred_evoked=simulate_evoked(fwd,stc,evoked.info,None,snr=np.inf)
#
GOF最高点の時間
best_idx=np.argmax(dip.gof)
best_time=dip.times[best_idx]
#
図の表示準備
fig,axes=plt.subplots(nrows=1,ncols=4,figsize=[10.,3.4])
vmin,vmax=-400,400 # 色表示のスケール
#
GOF最高時間のトポグラフィ表示
plot_params=dict(times=best_time,ch_type='mag',outlines='skirt',colorbar=False)
evoked.plot_topomap(time_format='Measured
field',axes=axes[0],**plot_params)
#
計算画像との比較
pred_evoked.plot_topomap(time_format='Predicted
field',axes=axes[1],**plot_params)
#
差分
diff=combine_evoked([evoked,-pred_evoked],weights='equal')
plot_params['colorbar']=True
diff.plot_topomap(time_format='Difference',axes=axes[2],**plot_params)
plt.suptitle('Comparison of measured and
predicted fields at {: 0f} ms'.format(best_time*1000.),fontsize=16)
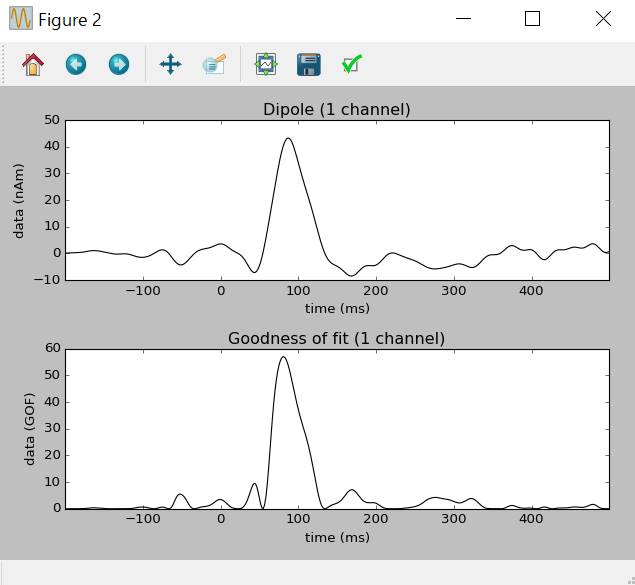
電流双極子の経時変化
dip_fixed=mne.fit_dipole(evoked_full,fname_cov,fname_bem,fname_trans,pos=dip.pos[best_idx],ori=dip.ori[best_idx])[0]
dip_fixed.plot()