内容
MNE-Python
tutorialのこの部分を試してみました。
センサ単位の周波数解析・時間周波数解析
いろいろ導入
いろいろ導入します。
import
numpy as np
import
matplotlib.pyplot as plt
import
mne
from
mne.time_frequency import tfr_morlet,psd_multitaper
from
mne.datasets import somato
ファイル読み込み・エポック切り出し
ファイルを読み込みます。
新たにsomatoというのを読み込みます。700MB強あり、downloadに時間がかかります。
data_path=somato.data_path()
# 764MB download
raw_fname=data_path+'\\MEG\\somato\\sef_raw_sss.fif'
raw=mne.io.read_raw_fif(raw_fname,add_eeg_ref=False)
エポックを切り出します。
events=mne.find_events(raw,stim_channel='STI
014')
picks=mne.pick_types(raw.info,meg='grad',eeg=False,eog=True,stim=False)
event_id,tmin,tmax=1,-1.,3.
baseline=(None,0)
reject=dict(grad=4000e-13,eog=350e-6)
epochs=mne.Epochs(raw,events,event_id,tmin,tmax,picks=picks,baseline=baseline,reject=reject,preload=True,add_eeg_ref=False)
高周波成分は使わないので再標本化してデータ量を減らして処理の高速化を図ります。
epochs.resample(150.,npad='auto')
# 高周波成分は使わないので再標本化でデータ量を削減
周波数解析
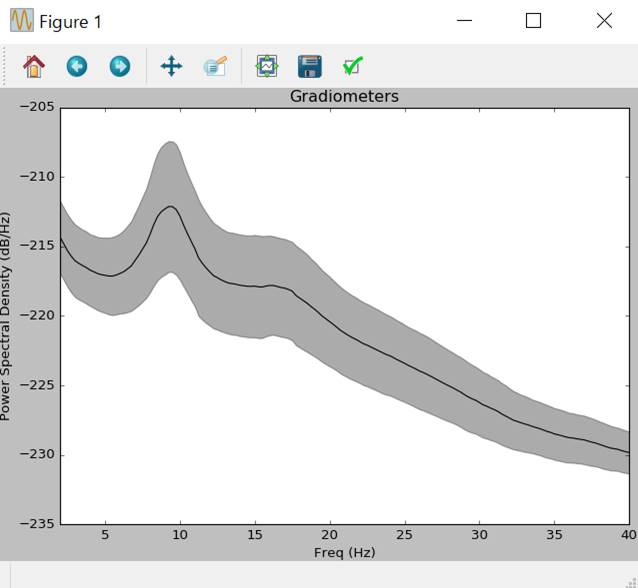
周波数分布をみます。パワースペクトラム密度を表示します。
epochs.plot_psd(fmin=2.,fmax=40.)
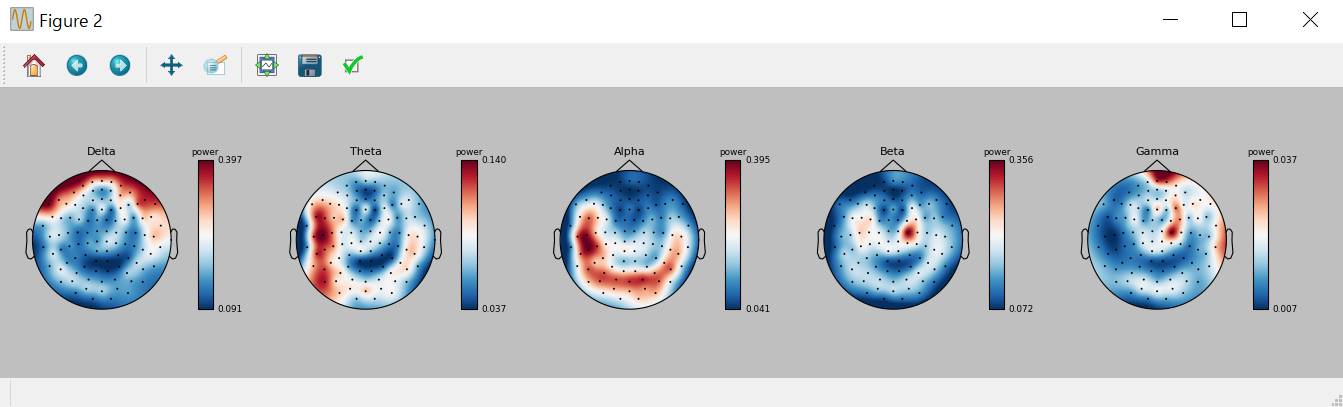
トポグラフィ表示してみます。
epochs.plot_psd_topomap(ch_type='grad',normalize=True)
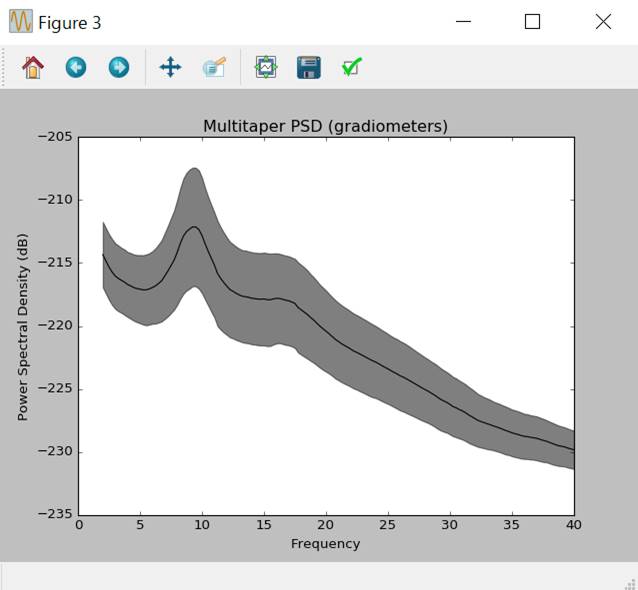
パワースペクトラム密度は以下の方法でも作図できます。
f,ax=plt.subplots()
psds,freqs=psd_multitaper(epochs,fmin=2,fmax=40,n_jobs=1)
psds=10*np.log10(psds)
psds_mean=psds.mean(0).mean(0)
psds_std=psds.mean(0).std(0)
ax.plot(freqs,psds_mean,color='k')
ax.fill_between(freqs,psds_mean-psds_std,psds_mean+psds_std,color='k',alpha=0.5)
ax.set(title='Multitaper
PSD (gradiometers)',xlabel='Frequency',ylabel='Power Spectral Density (dB)')
plt.show()
時間周波数解析
周波数帯域別パワー値の計算です。
freqs=np.arange(6,30,3)
n_cycles=freqs/2.
power,itc=tfr_morlet(epochs,freqs=freqs,n_cycles=n_cycles,use_fft=True,return_itc=True,decim=3,n_jobs=1)
power.plot_topo(baseline=(-0.5,0.),mode='logratio',title='Average
power')
power.plot([82],baseline=(-0.5,0.),mode='logratio')
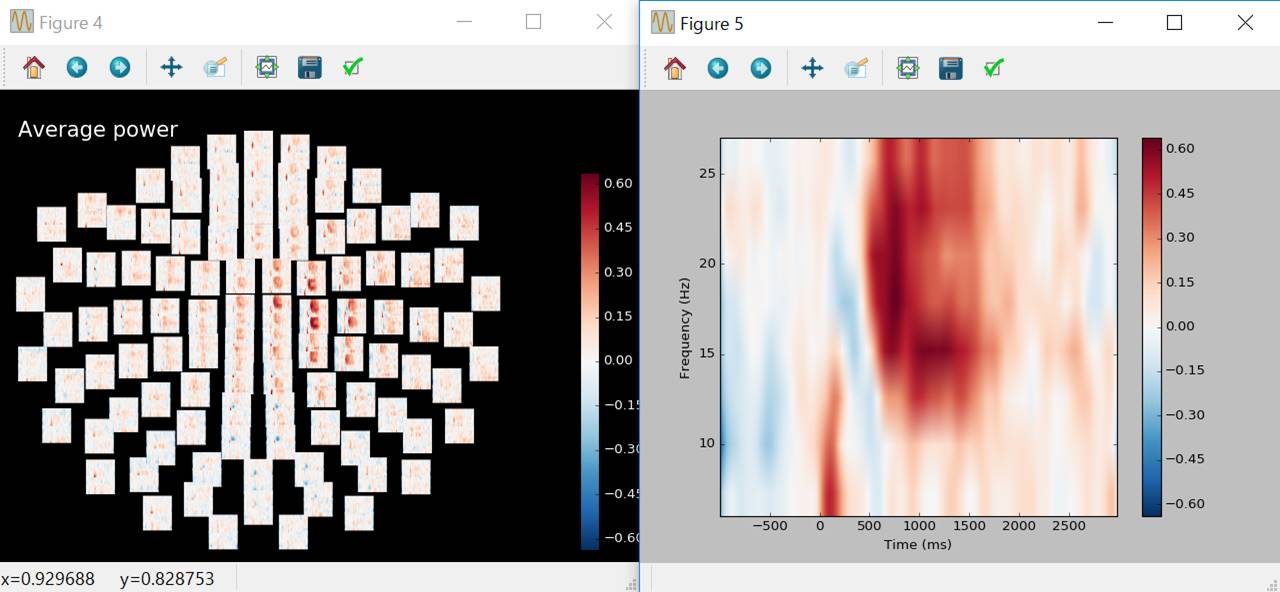
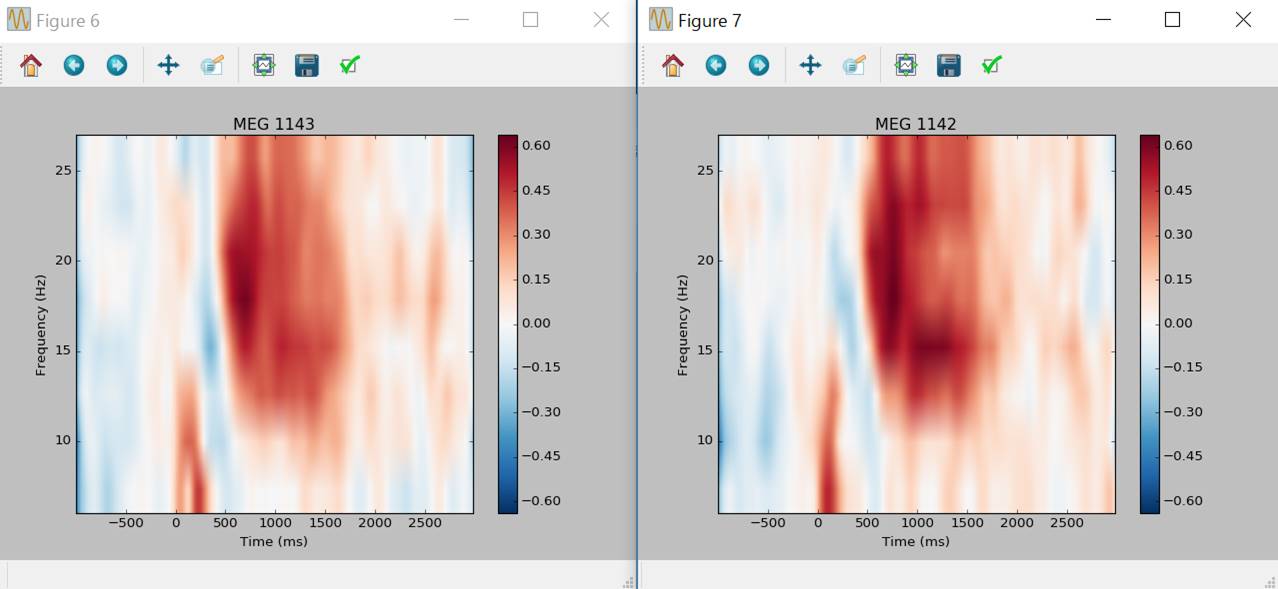
左図ではGradiometer2つが上下に表示されています。センサをクリックすれば時間周波数マップが表示されます。
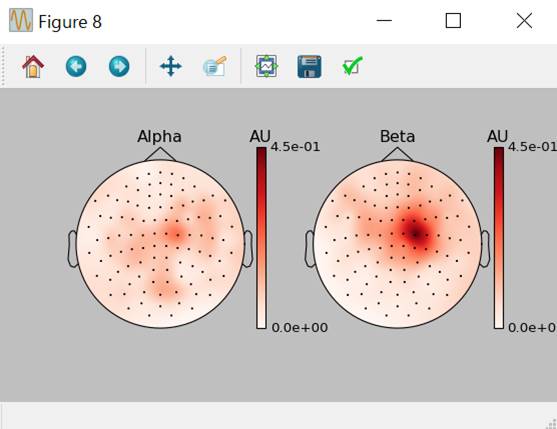
周波数帯域別のトポグラフィを提示します。
fig,axis=plt.subplots(1,2,figsize=(7,4))
power.plot_topomap(ch_type='grad',tmin=0.5,tmax=1.5,fmin=8,fmax=12,baseline=(-0.5,0),mode='logratio',axes=axis[0],title='Alpha',vmax=0.45,show=False)
power.plot_topomap(ch_type='grad',tmin=0.5,tmax=1.5,fmin=13,fmax=25,baseline=(-0.5,0),mode='logratio',axes=axis[1],title='Beta',vmax=0.45,show=False)
mne.viz.tight_layout()
plt.show()
Intra-Trial Coherence
トライアル間のコヒーレンスinter-trial coherence (ITC)を計算します。
エポックごとの再現性をみてるのかな?と思いますが、よくわかりません。
itc.plot_topo(title='Inter-Trial
coherence',vmin=0.,vmax=1.,cmap='Reds')
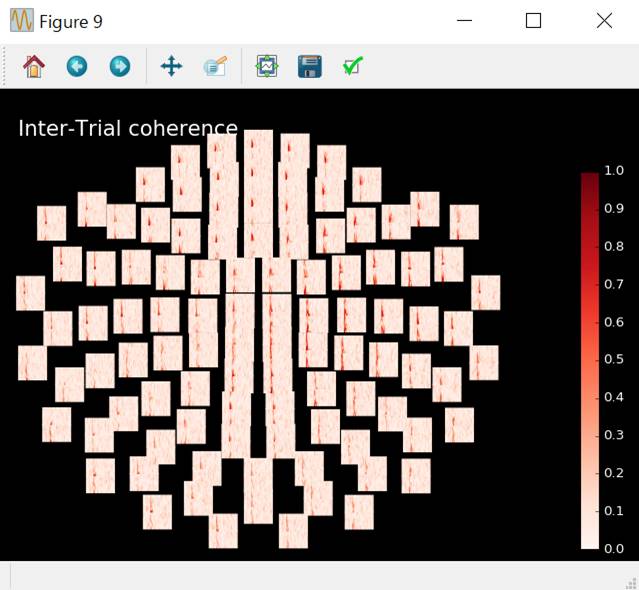
センサ部分をクリックすれば拡大表示されます。
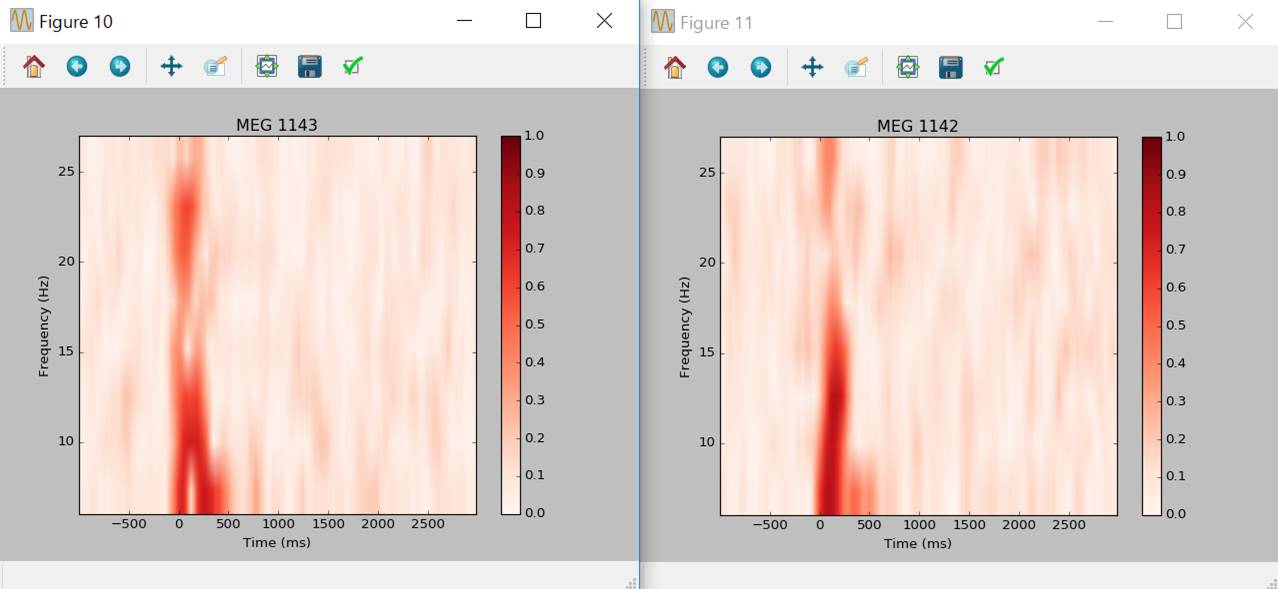
ウインドウを閉じます。
plt.close(‘all’)
センサレベルでの復号
内容は理解してません。コードが実行できるかどうかの確認です。
雑音を除去し、センサレベルで信号を復号します。
多変量パターン分析Multivariate pattern analysis (MVPA)やsupervised
machine learningという手法を用います。
いろいろ導入
いろいろ導入します。
import
numpy as np
import
matplotlib.pyplot as plt
from
sklearn.metrics import roc_auc_score
from
sklearn.cross_validation import StratifiedKFold # 警告が出る
警告が出ますが無視します。
C:\Users\akira\Anaconda3\lib\site-packages\sklearn\cross_validation.py:44:
DeprecationWarning: This module was deprecated in version 0.18 in favor of the
model_selection module into which all the refactored classes and functions are
moved. Also note that the interface of the new CV iterators are different from
that of this module. This module will be removed in 0.20.
"This module will
be removed in 0.20.", DeprecationWarning)
import
mne
from
mne.datasets import sample
from
mne.decoding import TimeDecoding,GeneralizationAcrossTime
ファイル読み込み
ファイルを読み込みます。
data_path='C:\\Users\\akira\\mne_data\\MNE-sample-data'
raw_fname=data_path+'\\MEG\\sample\\sample_audvis_filt-0-40_raw.fif'
event_fname=data_path+'\\MEG\\sample\\sample_audvis_filt-0-40_raw-eve.fif'
min,tmax=-0.2,0.5
event_id=dict(aud_l=1,vis_l=3)
raw=mne.io.read_raw_fif(raw_fname,preload=True,add_eeg_ref=False)
エポックの切り出し
エポックを切り出します。
raw.set_eeg_reference()
# 脳波 平均を基準に
raw.filter(2,None,method='iir')
# 基準線仕様の代わりにhigh passを使用
events=mne.read_events(event_fname)
raw.info['bads']+=['MEG
2443','EEG 053']
picks=mne.pick_types(raw.info,meg='grad',eeg=False,stim=True,eog=True,exclude='bads')
reject=dict(grad=4000e-13,eog=150e-6)
epochs=mne.Epochs(raw,events,event_id,tmin,tmax,proj=True,picks=icks,baseline=None,preload=True,reject=reject,add_eeg_ref=False)
epochs_list=[epochs[k]
for k in event_id]
mne.epochs.equalize_epoch_counts(epochs_list)
data_picks=mne.pick_types(epochs.info,meg=True,exclude='bads')
Temporal Decoding 時間的復号
時間的変化があったとして、どのような信号の変化があったかをSupport Vector Machineを使って復号します。Support
Vector Machineは交差検証cross-validationという統計手法を使っているそうです。
td=TimeDecoding(predict_mode='cross-validation',n_jobs=1)
# 交差検証法 cross-validation
td.fit(epochs)
いろいろ表示されます。
<TimeDecoding
| fitted, start : -0.200 (s), stop : 0.499 (s), no prediction, no score>
td.score(epochs)
[0.52033333333333331,
0.55335897435897441, 0.52002564102564097, 0.52802564102564109,
0.50464102564102575, 0.54500000000000004, 0.5126666666666666,
0.41397435897435902, 0.48861538461538467, 0.53058974358974365,
0.54656410256410259, 0.56066666666666665, 0.54628205128205132,
0.50628205128205139, 0.57933333333333337, 0.63343589743589745,
0.54023076923076929, 0.5146153846153847, 0.44825641025641022,
0.53730769230769226, 0.46269230769230763, 0.55717948717948729,
0.62576923076923074, 0.52033333333333331, 0.49435897435897436,
0.48730769230769228, 0.52641025641025641, 0.6071282051282052,
0.65105128205128193, 0.60138461538461541, 0.60907692307692307,
0.59435897435897433, 0.64305128205128192, 0.65007692307692311,
0.63210256410256416, 0.69848717948717942, 0.77282051282051278,
0.75615384615384618, 0.68151282051282047, 0.72156410256410264,
0.77794871794871789, 0.82697435897435889, 0.93464102564102569,
0.93366666666666664, 0.96699999999999997, 0.96730769230769231,
0.95064102564102571, 0.93430769230769228, 0.91828205128205131,
0.90961538461538471, 0.91733333333333333, 0.86864102564102574,
0.8673333333333334, 0.93399999999999994, 0.94299999999999995,
0.893948717948718, 0.93430769230769228, 0.87761538461538469,
0.85292307692307701, 0.81861538461538463, 0.82633333333333314, 0.84330769230769231,
0.77217948717948715, 0.73982051282051275, 0.73243589743589743,
0.74717948717948723, 0.70553846153846156, 0.76323076923076916,
0.72312820512820508, 0.77123076923076916, 0.76223076923076927,
0.73884615384615393, 0.72343589743589742, 0.63505128205128192,
0.6171282051282051, 0.6193333333333334, 0.63664102564102554,
0.61074358974358978, 0.62, 0.59310256410256401, 0.58674358974358976,
0.64402564102564108, 0.65171794871794864, 0.70651282051282061,
0.79589743589743578, 0.81225641025641016, 0.73087179487179488,
0.62612820512820522, 0.62515384615384617, 0.59182051282051285,
0.50528205128205128, 0.48223076923076924, 0.53669230769230769,
0.55530769230769228, 0.60210256410256413, 0.52966666666666673,
0.55371794871794866, 0.48125641025641019, 0.52905128205128205,
0.48766666666666669, 0.6322051282051282, 0.60335897435897423,
0.56007692307692314, 0.59343589743589742, 0.43961538461538457,
0.62738461538461543]
td.plot(title='Sensor
space decoding')
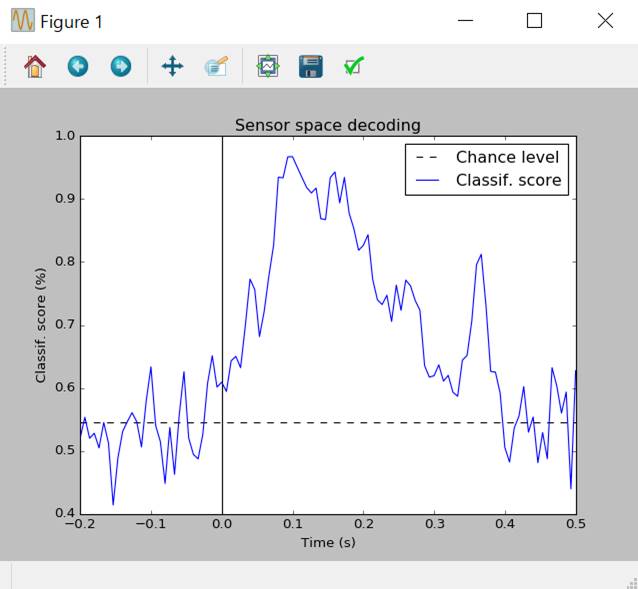
よくわかりませんが、たぶん0.0〜0.4秒になんらかの反応があったということが復号されたんだと思います。
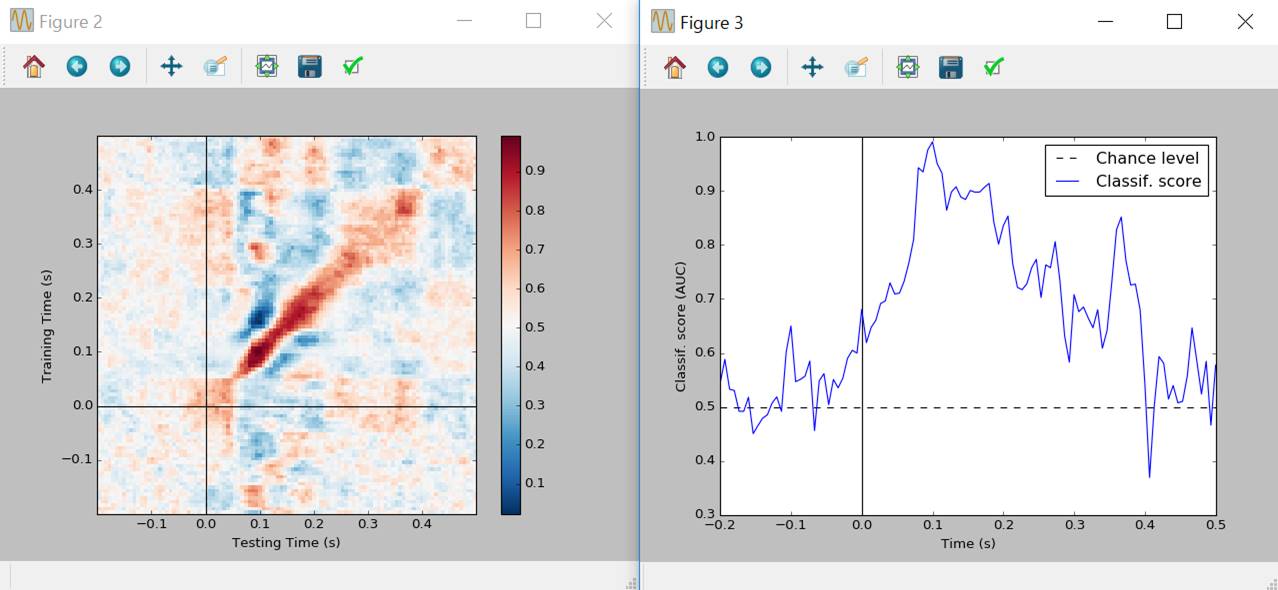
Generalization Across Time
時間間での一般化と訳すんでしょうか?全然理解してません。層別交差検証法stratified cross validationを使っているようです。
y=np.zeros(len(epochs.events),dtype=int)
y[epochs.events[:,2]==3]=1
cv=StratifiedKFold(y=y)
# 層別交差検証法
#
GeneralizationAcrossTimeオブジェクトの定義
gat=GeneralizationAcrossTime(predict_mode='crossvalidation',n_jobs=1,cv=cv,scorer=roc_auc_score)
gat.fit(epochs,y=y)
gat.score(epochs)
<GAT
| fitted, start : -0.200 (s), stop : 0.499 (s), no prediction, no score>
array([[
0.54373637, 0.58028608, 0.54557782, ..., 0.48401637,
0.49764233, 0.50847722],
[
0.62106958, 0.58821626, 0.60177866, ..., 0.4948763 ,
0.52194523, 0.52210318],
[
0.50357311, 0.51033215, 0.53273005, ..., 0.47142863,
0.48897249, 0.52198376],
...,
[
0.51389563, 0.49013013, 0.46789019, ..., 0.58449483,
0.5381735 , 0.54014786],
[
0.4896428 , 0.49181364, 0.49464323, ..., 0.54356494,
0.46603526, 0.50046421],
[
0.56781777, 0.51759201, 0.54052732, ..., 0.48876253,
0.52079914, 0.5773544
]])>> >
図示します。
gat.plot()
gat.plot_diagonal()