内容
MNE-Python
tutorialのこの部分を試してみました。

いろいろ導入
いろいろ読み込みます。
import
numpy as np
import
mne
from
mne.datasets import sample
from
mne.preprocessing import compute_proj_ecg,compute_proj_eog
raw_fname='c:\\Users\\akira\\mne_data\\MNE-sample-data\\MEG\\sample\\sample_audvis_filt-0-40_raw.fif'
raw=mne.io.read_raw_fif(raw_fname,preload=True,add_eeg_ref=False)
raw.set_eeg_reference()
raw.pick_types(meg=True,ecg=True,eog=True,stim=True)
Signal Space Projectionの計算
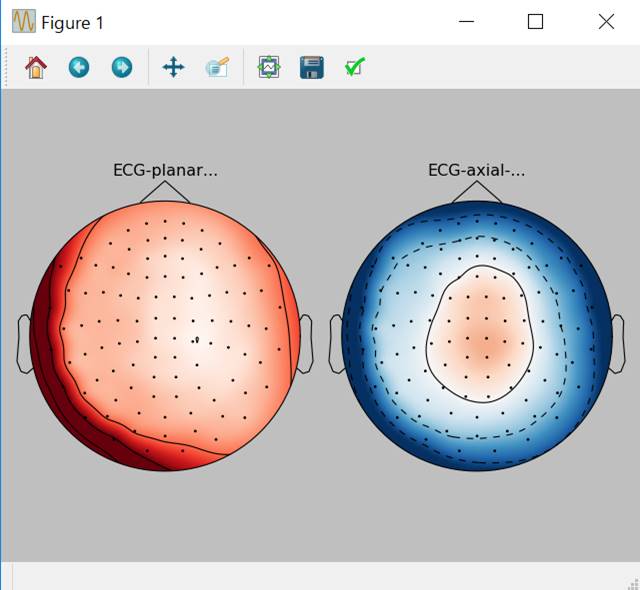
心電図を使ってcompute_proj_ecg()を使って固有値ベクトルを求めます。Gradiometer用が1個、magnetometer用が1個です。
projs,events=compute_proj_ecg(raw,n_grad=1,n_mag=1,average=True)
print(projs)

projsの中身です。最初の4つは使いません。5,6のトポグラフィを書かせます。
<Projection |
PCA-v1, active : False, n_channels : 102>,
<Projection |
PCA-v2, active : False, n_channels : 102>,
<Projection |
PCA-v3, active : False, n_channels : 102>,
<Projection |
Average EEG reference, active : False, n_channels
: 60>,
<Projection |
ECG-planar--0.200-0.400-PCA-01, active : False, n_channels
: 203>
<Projection |
ECG-axial--0.200-0.400-PCA-01, active : False, n_channels
: 102>
ecg_projs=projs[-2:]
mne.viz.plot_projs_topomap(ecg_projs)
# Matlabだとecg_projcs(end-1:end)
眼電図を使って同じようにgradiometer、magnetometerに1個ずつの固有ベクトルを求めます。
projs,events=compute_proj_eog(raw,n_grad=1,n_mag=1,average=True)
print(projs)
eog_projs=projs[-2:]
mne.viz.plot_projs_topomap(eog_projs)

SSPをかける
SSPをかけてみます。
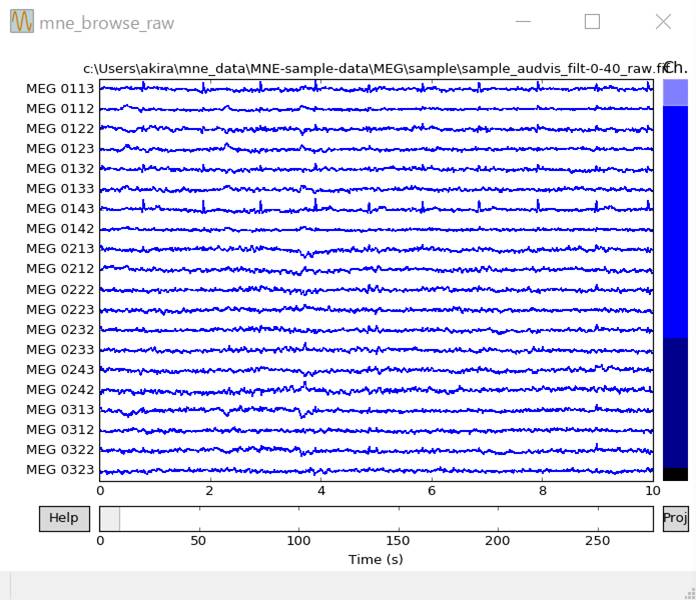
raw.plot(duration=10,n_channels=20)
raw.info['projs']+=eog_projs+ecg_projs
raw.plot(duration=10,n_channels=20)
かける前です。
かけた後です。
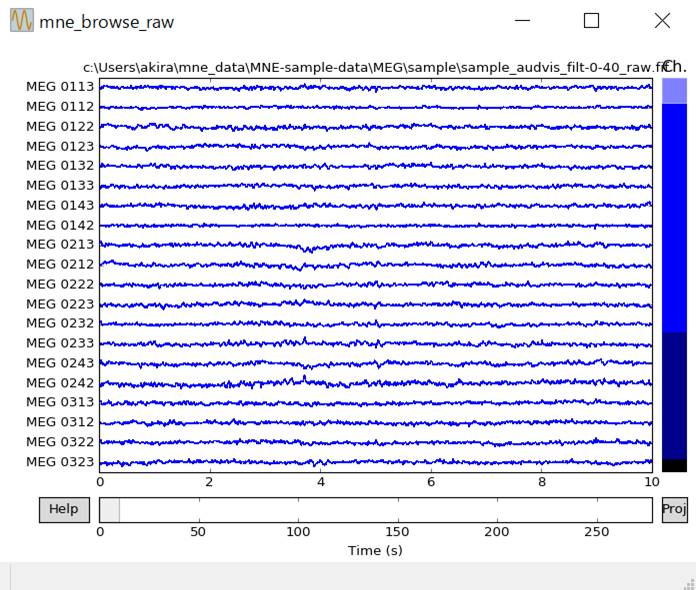
心電図の影響が消えています。たぶん眼電図の影響も消えてます。
加算波形にSSPをかける
STI
014=1をトリガーにしてgradiometer 4000×10-13以上、magnetometer
4×10-12以上、脳波150×10-6以上のエポックは無視し、-0.2〜0.3秒の加算平均を作成します。基線は0秒以前で補正します。SSPをかける前の波形を見てみます。
events=mne.find_events(raw,stim_channel='STI
014')
reject=dict(grad=4000e-13,mag=4e-12,eog=150e-6)
event_id={'auditory/left':1}
epochs_no_proj=mne.Epochs(raw,events,event_id,tmin=-0.2,tmax=0.5,proj=False,baseline=(None,0),reject=reject)
epochs_no_proj.average().plot(spatial_colors=True)
エラーが出ますが無視します。
C:\Users\akira\Anaconda3\lib\site-packages\matplotlib\figure.py:1742:
UserWarning: This figure includes Axes that are not
compatible with tight_layout, so its results might be
incorrect.
warnings.warn("This
figure includes Axes that are not "
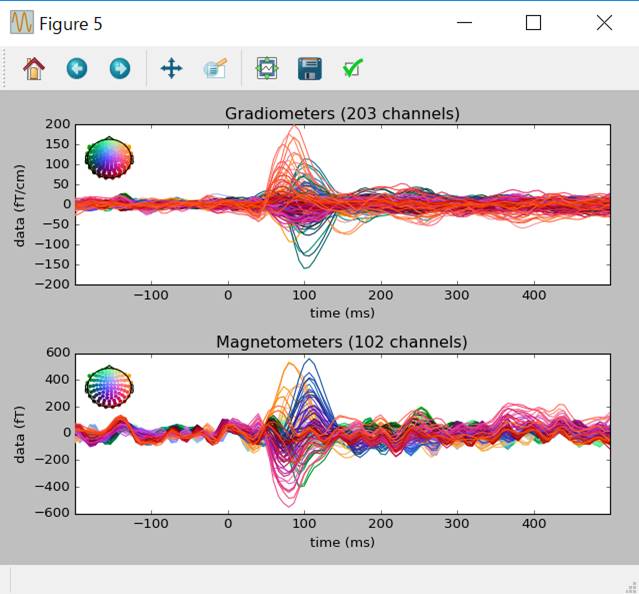
SSPをかけた後です。
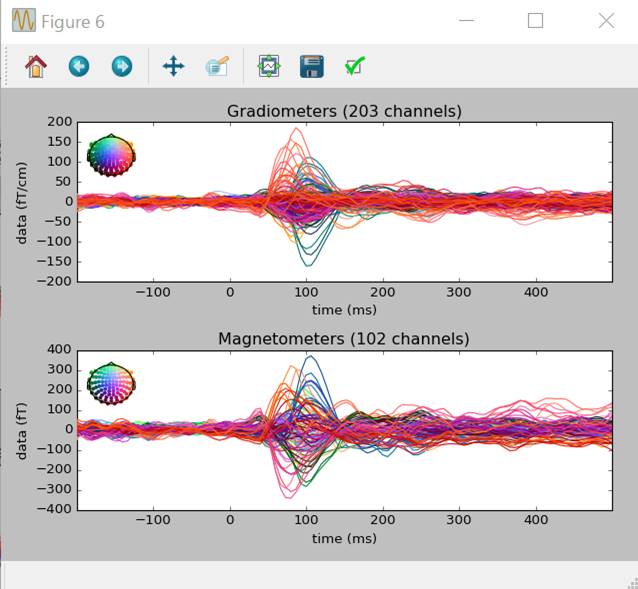
違いがよくわかりませんね。細かく見ていきます。
times=np.arange(0.05,0.15,0.01)
times=np.arange(0.05,0.15,0.01)
evoked=mne.Epochs(raw,events,event_id,tmin=-0.2,tmax=0.5,proj=False,baseline=(None,0),reject=reject).average()
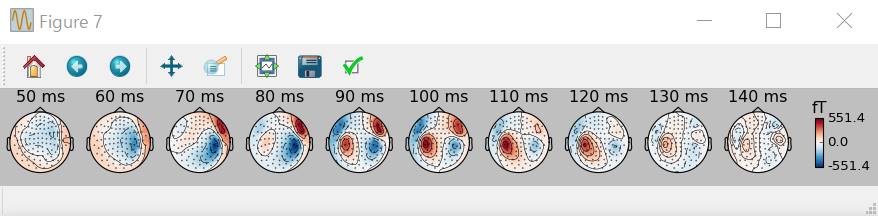
evoked.plot_topomap(times)
evoked=mne.Epochs(raw,events,event_id,tmin=-0.2,tmax=0.5,proj=True,baseline=(None,0),reject=reject).average()
evoked.plot_topomap(times)
SSPなしです。
SSPありです。
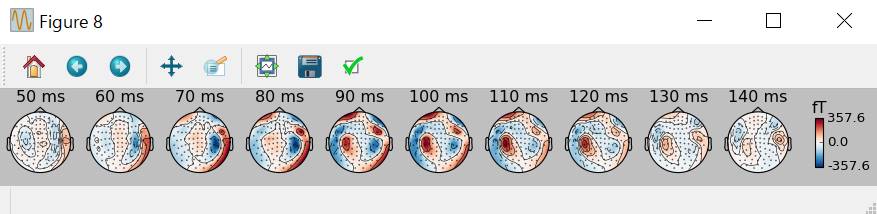
上の図の方が素直な感じがします。下の図は眼電図がはいっているような感じ。但しfTの値をみれば下の図の方が小さいのでSSPはかかってるんだと思います。
Interactiveに操作できるそうです。
evoked=mne.Epochs(raw,events,event_id,tmin=-0.2,tmax=0.5,proj='delayed',baseline=(None,0),reject=reject).average()
evoked.plot_topomap(times,proj='interactive')

SSPなしの時と同じです。このチェックボックスをクリックすれば変化が分かるはず・・・

SSP
projection vectorsのチェックボックスをクリックしてみます。

落ちてしまいました。Bugですかね。