NiBabel導入¶
BrainVISAではpyton 2.7系のPyAIMSなどが使われているようですが、導入方法が不明です。
そっちを使う方が便利なんでしょうが、いろいろ導入を試みたのですが諦めました。
NiBabelはPythonでN次元配列データであるNIfTIファイルやメッシュデータであるGIfTIファイルの読み書きを行うpackageです。こちらを使うことにしました。
BrainVISAではvolume dataにはNIfTIを採用してます。
Ubuntuに組み込むにはpython-pipを
sudo apt-get install python-pip
sudo apt-get install python-numpy
sudo pip install nibabelで導入すればいいのですが、その後のいろいろ画像処理を行うには統合環境の方がなにかと便利だと思います。
そこでanaconda (Python 2.7)のcondaを使って導入することにします。
以下のように端末にタイプします。
conda install -c conda-forge nibabelimport sys
print(sys.version_info) # pythonのversion確認
import numpy as np
import matplotlib.pyplot as plt # 描画用
import scipy.interpolate as interpolate
import nibabel as nib # NiBabelの導入の確認 エラーが出なければ成功
NIfTIファイルの読み込み¶
NiBabel自体はDICOMファイルをNIfTIに変換する関数は用意されていないようです。
manualではmricronのdcm2niiの記載があります。
この部分は省略して既にmricronのdcm2niiでDICOMファイルをNIfTIファイル、拡張子nii.gzに変換されているものとします。
ファイルの読み込みと確認を行います。
pathname='C:/Users/akira/' # windowsでは\を/に変えておく
filename='20130131_101135T13DAXI3s003a1001.nii.gz'
nii0=nib.load(pathname+filename)
img0=nii0.get_data()
print(img0.shape) # 三次元配列の大きさ表示
plt.imshow(img0[:,:,img0.shape[2]/2].T,cmap='gray',origin='lower')
plt.show()
512×512を256×256に¶
BrainVISAは512×512だと処理にえらく時間がかかるので256×256にします。
# affine変換部分の変更
affine=nii0.affine.copy()
print(affine)
affine[[0,0]]=affine[[0,0]]*2
affine[[1,1]]=affine[[1,1]]*2
print(affine)
# 3次元配列部分の変更
img1=np.zeros((img0.shape[0]/2,img0.shape[1]/2,img0.shape[2]),dtype=np.int16)
print(img1.shape)
x0=range(0,img0.shape[0])
y0=range(0,img0.shape[1])
x1=np.linspace(0,img0.shape[0]-1,img1.shape[0])
y1=np.linspace(0,img0.shape[1]-1,img1.shape[1])
for i in range(0,img1.shape[2]):
f=interpolate.interp2d(x0,y0,img0[:,:,i],kind='linear') # fは関数でMatlabのinterp2の使い方と異なる
img1[:,:,i]=f(x1,y1)
plt.imshow(img1[:,:,img1.shape[2]/2].T,cmap='gray',origin='lower')
plt.show()
NIfTIファイルとして保存¶
三次元配列、Affine行列さえあればNIfTI1ファイルとして保存できます。
# NIfTIファイルとして保存
nii1=nib.Nifti1Image(img1,affine=affine)
nib.save(nii1,pathname+'512to256.nii.gz')
voronoiデータを使って大脳を抽出¶
BrainVISAでsplit_brainをやると左右大脳半球と脳幹に色分けされます。このデータを使って大脳半球だけを抽出してみます。
filename='voronoi_20000406_132343s002a1001.nii.gz'
nii2=nib.load(pathname+filename)
img2=nii2.get_data()
print(img2.shape) # 三次元配列の大きさ表示
plt.imshow(img2[:,100,:].T,cmap='gray',origin='lower')
plt.show()
色分けは左半球=1、右半球=2、脳幹+小脳=3、眼球=4、その他=0とその他です。本来0,1,2,3,4なのですが、そうでない場合もあるようです。
脳幹、小脳、眼球はなしにして、左右半球を色分け1で表示してみます。
img2[img2==2]=1 # 脳幹・眼球は無しにする
img2[img2!=1]=0 # 左右半球の色分けをしない
plt.imshow(img2[:,100,:].T,cmap='gray',origin='lower')
plt.show()
filename='20000406_132343s002a1001.nii.gz'
nii3=nib.load(pathname+filename)
img3=nii3.get_data()
img4=img3*img2
plt.imshow(img4[:,100,:].T,cmap='gray',origin='lower')
plt.show()
なんか変です。左右が変わってる感じです。
print(nii2.affine)
print(nii3.affine)
x軸の方向が逆になっています。
img4=np.flipud(img3)*img2 # 基準はvoronoiとする
plt.imshow(img4[:,100,:].T,cmap='gray',origin='lower')
plt.show()
# Analyze形式で保存
nii4=nib.Nifti1Image(img4,affine=nii2.affine.copy()) # 基準はvoronoiとする。
nib.save(nii4,pathname+'brain.img') # 拡張子をnii.gzにすれば圧縮NIfTI形式で保存される
Analyze形式で保存したのでMRIcroで開けます。大脳の抽出がこれでずいぶん楽になりそうです。
GIfTIファイルの読み込み¶
% reset
import numpy as np
import matplotlib.pyplot as plt # 描画用
import scipy.interpolate as interpolate
import nibabel as nib # NiBabelの導入の確認 エラーが出なければ成功
pathname='C:/Users/akira/' # windowsでは\を/に変えておく
filename='20000406_132343s002a1001_Lhemi.gii' # 左半球
gii=nib.load(pathname+filename)
print(dir(gii))
print(dir(gii.header)) # headerの中身・・・たいしたこと書いてません
print(gii.numDA) # ndarrayの数
print(dir(gii.file_map)) # textureだと思うけど 中身なさそう
print(dir(gii.extra)) # 中身なさそう
print(gii.labeltable.labels) # label名
x=gii.darrays
print(len(x)) # gii.numDAと同じ
print(x[0].data.dtype)
print(x[0].data.shape)
print(x[0].data) # 頂点座標
print(x[0].coordsys.xform)
print(x[1].data.dtype)
print(x[1].data.shape)
print(x[1].data) # 三角メッシュの組み合わせ
print(x[1].coordsys.xform)
三次元表示する時はmayaviを使います。mayaviは端末を開いて
conda install mayaviで導入します。
では三次元表示させてみます。
from mayavi import mlab
mlab.figure(bgcolor=(0,0,0)) # 背景は黒
mlab.triangular_mesh(x[0].data[:,0],x[0].data[:,1],x[0].data[:,2],x[1].data,color=(1,1,0)) # 脳皮質は黄色
mlab.show()
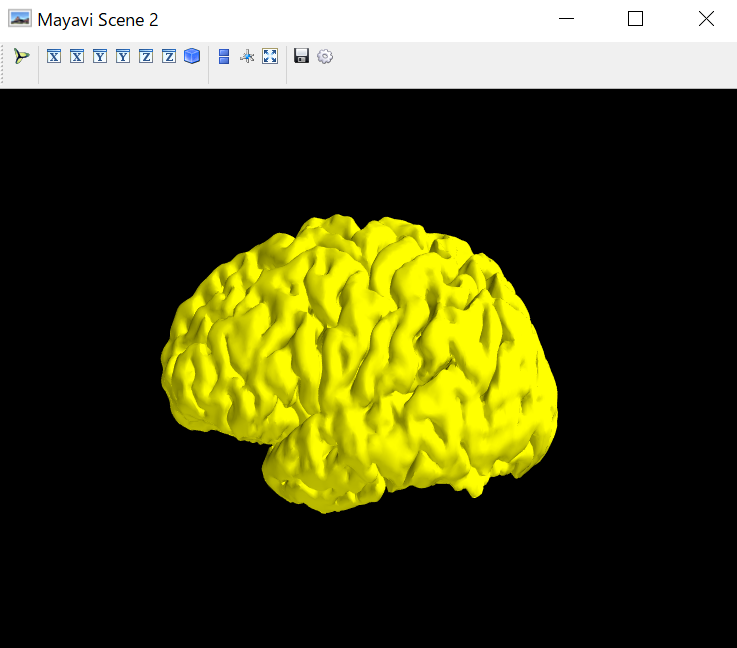
新しくウインドウが開き以下の画像が表示されます。左ドラッグで画像が回転、マウスホイールドラッグでへこう移動します。
左半球を読み込んだはずが・・・左右反転しています。
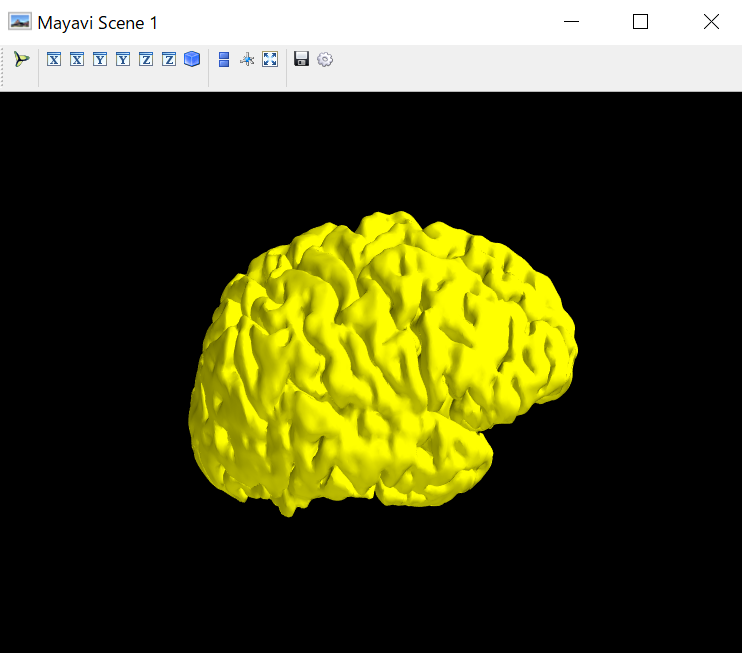
x座標のデータをマイナスにします。
mlab.figure(bgcolor=(0,0,0)) # 背景は黒
mlab.triangular_mesh(-x[0].data[:,0],x[0].data[:,1],x[0].data[:,2],x[1].data,color=(1,1,0)) # x座標をマイナスにする
mlab.show()
これで左半球の左右反転が直りました。