内容
NIfTIファイルの読み書き
Matlab R2017bからimage processing toolboxでNIfTIファイルの読み書きができるようになりました。
GIfTIファイルには対応していません。
A MATLAB GIfTI library
https://www.artefact.tk/software/matlab/gifti/
のgifti-1.6.zip (v1.6)を使うことにします。
Matlabのコード
Matlabのコマンドは以下の通りです。
voronoi_〇〇は1 右半球、2 左半球、3 小脳、4 左眼球でラベルされた三次元配列です。通常4はないのでn=1:3で十分です。
%% パスの設定
addpath('c:/ToBeInstalled/MATLAB/gifti-1.6');%
GIfTI Libraryのフォルダ
pathname='c:/Users/akira/';%
データのあるフォルダ
%% データの読み込み
filename='voronoi_20000406_132343s002a1001.nii.gz';
info=niftiinfo([pathname,filename]);
data=niftiread([pathname,filename]);
data=data(end:-1:1,end:-1:1,end:-1:1); %
データの並び替え
pixscale=diag(info.PixelDimensions);
for n=1:4 %
通常は
n=1:3
v=double(data==n);
v=smooth3(v);
v=isosurface(v,0.7);
v.vertices=v.vertices(:,[2,1,3])*pixscale;
g=gifti(v);
switch n
case
1;savename='Rhemi.gii';
case
2;savename='Lhemi.gii';
case
3;savename='cerebellum.gii';
case
4;savename='Leye.gii';
end
save(g,[pathname,savename],'Base64Binary');
end
Anatomistで確認
Ubuntuを起動し、4つのファイルをコピーします。場所は~/default_acquisition/default_analysis/meshとします。
BrainVISAを起動し、ViewersのAnatomist Show Meshをダブルクリックします。
![]() をクリックします。
をクリックします。
なんでもいいんですが、cerebellum.giiを選択します。
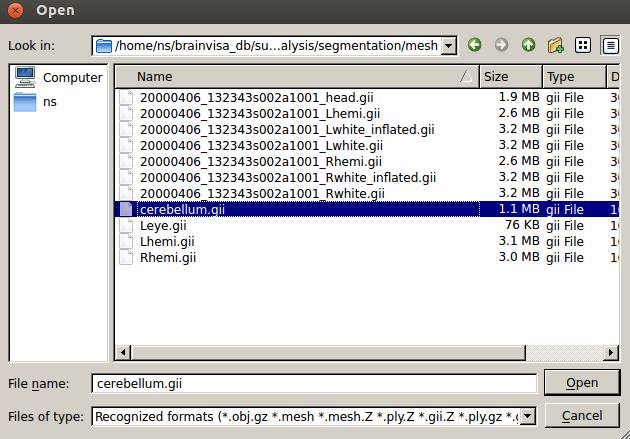
Runを押します。
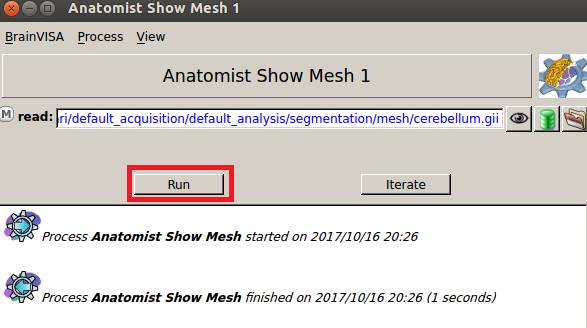
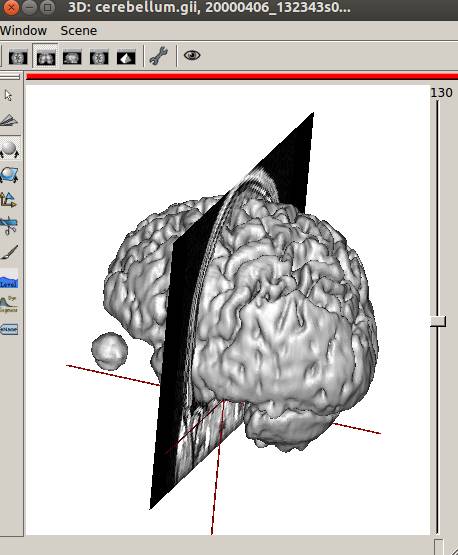
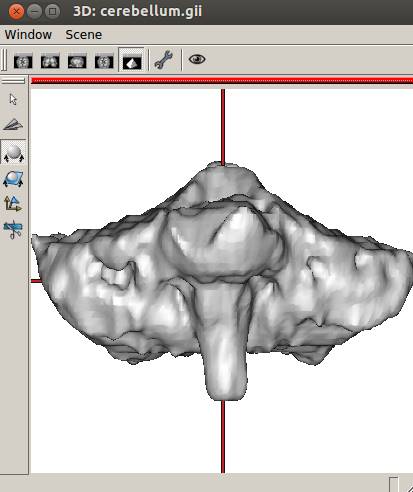
Anatomistが起動し、小脳が表示されます。
![]() をクリックします。
をクリックします。
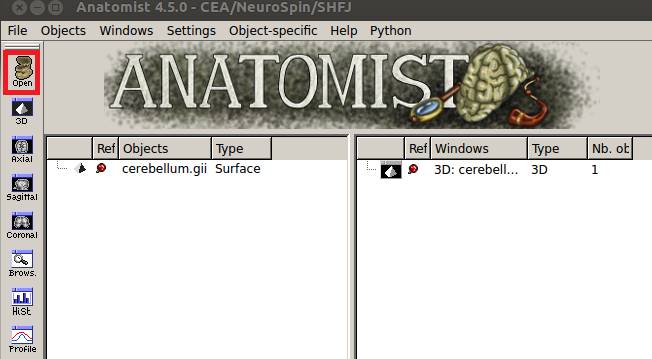
小脳以外の作成したメッシュを選択します。
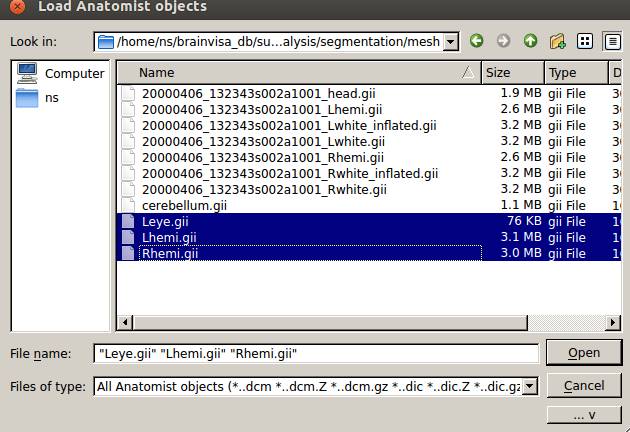
![]() をクリックします。
をクリックします。
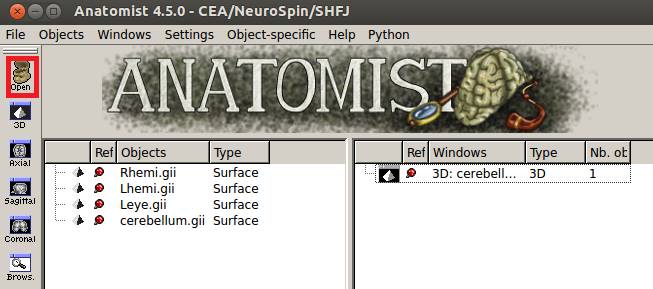
元データを選択します。
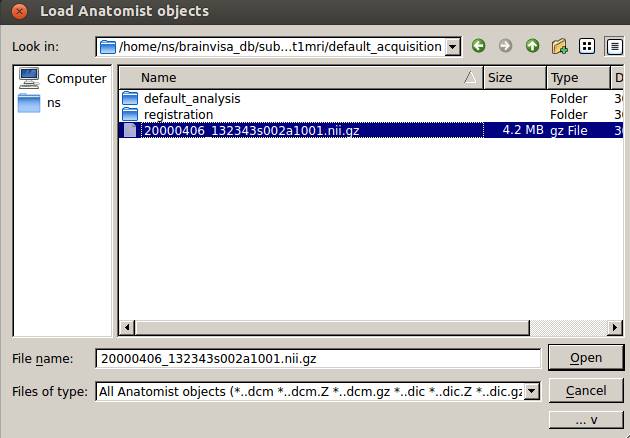
小脳以外をドラッグします。
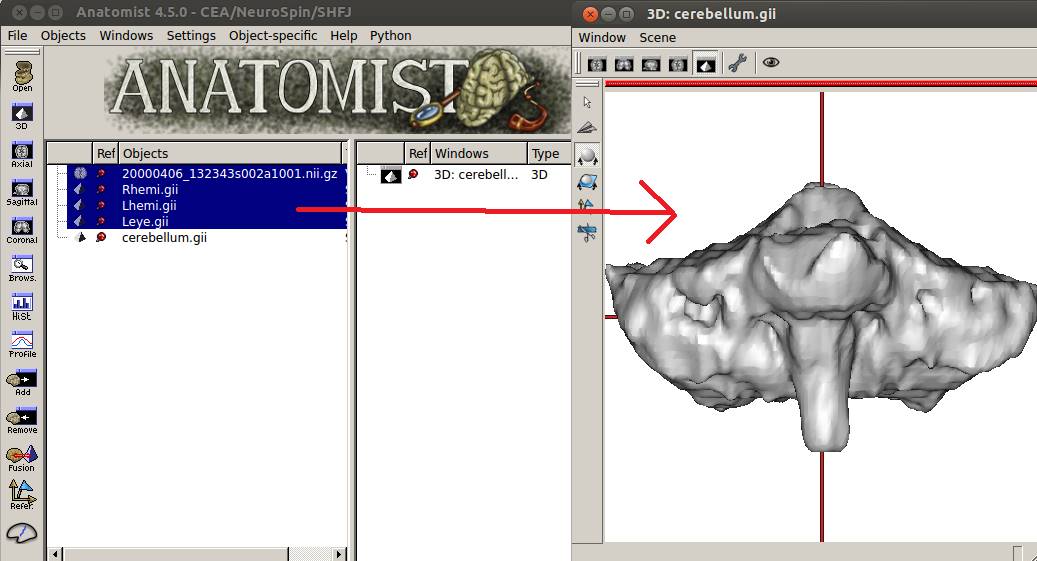
小脳と左眼球が表示されています。また
元データの断面とメッシュが一致していることが確認できました。