SPMは頭蓋骨が長頭型の欧米人を雛形としていて、短頭型のアジア人だと左右方向の情報がつぶれているかもしれません。
そこで雛形を横に伸ばしてみました。
NiBabelで雛形を横に膨らませる
雛形は~/spm12/tpm/TPM.niiです。pythonのNiBabelで読み込んで、Scipyで横方向に伸ばしてみます。Pythonのコードは以下の通りです。
import numpy as np
import matplotlib.pyplot as plt # 描画用
import scipy.interpolate as interpolate
import nibabel as nib # NiBabelの導入の確認 エラーが出なければ成功
pathname='C:/170207/spm12/tpm/' # 著者のSPMのあるフォルダの場所です。
filename='TPM.nii'
nii1=nib.load(pathname+filename)
data1=nii1.get_data()
print(data1.shape)
print(data1.dtype)
(121L, 145L, 121L, 6L)
float32
# 頭蓋骨を横に膨らませる
data2=np.zeros((131,145,121,6),dtype=np.float32)
# 4次元配列です
x0=range(0,data1.shape[0])
y0=range(0,data1.shape[1])
x1=np.linspace(0,data1.shape[0]-1,131)
y1=np.linspace(0,data1.shape[1]-1,145)
for i3 in range(0,data1.shape[3]):
for i2 in
range(0,data1.shape[2]):
f=interpolate.interp2d(x0,y0,data1[:,:,i2,i3].T,kind='linear') # 画像の時はy軸とx軸が逆転するようです。転置で調整します。
data2[:,:,i2,i3]=f(x1,y1).T
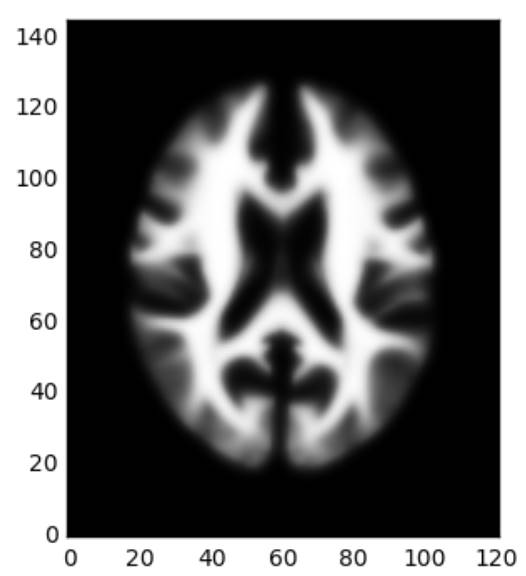
plt.imshow(data1[:,:,61,1].T,cmap='gray',origin='lower')
plt.show()
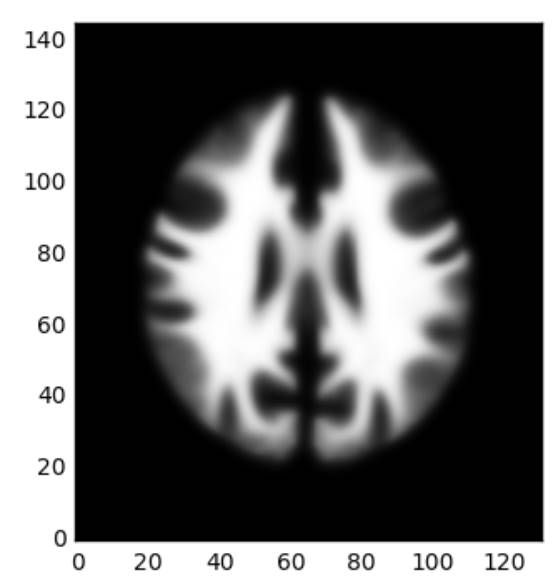
plt.imshow(data2[:,:,66,1].T,cmap='gray',origin='lower')
plt.show()
# NIfTIファイルとして保存
nii2=nib.Nifti1Image(data2,affine=nii1.affine)
nib.save(nii2,pathname+'newTPM.nii')
SPM12で横長雛形を使って標準化
自分の三次元MRIデータをNIfTI形式のakira.niiという名前にします。
Image to AlignとImages to Writeは両方ともakira.niiです。
Tissue probability mapはNiBabelで作成した横長雛形newTPM.niiです。121
Bounding boxをクリックします。
これはx、y、z軸方向のトリミングの幅です。横長なのでx軸方向の値をいろいろ試してみてください。

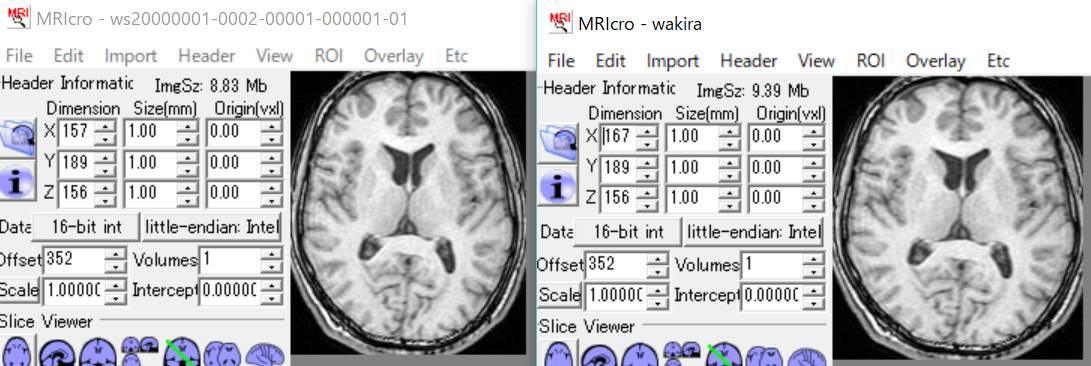
MRIcroで結果を見てみます。左が通常の雛形で標準化した場合、右が横長の雛形で標準化した場合です。
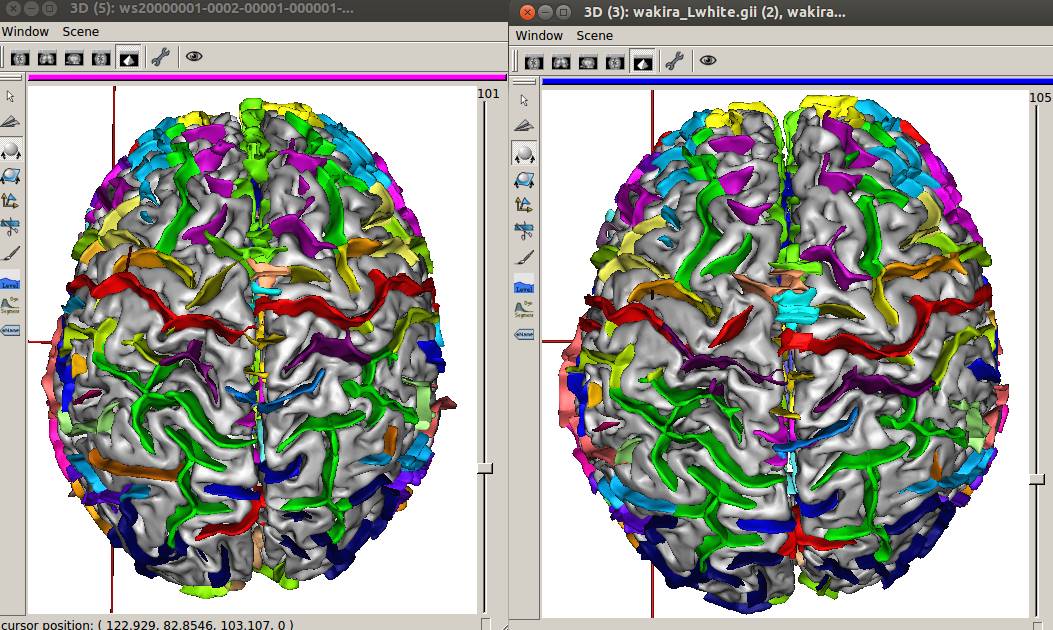
BrainVISAで脳溝抽出をしてみました。
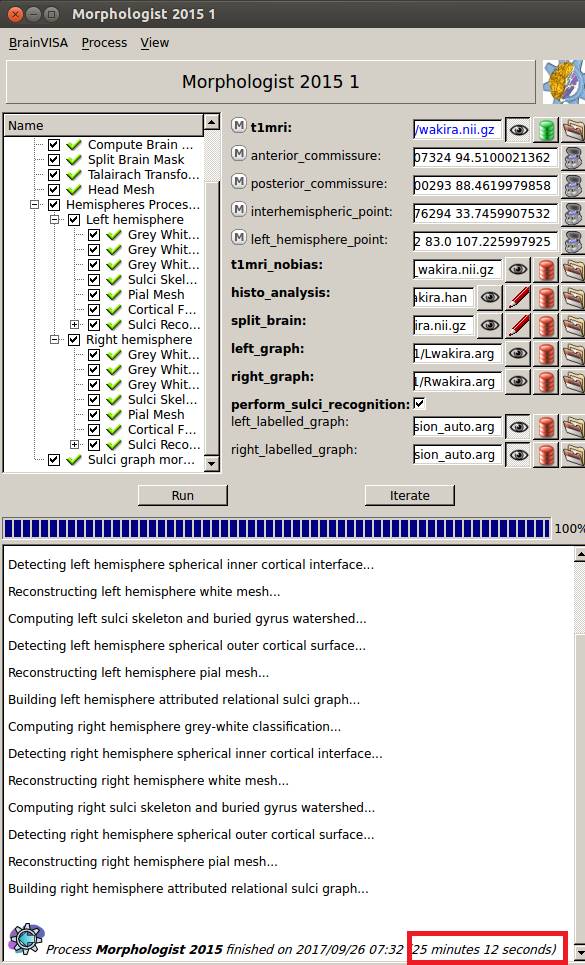
25分かかりました。
左が通常の標準化、右が横長の標準化です。脳溝はちゃんと抽出しているようですが、左中心溝の同定が異なっているようです。