BrainVISAの結果をMNE-Cで使う方法2
「BrainVISAの結果をMNE-Cで使う方法」http://meg.aalip.jp/python/BrainVISA_MNE.htm
はMATLABを使いましたが、そのPython版です。
変更点のみ記しています。
NiBabelのインストール
PythonでNIfTIやGIfTIの読み書きを行うにはNiBabelを使います。
本来Python 3.xでは三次元表示をおこなうPython2.7用のMayaviを使用するのは難しいのですが、MNE-python 0.16以降をインストールすることで解決しています。MNE-python 0.16がインストールされている状態のPython 3.6以降での話とします。Python 2.7を使う場合にはMNE-python 0.16のインストールは不要です。
Anaconda Promptを起動します。
Anacondaとpythonのversionです。
(base)
C:\Users\akira>conda --version && python --version
conda 4.5.11
Python
3.6.5 :: Anaconda, Inc.
NiBabelをインストールします。途中でyを押します。
(base)
C:\Users\akira>conda install -c conda-forge nibabel
Solving
environment: done
##
Package Plan ##
environment location:
C:\Users\akira\Anaconda3.6
added / updated specs:
- nibabel
The
following packages will be downloaded:
package
|
build
---------------------------|-----------------
pydicom-1.1.0
|
py_0
5.2 MB conda-forge
certifi-2018.4.16
|
py36_0
143 KB conda-forge
nibabel-2.3.0
| pyh24bf2e0_1
2.7 MB conda-forge
conda-4.5.11
|
py36_0
654 KB conda-forge
------------------------------------------------------------
Total:
8.7 MB
The
following NEW packages will be INSTALLED:
nibabel:
2.3.0-pyh24bf2e0_1 conda-forge
pydicom:
1.1.0-py_0
conda-forge
The
following packages will be UPDATED:
certifi:
2018.4.16-py36_0
--> 2018.4.16-py36_0 conda-forge
conda: 4.5.11-py36_0
--> 4.5.11-py36_0
conda-forge
Proceed
([y]/n)? y
Downloading
and Extracting Packages
pydicom-1.1.0 |
5.2 MB |
############################################################################ |
100%
certifi-2018.4.16 | 143 KB | ############################################################################
| 100%
nibabel-2.3.0 |
2.7 MB |
############################################################################ |
100%
conda-4.5.11
| 654 KB |
############################################################################ |
100%
Preparing
transaction: done
Verifying
transaction: done
Executing
transaction: done
(base)
C:\Users\akira>
動作確認をします。
(base)
C:\Users\akira>python
Python
3.6.5 |Anaconda, Inc.| (default, Mar 29 2018, 13:32:41) [MSC v.1900 64 bit
(AMD64)] on win32
Type
"help", "copyright", "credits" or
"license" for more information.
>>>
import nibabel
C:\Users\akira\Anaconda3.6\lib\site-packages\h5py\__init__.py:36:
FutureWarning: Conversion of the second argument of issubdtype from `float` to `np.floating`
is deprecated. In future, it will be treated as `np.float64 == np.dtype(float).type`.
from ._conv import register_converters
as _register_converters
>>>
認識されました。
Spyder (mne)の起動
mneをactivateしたSpyder (mne)を起動します。
以下を実行します。
fimport numpy as np
import matplotlib.pyplot
as plt #描画用
import nibabel as
nib
結果です。
Python
3.6.6 |Anaconda, Inc.| (default, Jun 28 2018, 11:27:44) [MSC v.1900 64 bit
(AMD64)]
Type
"copyright", "credits" or "license" for more
information.
IPython 6.5.0 -- An enhanced Interactive Python.
In
[1]: from mayavi import mlab
...: import numpy
as np
...: import matplotlib.pyplot
as plt #描画用
...: import nibabel
as nib
MayaviのバックエンドをQt4に変更しています
In
[2]:
NIfTIの書き換え
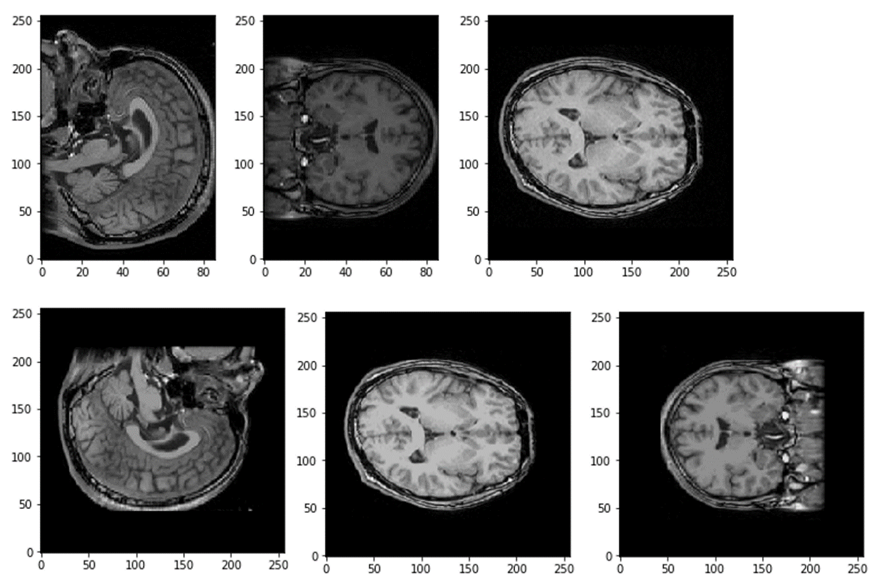
BrainVISAとFreeSurferの座標の違いを示します。以下を実行します。
a_T1=nib.load('c:\\test\\a\\mri\\T1.nii.gz')
a_brain=nib.load('c:\\test\\a\\mri\\brain.nii.gz')
b_T1=nib.load('c:\\test\\b\\mri\\T1.nii.gz')
for T1 in [a_T1,b_T1]:
scale=np.linalg.norm(T1.affine[0:3,0:3],axis=1)
img0=T1.get_data()
sz=round(img0.shape[0]/2)
aspect=scale[1]/scale[2]
plt.imshow(img0[sz,:,:],aspect=aspect,cmap='gray',origin='lower')
plt.show()
sz=round(img0.shape[1]/2);
aspect=scale[0]/scale[2]
plt.imshow(img0[:,sz,:],aspect=aspect,cmap='gray',origin='lower')
plt.show()
sz=round(img0.shape[2]/2);
aspect=scale[0]/scale[1]
plt.imshow(img0[:,:,sz],aspect=aspect,cmap='gray',origin='lower')
plt.show()
マスクa_brainと頭部a_T1を掛け合わせ、脳実質を抽出データに変換します。
img0=a_T1.get_data()
img1=a_brain.get_data()
img1[img1>0]=1
img1[img1<1]=0
img1=img1*img0
NIfTIデータの書き換えを行います。MATLABのniftiwrite関数と違ってnib.save(データ、アッフィン変換)だけでOKです。
sz=img0.shape
new_data=np.arange(img0.size,dtype=np.int16)
new_data=new_data.reshape([sz[0],sz[2],sz[1]])
data=img0
for z in range(sz[2]):
new_data[:,z,:]=data[:,:,sz[2]-z-1]
affine=a_T1.affine.copy().T
pixscale=np.linalg.norm(affine,axis=1)
affine[:,1]=a_T1.affine[2,:]
affine[:,2]=a_T1.affine[1,:]
affine[2,1]=-affine[2,1]
affine[3,1]=-affine[3,1]+affine[2,1]
affine[:,0:2]=-affine[:,0:2]
affine[0,0]=-affine[0,0]
affine[3,0]=data.shape[0]*scale[0]/2;
affine=affine.T
array_img=nib.Nifti1Image(new_data,affine)
nib.save(array_img,'c:\\test\\a\\mri\\newT1.nii.gz')
data=img1
for z in range(sz[2]):
new_data[:,z,:]=data[:,:,sz[2]-z-1]
array_img=nib.Nifti1Image(new_data,affine)
nib.save(array_img,'c:\\test\\a\\mri\\newbrain.nii.gz')
確認してみます。
for T1 in [a_newT1,b_T1]:
scale=np.linalg.norm(T1.affine[0:3,0:3],axis=1)
img0=T1.get_data()
sz=round(img0.shape[0]/2)
aspect=scale[1]/scale[2]
plt.imshow(img0[sz,:,:],aspect=aspect,cmap='gray',origin='lower')
plt.show()
sz=round(img0.shape[1]/2);
aspect=scale[0]/scale[2]
plt.imshow(img0[:,sz,:],aspect=aspect,cmap='gray',origin='lower')
plt.show()
sz=round(img0.shape[2]/2);
aspect=scale[0]/scale[1]
plt.imshow(img0[:,:,sz],aspect=aspect,cmap='gray',origin='lower')
plt.show()
座標が揃いました。
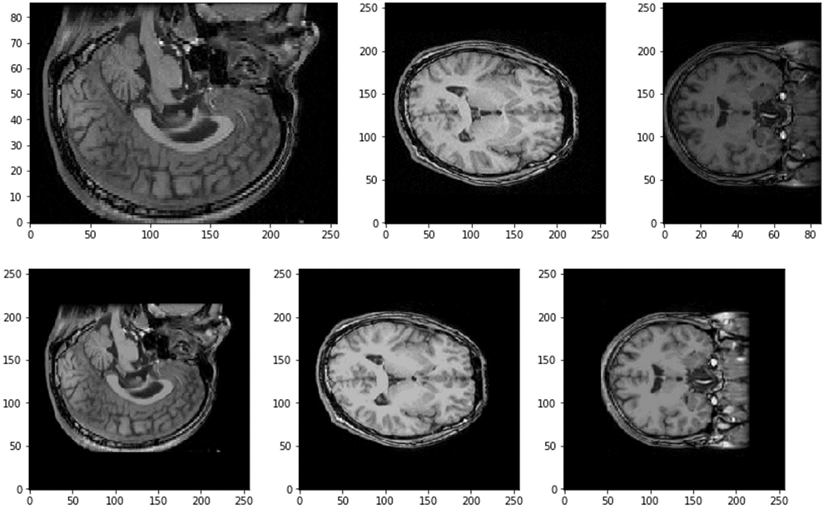
GIfTIの書き換え
BrainVISAとFreeSurferの座標の違いを示します。
from mayavi import mlab
import numpy as np
import nibabel as
nib
a_T1=nib.load('c:\\test\\a\\mri\\T1.nii.gz')
a_Lwhite=nib.load('C:\\test\\a\\surf\\Lwhite.gii')
a_Rwhite=nib.load('C:\\test\\a\\surf\\Rwhite.gii')
b_Lwhite=nib.load('C:\\test\\b\\surf\\lh.white.gii')
b_Rwhite=nib.load('C:\\test\\b\\surf\\rh.white.gii')
mlab.figure(bgcolor=(1,1,1))
# BrainVISA x座標は反転
p=a_Lwhite.darrays[0].data
mesh=a_Lwhite.darrays[1].data
mlab.triangular_mesh(-p[:,0],p[:,1],p[:,2],mesh,color=(0,0,1))
p=a_Rwhite.darrays[0].data
mesh=a_Rwhite.darrays[1].data
mlab.triangular_mesh(-p[:,0],p[:,1],p[:,2],mesh,color=(0,1,0))
# FreeSurfer x座標は反転せず
p=b_Lwhite.darrays[0].data
mesh=b_Lwhite.darrays[1].data
mlab.triangular_mesh(p[:,0],p[:,1],p[:,2],mesh,color=(0,0.5,1))
p=b_Rwhite.darrays[0].data
mesh=b_Rwhite.darrays[1].data
mlab.triangular_mesh(p[:,0],p[:,1],p[:,2],mesh,color=(0,1,0.5))
mlab.show()
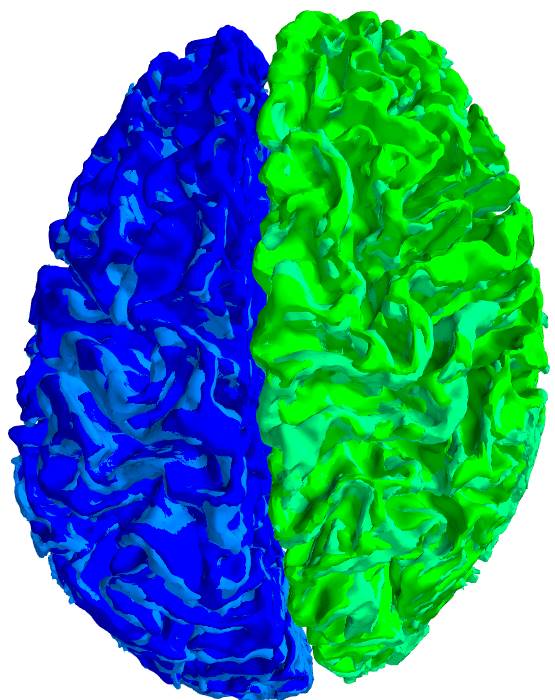
上がBrainVISA、下がFreeSurferです。
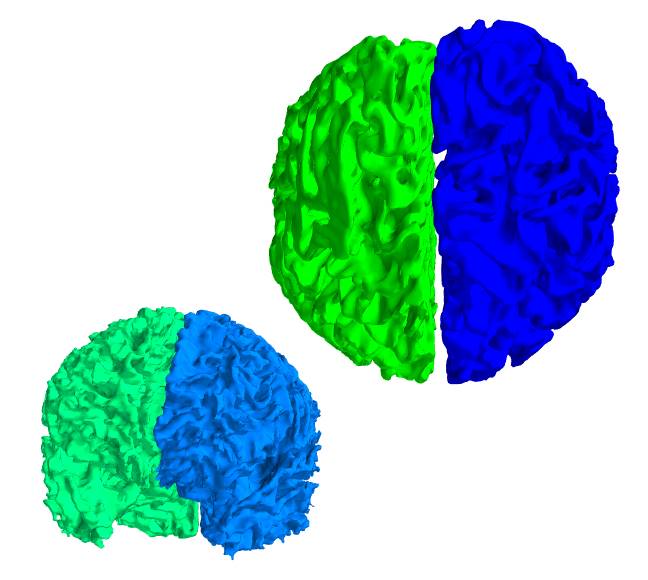
座標変換を行います。
dsize=np.array(np.mat(a_T1.shape))
pixscale=np.linalg.norm(a_T1.affine[0:3,0:3],axis=1)
d=dsize*pixscale
#
X0=np.array([[1,0,0,-d[0,0]/2],[0,1,0,-d[0,1]/2],[0,0,1,-d[0,2]/2],[0,0,0,1]])
X0=np.array([[1,0,0,0],[0,1,0,0],[0,0,1,0],[-d[0,0]/2,-d[0,1]/2,-d[0,2]/2,1]])
X1=np.array([[-1.,0,0,0],[0,-1,0,0],[0,0,-1,0],[0,0,0,1]])
X2=np.dot(X0,X1)
for surf in [a_Lwhite,a_Rwhite]:
p=np.ones((surf.darrays[0].data.shape[0],4))
p[:,0:3]=surf.darrays[0].data.copy()
p=np.dot(p,X2)
surf.darrays[0].data[:,:]=p[:,0:3]#
data=p[:,0:3]は不可
f=surf.darrays[1].data.copy()
surf.darrays[1].data[:,1]=f[:,2]
surf.darrays[1].data[:,2]=f[:,1]
nib.save(a_Lwhite,'C:\\test\\a\\surf\\newLwhite.gii')
nib.save(a_Rwhite,'C:\\test\\a\\surf\\newRwhite.gii')
確認します。
a_newLwhite=nib.load('C:\\test\\a\\surf\\newLwhite.gii')
a_newRwhite=nib.load('C:\\test\\a\\surf\\newRwhite.gii')
mlab.figure(bgcolor=(1,1,1))
# BrainVISAを座標変換 x座標は反転させず
p=a_newLwhite.darrays[0].data
mesh=a_newLwhite.darrays[1].data
mlab.triangular_mesh(p[:,0],p[:,1],p[:,2],mesh,color=(0,0,1))
p=a_newRwhite.darrays[0].data
mesh=a_newRwhite.darrays[1].data
mlab.triangular_mesh(p[:,0],p[:,1],p[:,2],mesh,color=(0,1,0))
# FreeSurfer x座標は反転せず
p=b_Lwhite.darrays[0].data
mesh=b_Lwhite.darrays[1].data
mlab.triangular_mesh(p[:,0],p[:,1],p[:,2],mesh,color=(0,0.5,1))
p=b_Rwhite.darrays[0].data
mesh=b_Rwhite.darrays[1].data
mlab.triangular_mesh(p[:,0],p[:,1],p[:,2],mesh,color=(0,1,0.5))
mlab.show()
座標が一致し、BrainVISAとFreeSurferのポリゴンメッシュがほぼ重なりました。ポリゴンメッシュそのものは異なっているので脳回の模様に微妙にムラがあります。