内容
NiBabelやAimsSubSamplingで256×256に変更したデータで実行
512×512画素は・・・
512×512の画像からなるnii.gzファイルをデータベースに登録します。
Data Managementのimport→T1 MRI→import T1 MRIをダブルクリックします。
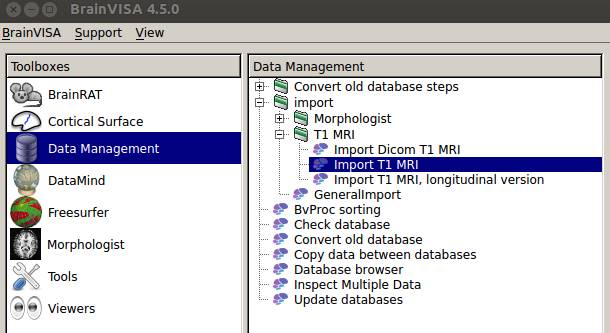
nii.gzファイルを選択してRunを押すと![]() が2つも出ます。
が2つも出ます。
BrainVISAは256×256仕様です。
一応登録はでき![]() でAnatomistが起動し、画像は表示されます。
でAnatomistが起動し、画像は表示されます。
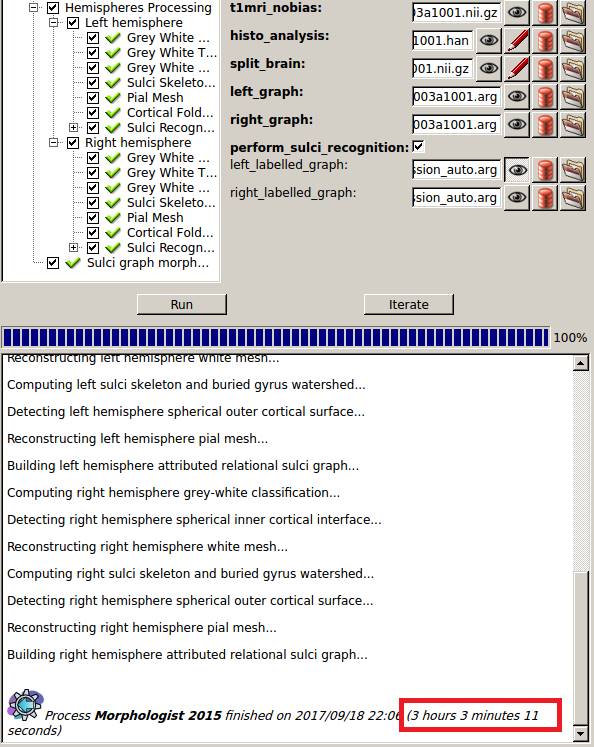
Morphologist 2015を起動します。

512×512のままRunを押すと、T1 Bias Correctionでエラーが出て止まります。
ん? の筈が時間かけたら(数時間)次の処理が開始し、Passしてしまいました。
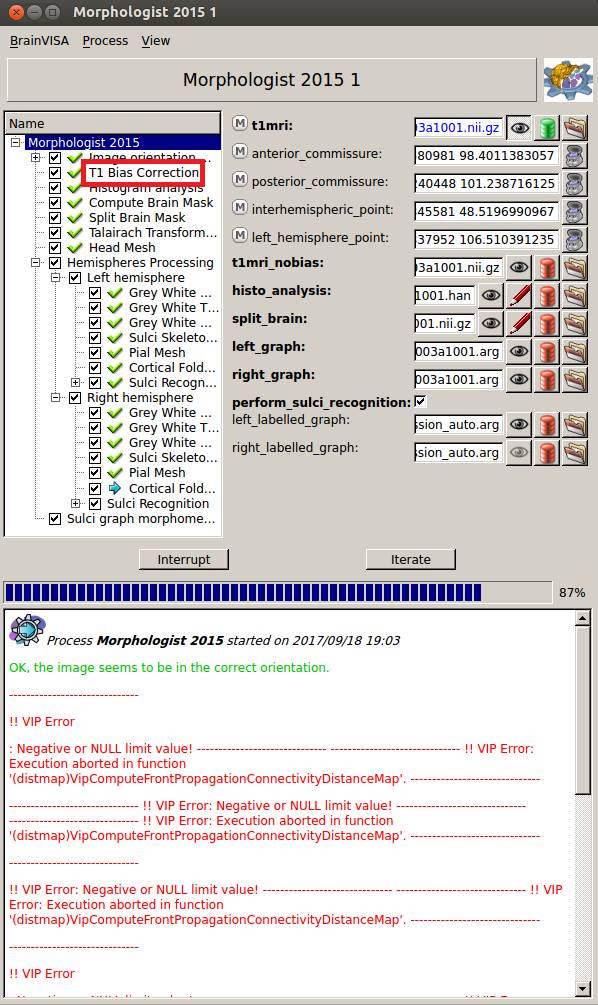
話の流れは、ここで512×512を256×256に変更して・・・となるので、そうします。
512×512の続きはあとでにします。
ToolsにSubsamplingというのがあります。
よさそうなので使ってみたのですが・・・
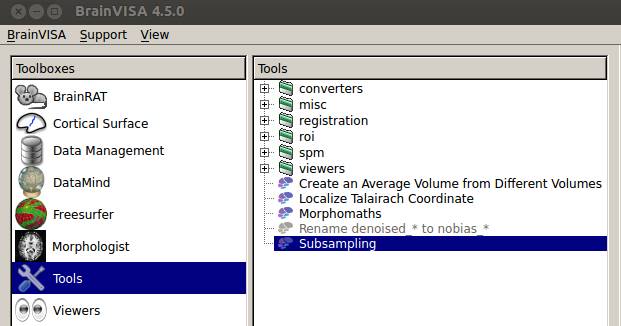
うまくいきませんでした。
いろいろ試したんですが・・・諦めました。
AimsSubSamplingで256×256に
SubSamplingの本体はたぶん/opt/brainvisa/bin/AimsSubSamplingです。
/opt/brainvisa/bin/AimsSubSampling -i 〇〇.nii.gz -o ●●.nii.gz -rx 2 -ry 2 -rz 1
でx軸、y軸の画素数が半分にz軸はそのままになります。

BrainVISAのAnatomistで確認しました。
Browseにドラッグします。
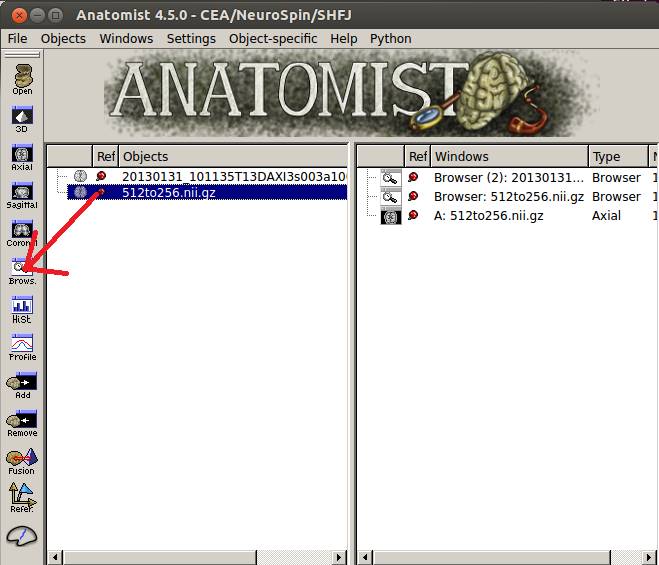
左が元データ、右が変換後データです。sizeXとsizeYの値が512から256になっています。
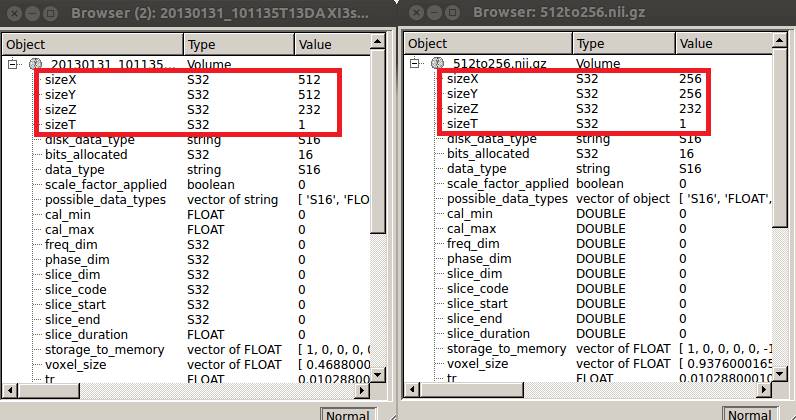
AimsSubSamplingでは解像度を整数分の1にしか変換できません。PythonのpackageのNiBabelを使うと、任意の解像度に変換できます。
詳細はNiBabelの導入のところを参照してください。
MRIcroで256×256に
一番簡単なのはMRIcroを使う方法です。残念ながらMRIcroはWindows版しかありません。
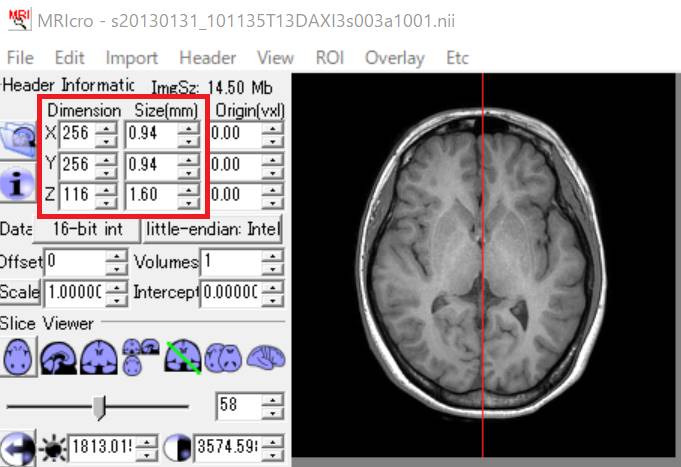
〇〇.nii.gzを読み込むと512×512×232画素の三次元配列、1画素の大きさ0.47×0.47×0.80 mmと表示されます。

Etc→Rescaleを選択します。
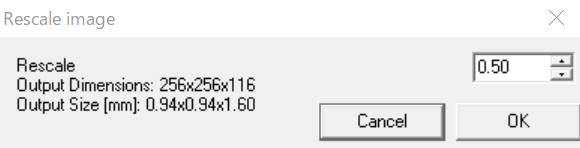
値を0.50にすると画素数は半分の256×256×116、画素の大きさは2倍の0.94×0.94×1.60となります。
OKを押します。
OKを押します。
2つファイルが作成されます。初期設定ではsがファイル名先頭に付加され、拡張子が.hdrと.imgのNIfTIの古い形式Analyze形式で保存されます。
MRIcroで開いてみます。
UbuntuにファイルをコピーしてImport T1で〇.imgを選択してデータベースに登録してみました。
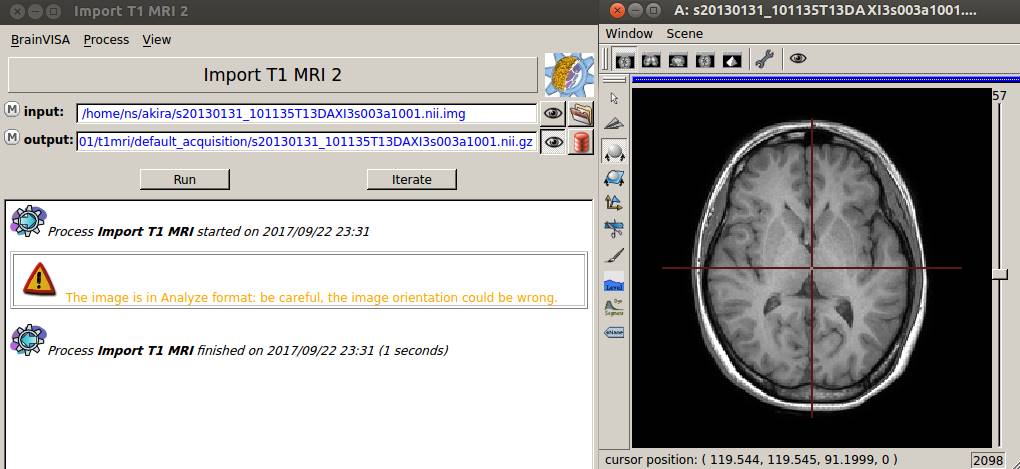
警告メッセージは出ますが、問題はないようです。
NiBabelやAimsSubSamplingで256×256に変更したデータで実行
MRIcroだとz軸の解像度まで半分になってしまうのでNiBabelでx軸とy軸方向だけを半分の256にしたデータ作成しました。データベース登録後Morphologist 2015を実行すると問題なく30分弱で処理が終了しました。
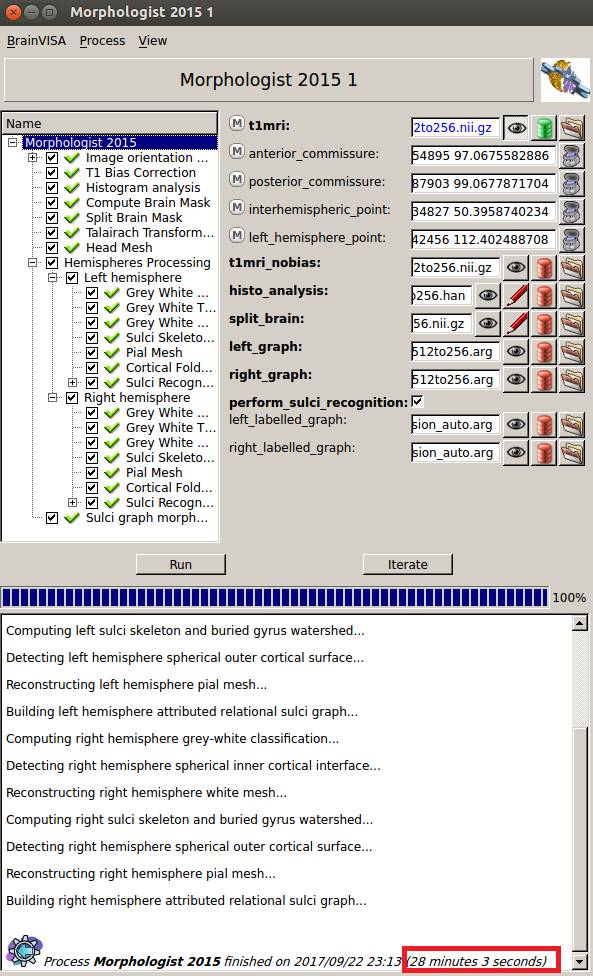
Anatomistで確認しました。大丈夫そうです。
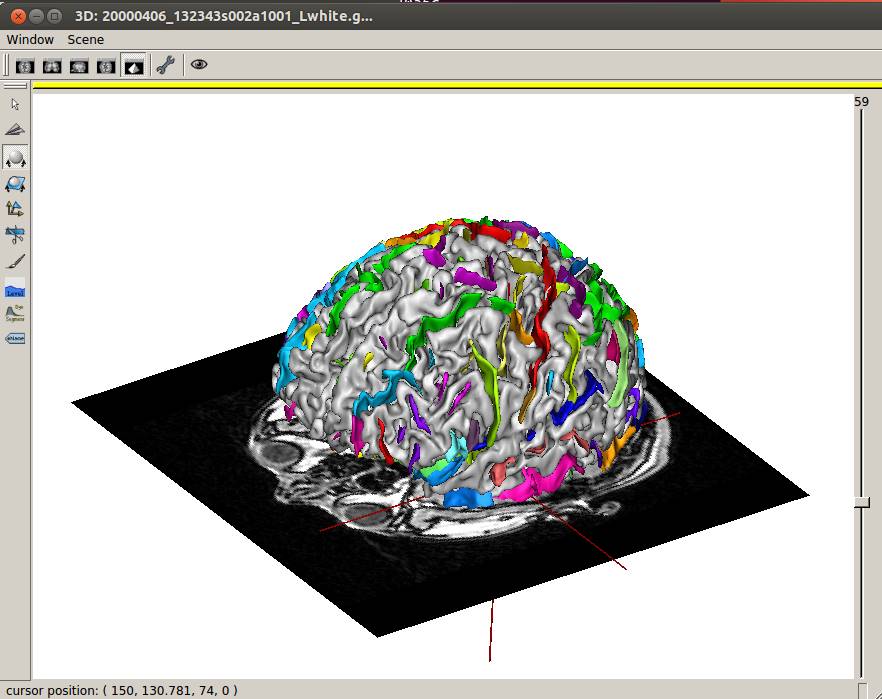
512×512の続き
512×512の続きです。本題ではないので無視して結構です。
計算途中ですが、left_labelled_graph:の![]() をクリックしてみます。
をクリックしてみます。
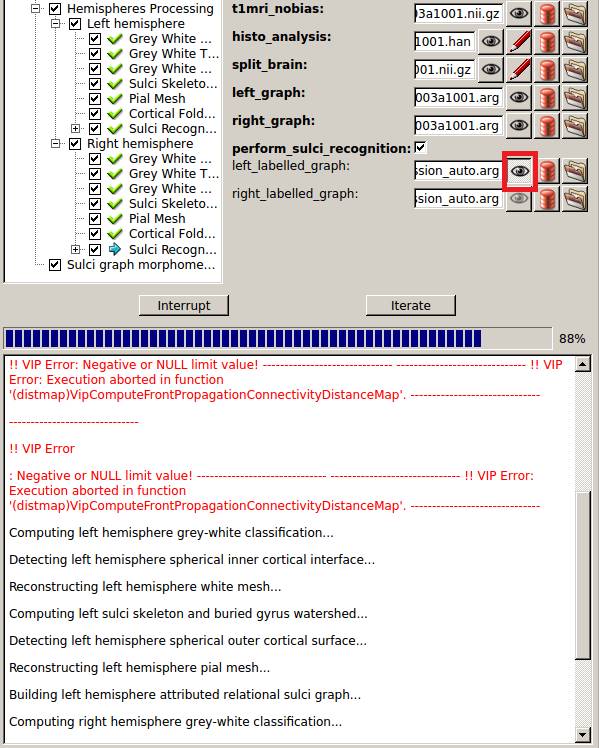
ちゃんとできてるっぽいです。
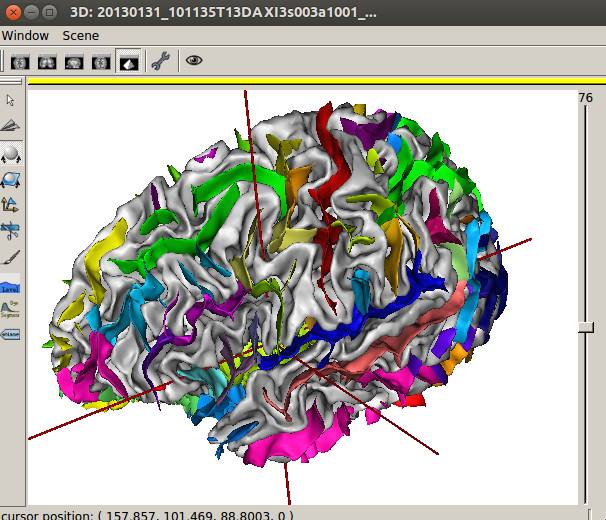
計算途中でもright_labelled_graph:の目玉が灰色から黒になったのでクリック。
なんかできたっぽい。
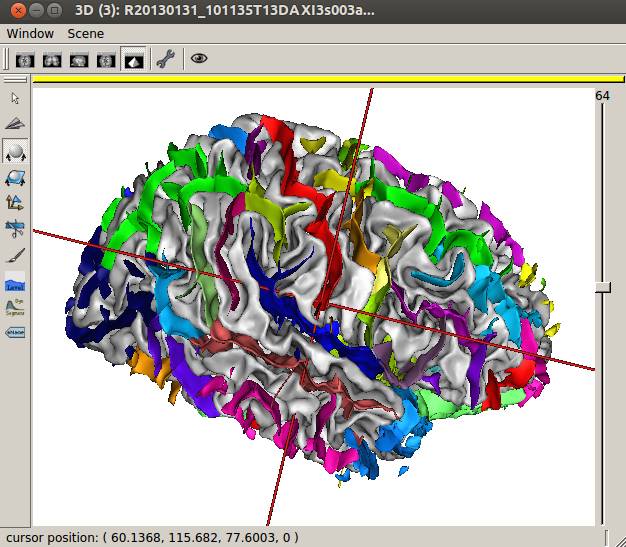
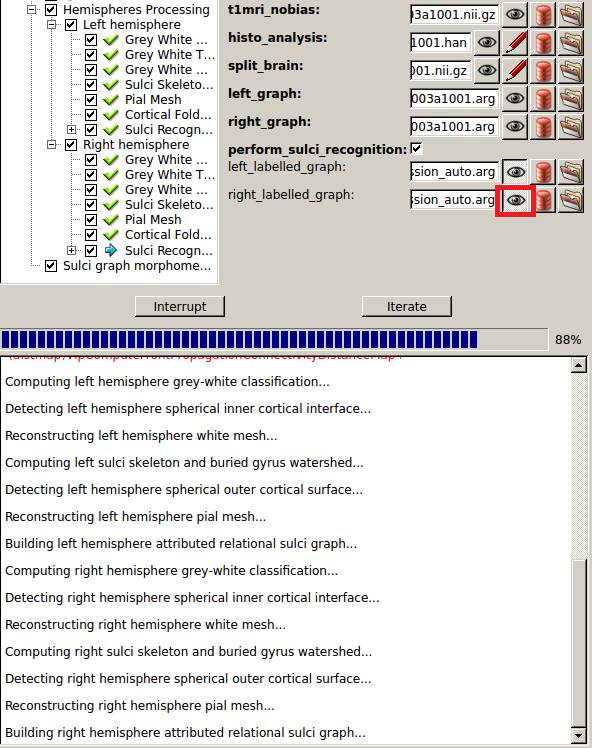
無事終了。3時間かかりました。