MNE実行とdSPM
SEFのデータを使ってみます。
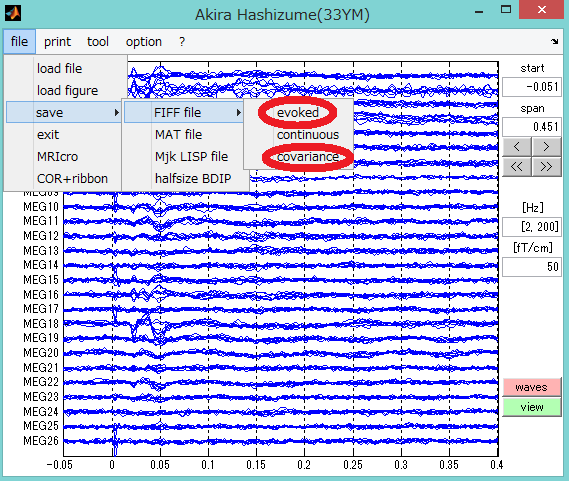
hns_megを起動し、SEFのFIFFデータを読み込み、
evokedを選択してSEFの脳磁図データを保存、covarianceを選択して-0.05〜0.4秒のデータの分散行列を保存します。
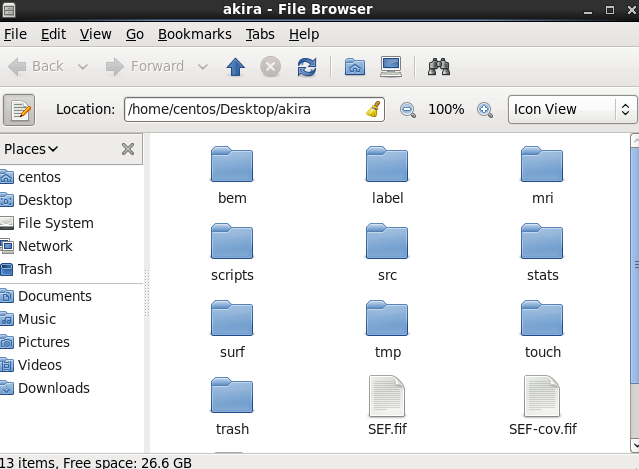
保存した2つのFIFファイルを、CentOSのデスクトップのakiraフォルダにコピーします。
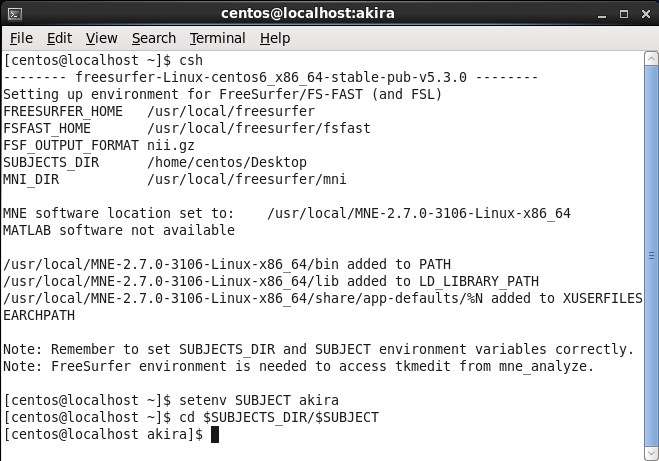
terminalを開き、
cshと入力し、
setenv SUBJECT akiraとタイプし、
cd $SUBJECTS_DIR/$SUBJECTと入力します。
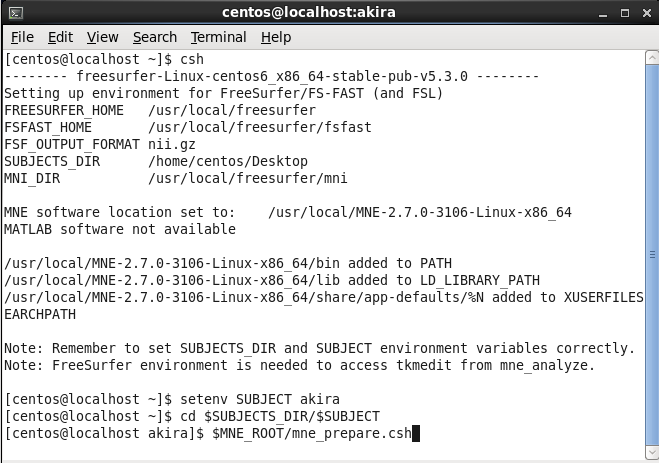
$MNE_ROOT/mne_prepare.cshと入力します。
実行中。時間がかかります。
Fine segmentationの辺り、時間かかってます。
終了後、mne_analyzeと入力します。
mne_analyzeの画面が表示されます。
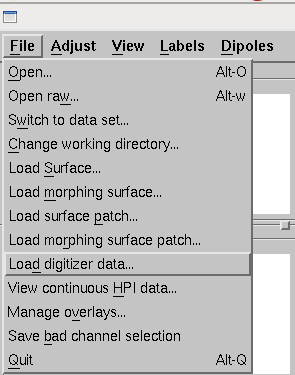
File→Load Digitizer data...を選択します。
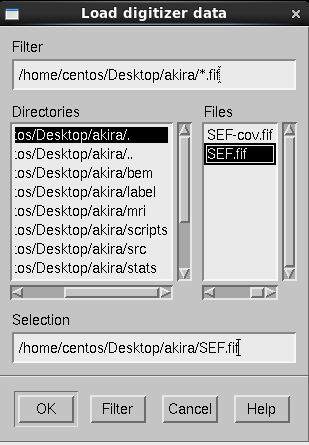
SEF.fifを選択し、OKボタンを押します。
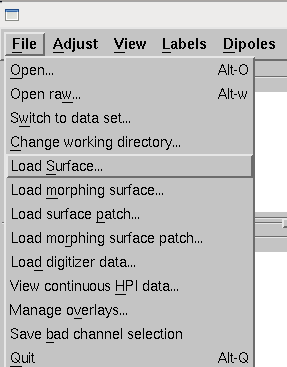
File→Load Surface...を選択します。
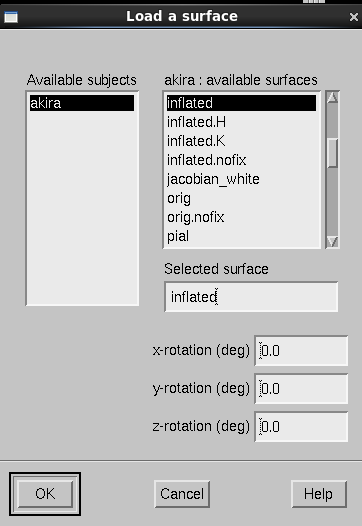
inflatedを選択し、OKボタンを押します。
View→Show Viewerを選択します。
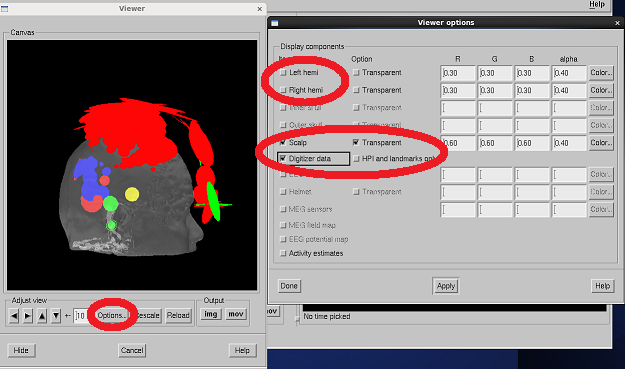
Options..ボタンを押し、Left hemiとRight hemiのチェックをOFFにし、
Scalp、Transparent、Digitizer dataのチェックをONにし、Doneボタンを押します。
Adjust→Coordinate alignment...を選択します。
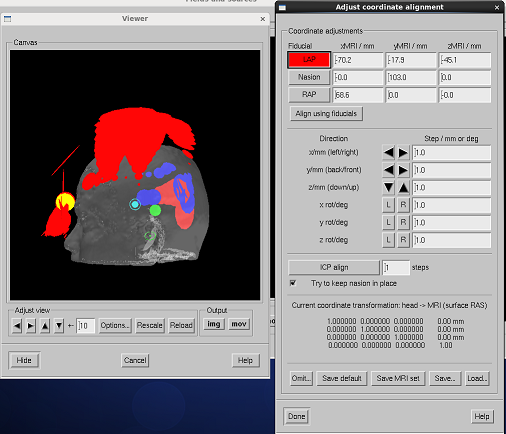
LAPをクリックし、Viewer上で左耳介の基準点付近をクリックします。
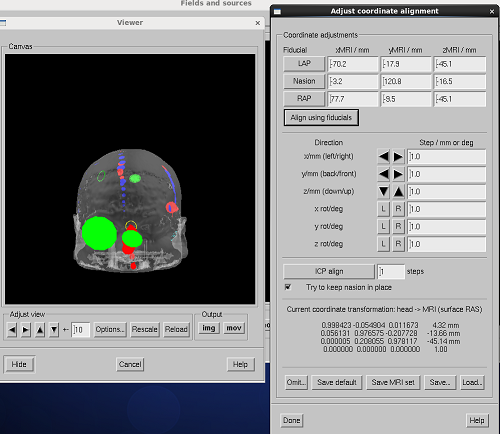
NAS、RAPもクリックして基準点座標を取得し、Align using fiducialsボタンをクリックします。
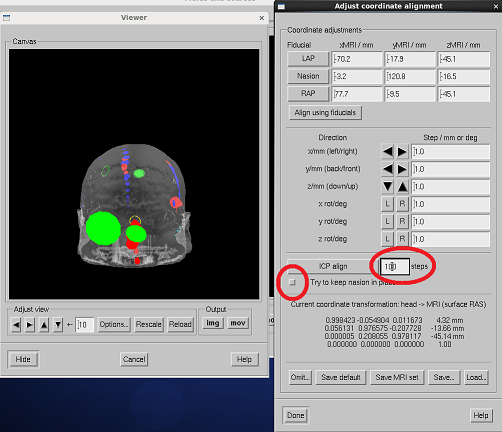
stepを100回にし、Try to keep nasion in placeのチェックをOFFにし、ICP alignボタンを押します。
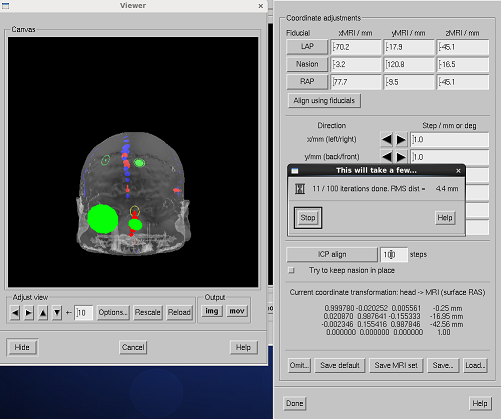
微調整中。

終了後、角度を90にして、三次元画像を回転させ、▼やLやRボタンを押してさらに微調整します。
納得できればSave defaultとSave MRI setを押した後、Doneボタンを押します。

Doneを押すと基準点、HPI、Digitized点の座標は座標合わせ前に戻ります。
File→Quitを選択してmne_analyzeを終了する。
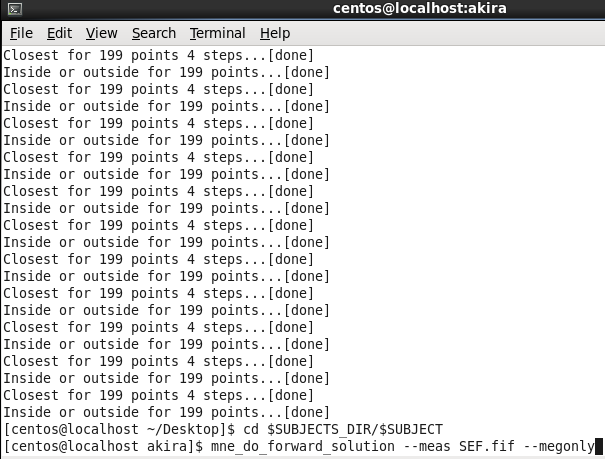
cd $SUBJECTS_DIR/$SUBJECTとし、
mne_do_forward_solution --meas SEF.fif --megonlyで順問題を解きます。
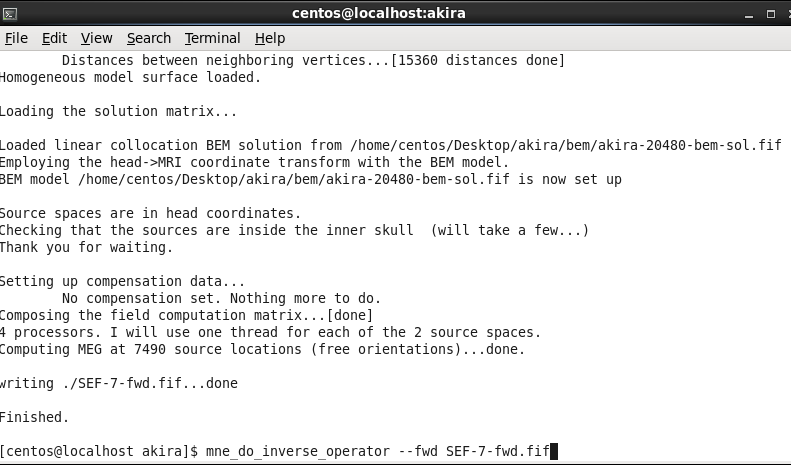
mne_do_inverse_operator --fwd SEF-7-fwd.fif で逆問題を解きます。
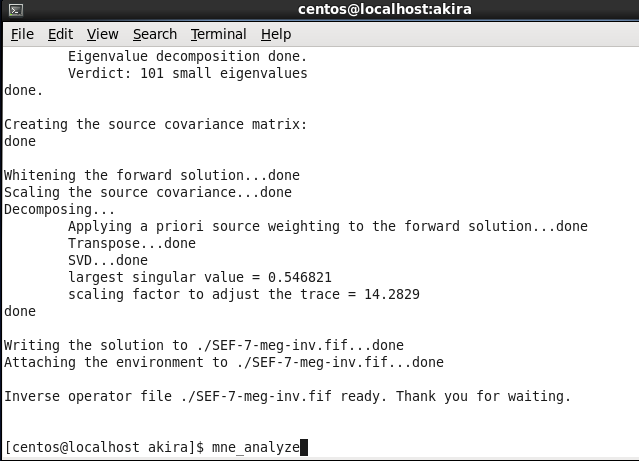
mne_analyzeでmne_analyzeを起動します。
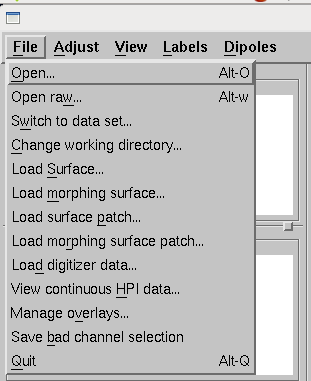
File→Openを選択します。
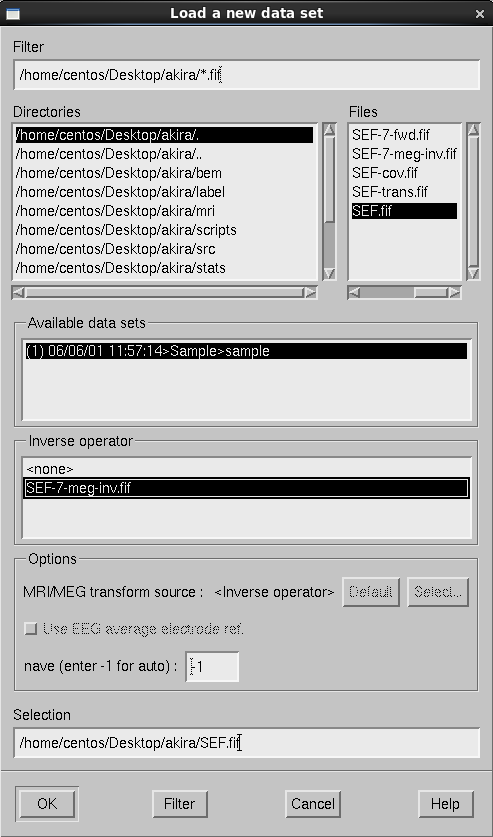
FilesはSEF.fifを、inverse operatorはSEF-7-meg-inv.fifを選択し、OKボタンを押します。
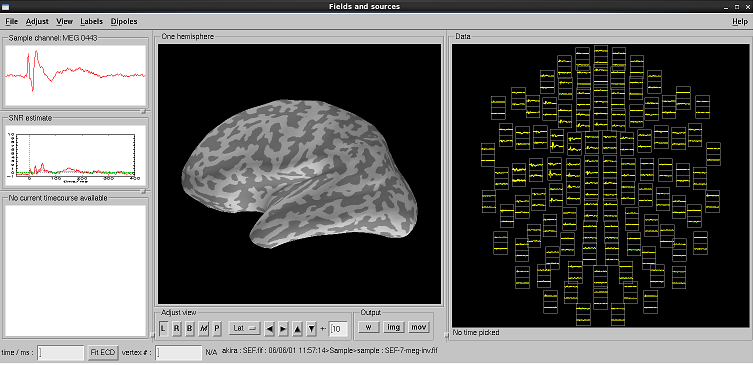
画像が表示されます。
Adjust→Estimates...を選択します。
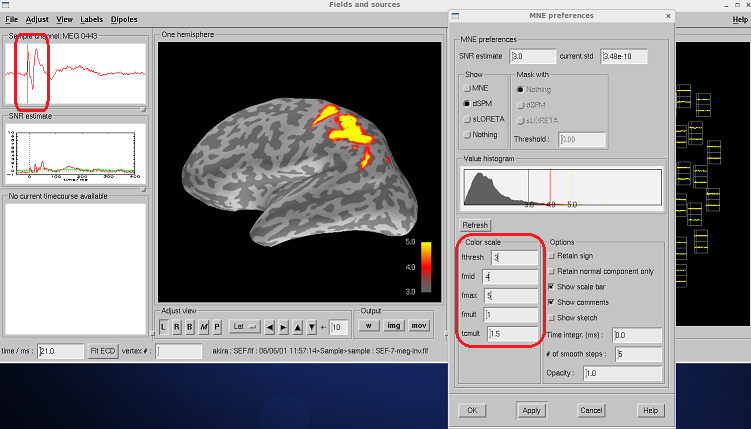
MNE preferenceのColor scaleを適当に設定し、左上の頂点付近をクリックするとSEFの20msのdSPMの電流解が表示されます。
File→Load Surface...を選択します。
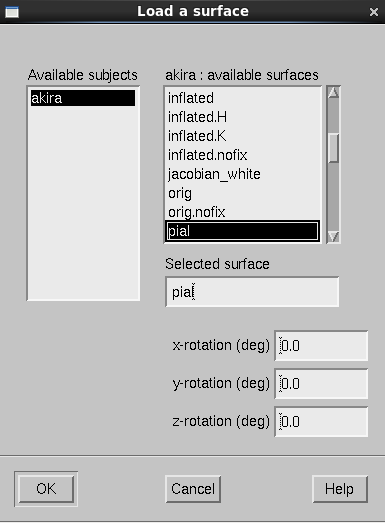
pialを選択し、OKボタンを押します。
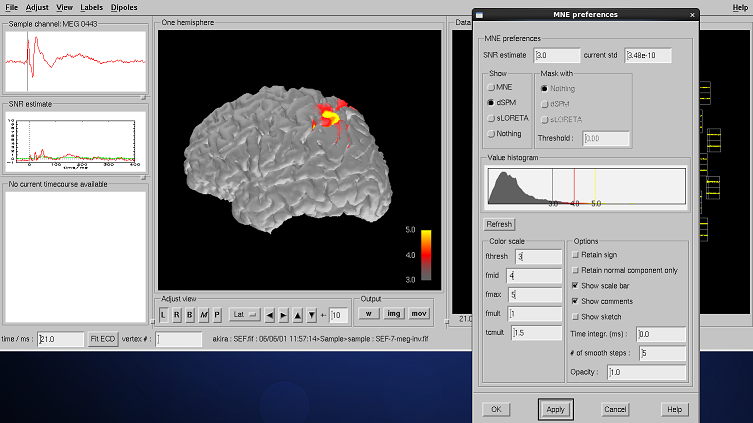
Applyボタンを押します。
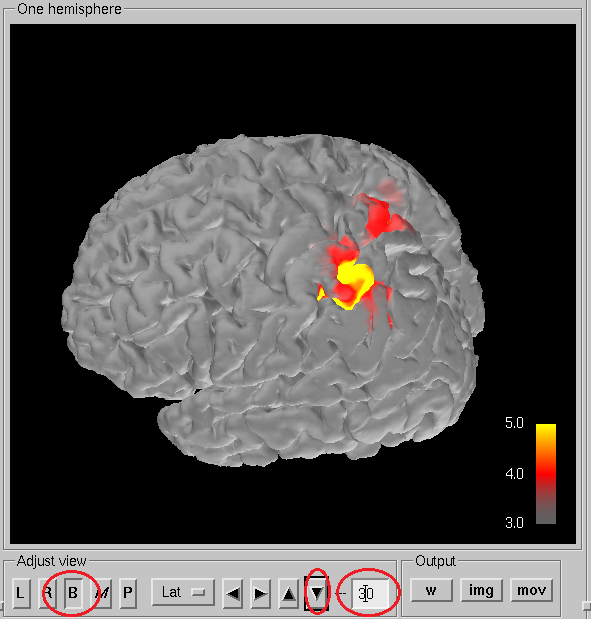
Bボタンを押して、30として▼ボタンを押します。
































