Contents
NIfTIファイルの読み書き
%Matlab R2017bからimage processing toolboxでNIfTIファイルの読み書きができるようになりました。 %残念ながらGIfTIファイルには対応していません。 % A MATLAB GIfTI library % https://www.artefact.tk/software/matlab/gifti/ % のgifti-1.6.zip (v1.6)を使うことにします。 % 但し、GIfTIファイルの一部は読めないようです。 clear; pathname='c:/users/akira/'; filename='20000406_132343s002a1001.nii.gz'; data1=niftiread([pathname,filename]);% データ本体 header=niftiinfo([pathname,filename]);
ヘッダーの詳細
構造体になっています。 header.Transform.Tが変換行列、 header.raw.dimが配列の大きさ、 header.raw.pixdimがボクセルの大きさ、 になっています。
header % ヘッダー header.Transform.T % 変換行列 header.raw.dim % 最初は〇次元配列の次数 header.raw.pixdim
header =
フィールドをもつ struct:
Filename: 'c:\users\akira\20000406_132343s002a1001.nii.gz'
Filemoddate: '17-9-2017 08:14:51'
Filesize: 4426587
Description: 'TE=2.799999952;sec=0.0000'
ImageSize: [256 256 86]
PixelDimensions: [0.9375 0.9375 2]
Datatype: 'int16'
BitsPerPixel: 16
SpaceUnits: 'Millimeter'
TimeUnits: 'Second'
AdditiveOffset: 0
MultiplicativeScaling: 1
TimeOffset: 0
SliceCode: 'Unknown'
FrequencyDimension: 0
PhaseDimension: 0
SpatialDimension: 0
DisplayIntensityRange: [0 0]
TransformName: 'Sform'
Transform: [1×1 affine3d]
Qfactor: -1
raw: [1×1 struct]
ans =
-0.9375 0 0 0
0 0.9375 0 0
0 0 2.0000 0
120.0000 -106.6625 -67.0000 1.0000
ans =
3 256 256 86 1 1 1 1
ans =
1 列から 7 列
-1.0000 0.9375 0.9375 2.0000 0.0450 1.0000 1.0000
8 列
0
画像表示
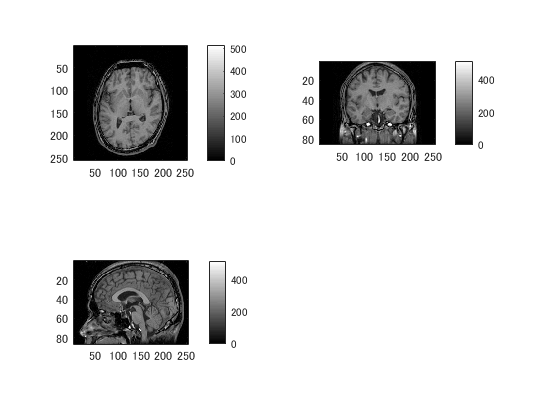
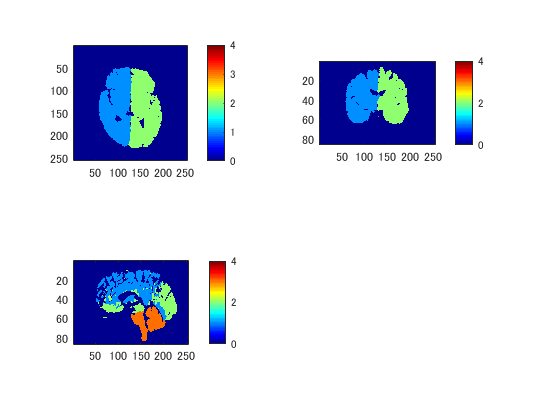
通常のMRIとBrainVISAでlabel付けしたデータとを比較します。 BrainVISAでは左右が反転するようです。
filename2='voronoi_20000406_132343s002a1001.nii.gz'; data2=niftiread([pathname,filename2]); dim=header.raw.dim(2:4);% 最初は〇次元配列の次数 cf fMRI pix=abs(header.raw.pixdim(2:4)); close all; for m=1:2 figure('color',[1,1,1]); switch m case 1 title('通常のMRI'); data=data1; clim=[min(data(:)),max(data(:))*0.5]; case 2 title('ラベル付き'); data=data2; clim=[min(data(:)),max(data(:))]; end for n=1:3 subplot(2,2,n); switch n case 1 d=squeeze(data(:,:,round(dim(3)/2))); p=pix([1,2,3]); case 2 d=squeeze(data(:,round(dim(2)/2),:)); p=pix([1,3,2]); case 3 d=squeeze(data(round(dim(1)/2),:,:)); p=pix([2,3,1]); end imagesc(rot90(d',2)); daspect(1./p); if m==1 % 昔と違ってcolormapはaxes別に指定できるようになってます colormap(gray); else colormap(jet); end set(gca,'clim',clim); colorbar; end end
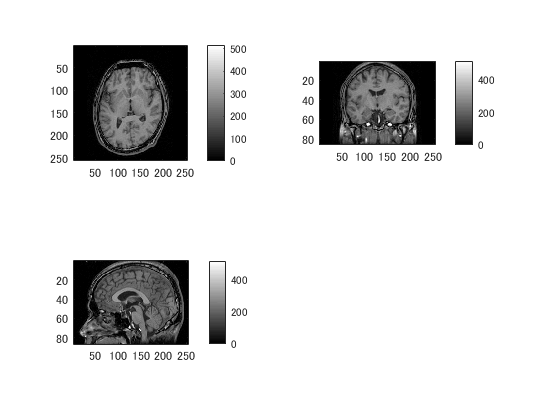
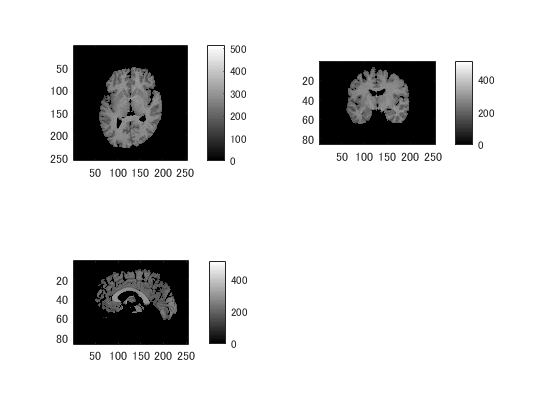
大脳半球だけ抽出してNIfTIファイルで保存
ヘッダーはMRIのものを使います。
close all; data3=0.5*((data2==1)+(data2==2)); data3=int16(data3(end:-1:1,:,:)).*data1; clim=[min(data1(:)),max(data1(:))*0.5]; for m=1:2 figure('color',[1,1,1]); switch m case 1 title('通常のMRI'); data=data1; clim=[min(data(:)),max(data(:))*0.5]; case 2 title('切り出したMRI'); data=data3; end for n=1:3 subplot(2,2,n); switch n case 1 d=squeeze(data(:,:,round(dim(3)/2))); p=pix([1,2,3]); case 2 d=squeeze(data(:,round(dim(2)/2),:)); p=pix([1,3,2]); case 3 d=squeeze(data(round(dim(1)/2),:,:)); p=pix([2,3,1]); end imagesc(rot90(d',2)); daspect(1./p); colormap(gray); set(gca,'clim',clim); colorbar; end end % 拡張子niiやnii.gzは不要です niftiwrite(data3,[pathname,'newbrain'],header,'compressed',true);
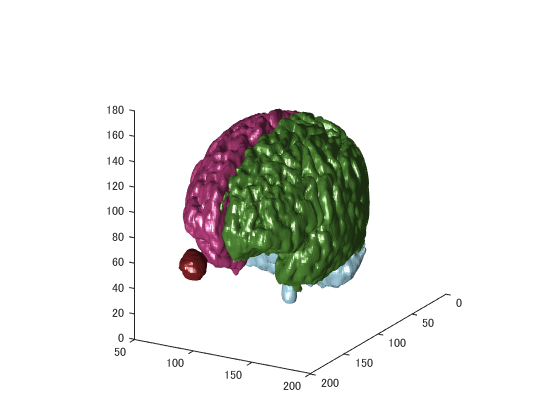
MESH化
MATLABのisosurface関数を使ってメッシュを作成します。 予めsmooth3関数で平滑化した方がいいようです。
close all; addpath('C:\ToBeInstalled\MATLAB\gifti-1.6');% A GIfTI Matlab libraryを組み込む figure('color',[1,1,1]); for n=1:4 v=double(data2==n); v=smooth3(v);% 平滑化 v=isosurface(v,0.7); v.vertices=v.vertices*diag(pix);% ボクセルサイズを考慮 % v=reducepatch(v,20000); % ポリゴン面を少なくするとき h=patch(v,'edgecolor','none','facecolor',rand(1,3),'tag',sprintf('patch%d',n)); if n==1 hold on; end end daspect([1,1,1]); view([120,20]); light('position',[1,0,0]); rotate3d on;
GIfTIファイル化
A MATLAB GIfTI Libraryを使うと簡単にポリゴンメッシュ表示やGIfTIファイルの作成ができます。
close all; addpath('C:\ToBeInstalled\MATLAB\gifti-1.6');% A GIfTI Matlab libraryを組み込む v=isosurface(smooth3(double(data2==1)),0.5); g=gifti(v); figure('color',[1,1,1]); plot(g); save(g,[pathname,'Lhemi.gii'],'Base64Binary');
警告: cameramenu は将来のリリースで削除される予定です。代わりに cameratoolbar を使用してくだ さい。

GIfTIファイルの読み込み
BrainVISAで作成したものは左右が逆になっているようです。 読み込んだ内容を表示させようとしたんですが、できませんでした。 GIfTIの内部構造にversionによる違いがあるのかもしれません。 python側で対応するしかないようです。
addpath('C:\ToBeInstalled\MATLAB\gifti-1.6');% \@gifti'); filename3='20000406_132343s002a1001_Lhemi.gii'; g=gifti([pathname,filename3]); figure('color',[1,1,1]); plot(g);
警告: cameramenu は将来のリリースで削除される予定です。代わりに cameratoolbar を使用してくだ さい。

読み込んだGIfTIファイルの中身表示
gとタイプしただけではエラーになります。
fieldnames(g) size(g.faces) size(g.vertices) % g.matはエラーになる。 g.vertices(:,1)=-g.vertices(:,1);% 左右逆転 % g.mat 変換行列は表示されません。 figure('color',[1,1,1]); plot(g);
ans =
1×3 の cell 配列
{'faces'} {'mat'} {'vertices'}
ans =
113508 3
ans =
56756 3
警告: cameramenu は将来のリリースで削除される予定です。代わりに cameratoolbar を使用してくだ
さい。
