hns_loadfiff5.p
Neuromag独自のFIFFは、MNE-python開発団によって、Windows、Linux、MacOSのpython上で読み書きできるようになりました。 MNE-CやMNE-pythonではFIFFのタグを独自に追加したFIFFファイルが出現しています。 順問題や逆問題の変数にも新たなFIFFのタグがつけられ、 epoch毎切りだした、時間×チャンネル×エポックからなる三次元配列のタグとかもあります。 MNE-pythonは、いろいろな解析手法が開陳されていますが、従来の解析方法はカバーしていません。 そこでまだまだMATLABを使い続ける必要がありそうです。 MNE-pythonのshow_fiff機能を参考に、新たに付加されたFIFFのタグに対応したhns_loadfiff5を作成しました。 FIFFの構造は一般には公開されてないと思いますので、pcodeでコードを隠匿しました。
hns_loadfiff5.pは http://meg.aalip.jp/matlab/data/hns_loadfiff5.p から入手してください。
尚、hns_loadfiff4と出力される構造体に変更があります。 MATLAB R2017bで実行しています。MATLABはR2016から大幅な変更があり、旧いversionだとうまく動作しないかもしれません。
Contents
加算波形表示
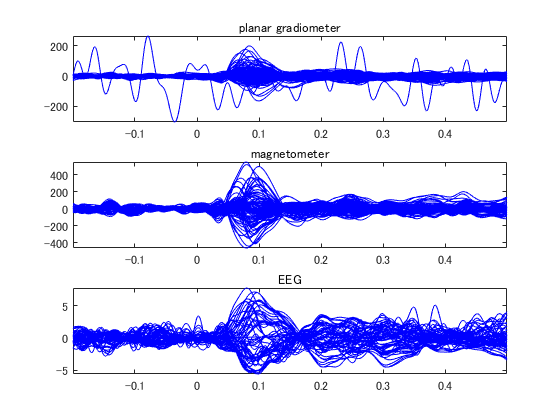
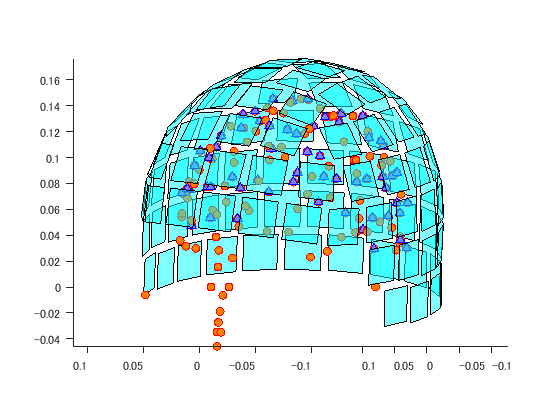
データはMNE-pythonのサンプルデータとします。 mne-sample-data-pathにパスを通しておいてください。 4つの加算波形があります。 pythonだと変数の中身を確認するのが大変ですが、MATLABだと簡単で便利です。 FIFFファイル自体はブロック構造が入れ子入れ子という複雑な構造になっています。 hns_fiff4は利用しやすく変数をまとめているのです。 hns_fiff5でも4との互換性を保つため主な変数は残しています。 また脳波用のSSPがありましたので、PCA_100xとして追加することにしました。 脳波のチャンネル情報の位置情報を表示させていますが、HPIでプロットしたものではなく、合ってるかどうか不明です。
F=hns_loadfiff5('sample_audvis-ave.fif') % ;はナシ % ベースラインの設定 time=F.ave_0001(:,end); t=find(time>=-0.2 & time<0);% baseline baseline=mean(F.ave_0001(t,1:end-1)); data=F.ave_0001(:,1:end-1)-ones(length(time),1)*baseline; % チャンネル名 nch=length(F.ChanName); ch=zeros(1,nch); for n=1:nch if contains(F.ChanName{n},'MEG') if ismember(F.ChanName{n}(end),{'2','3'}) ch(n)=1;% gradiometer else ch(n)=2;% magnetometer end elseif contains(F.ChanName{n},'EEG') ch(n)=3; % EEG end end % SSPの設定 脳波はPCA_100nとしました。 for m=1:2 if m==1 % MEG x=[1,2]; ch0=1; ch1=999; elseif m==2 % EEG x=3; ch0=1001; ch1=1999; end chs=find(ismember(ch,x)); SSP=eye(length(chs)); for n=ch0:ch1 nPCA=sprintf('PCA_%04.0f',n); if isfield(F,nPCA) nPCA=eval(['F.',nPCA]); SSP=SSP-nPCA*nPCA'; else break; end end data(:,chs)=data(:,chs)*SSP; end % 波形表示 figure; for n=1:3 subplot(3,1,n); x=find(ismember(ch,n)); plot(time,data(:,x),'b'); axis tight; switch n case 1;title('planar gradiometer'); case 2;title('magnetometer'); case 3;title('EEG'); end end % 頭皮プロット点とセンサ表示 figure('color',[1,1,1]); plot3(F.DigitPts(:,1),F.DigitPts(:,2),F.DigitPts(:,3),'ro','markerfacecolor',[1,0.5,0]); hold on; p=F.ChInform(:,ch==2); N=size(p,2); X=F.MEG2HEAD; p0=X(:,1:3)*p(1:3,:)+diag(X(:,4))*ones(3,N); px=X(:,1:3)*p(4:6,:)*0.014; py=X(:,1:3)*p(7:9,:)*0.014; pz=X(:,1:3)*p(10:12,:)*0.0003; p0=p0+pz; V=zeros(3,N,4); V(:,:,1)=p0+px+py; V(:,:,2)=p0+px-py; V(:,:,3)=p0-px-py; V(:,:,4)=p0-px+py; h=fill3(squeeze(V(1,:,:))',squeeze(V(2,:,:))',squeeze(V(3,:,:))',ones(4,N)); set(h,'facecolor',[0,1,1],'facealpha',0.5); p0=F.ChInform(:,ch==3); plot3(p0(1,:),p0(2,:),p0(3,:),'b^','markerfacecolor',[0.5,0.5,1]);% HPIでプロットした脳波位置と同一かどうか不明 daspect([1,1,1]); view([-150,0]); axis tight; rotate3d on;
F =
フィールドをもつ struct:
file_id: [1×1 struct]
dir_pointer: 5535745
free_list: -1
meas: [1×1 struct]
nop: []
dir: [677×4 int32]
filename: 'sample_audvis-ave.fif'
ave_0001: [421×377 double]
ave_0002: [421×377 double]
ave_0003: [421×377 double]
ave_0004: [421×377 double]
DigitPts: [146×3 double]
ChInform: [12×376 double]
ChanName: {1×376 cell}
ExamDate: '03-Dec-2002 04:01:10'
MEG2HEAD: [3×8 single]
CalXRnge: [1×377 double]
PCA_0001: [306×1 double]
PCA_0002: [306×1 double]
PCA_0003: [306×1 double]
PCA_1001: [60×1 double]
version: 'Released on 13-Nov-2017↵Since 13-Nov-2017↵by Akira Hashizume'
list: [677×5 int32]
自発波形表示
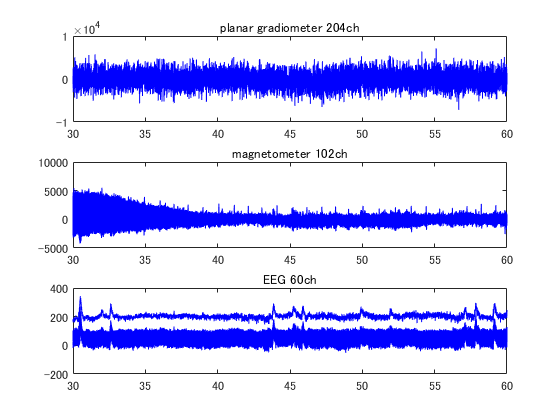
サンプルデータには脳波のSSP情報はないようです。 加算波形とは異なり、CalXRngeとraw_dataをかけて波形を表示させます。
F=hns_loadfiff5('sample_audvis_raw.fif') % ;はナシ % % チャンネル名 nch=length(F.ChanName); ch=zeros(1,nch); for n=1:nch if contains(F.ChanName{n},'MEG') if ismember(F.ChanName{n}(end),{'2','3'}) ch(n)=1;% gradiometer else ch(n)=2;% magnetometer end elseif contains(F.ChanName{n},'EEG') ch(n)=3; % EEG end end % SSP SSP=eye(length(F.PCA_0001)); for n=1:999 if isfield(F,sprintf('PCA_%04.0f',n)) nPCA=eval(sprintf('F.PCA_%04.0f;',n)); SSP=SSP-nPCA*nPCA'; end end % 30~60秒を表示することとする。 sfreq=F.CalXRnge(end); t=round([30,60]*sfreq); t=t(1):t(end); time=t/sfreq; figure; for n=1:3 subplot(3,1,n); chn=find(ch==n); nch=length(chn); data=diag(F.CalXRnge(chn))*double(F.raw_data(chn,t)); plot(time,data,'b'); set(gca,'xlim',[time(1),time(end)]); switch n case 1;title(sprintf('planar gradiometer %dch',nch)); case 2;title(sprintf('magnetometer %dch',nch)); case 3;title(sprintf('EEG %dch',nch)); end end
F =
フィールドをもつ struct:
file_id: [1×1 struct]
dir_pointer: -1
free_list: -1
meas: [1×1 struct]
nop: []
bad_channels: [1×1 struct]
ch_name: 'MEG 2443:EEG 053'
filename: 'sample_audvis_raw.fif'
DigitPts: [146×3 double]
ChInform: [12×376 double]
ChanName: {1×376 cell}
ExamDate: '03-Dec-2002 04:01:10'
MEG2HEAD: [3×8 single]
CalXRnge: [1×377 double]
raw_data: [376×166800 int16]
PCA_0001: [306×1 double]
PCA_0002: [306×1 double]
PCA_0003: [306×1 double]
version: 'Released on 13-Nov-2017↵Since 13-Nov-2017↵by Akira Hashizume'
list: [918×5 int32]
エポックファイル
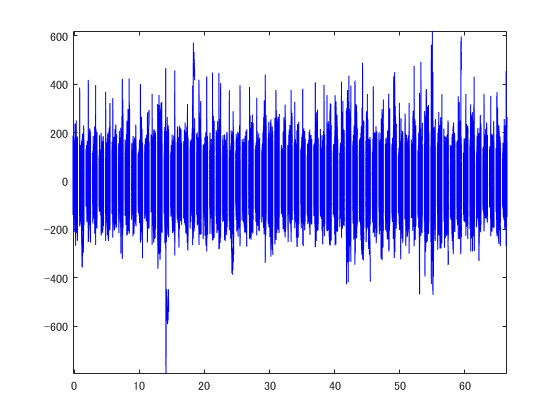
F.EpocDatahは時間×チャンネル×試行からなる三次元配列です。 planar gradiometerでエポックを切り出し、aaa-epo.fifで保存したファイルを読み込みんでみます。 planar gradiometerだけですのでSSPはありません。 stack波形を試行順にx軸方向に並べました。
F=hns_loadfiff5('aaa-epo.fif') % ;はナシ figure; t=F.EpocTime;%/F.meas.meas_info.sfreq; for n=1:size(F.EpocData,3) xx=squeeze(F.EpocData(:,:,n)); plot(t+(n-1),xx,'b'); if n==1;hold on;end end axis tight;
F =
フィールドをもつ struct:
file_id: [1×1 struct]
dir_pointer: -1
free_list: -1
meas: [1×1 struct]
nop: []
filename: 'aaa-epo.fif'
EpocTime: [106×1 double]
EpocData: [106×204×67 double]
DigitPts: [146×3 double]
ChInform: [12×204 double]
ChanName: {1×204 cell}
ExamDate: '03-Dec-2002 04:01:10'
MEG2HEAD: [3×8 single]
CalXRnge: [1×205 double]
version: 'Released on 13-Nov-2017↵Since 13-Nov-2017↵by Akira Hashizume'
list: [471×5 int32]
空間フィルタファイル
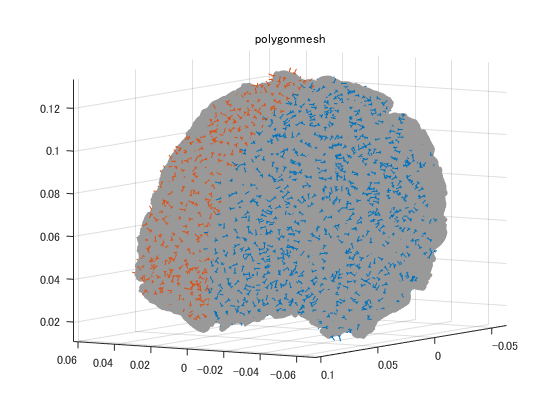
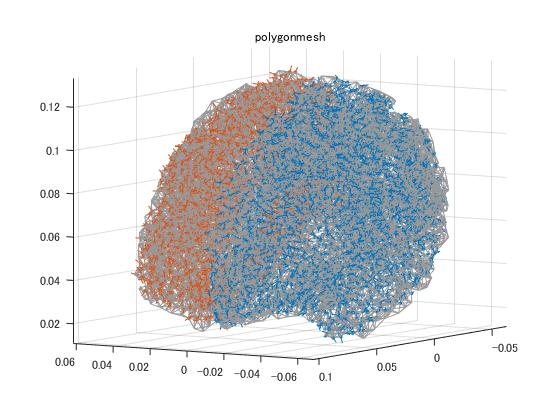
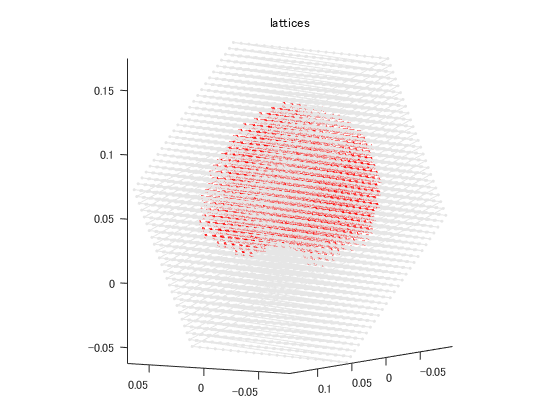
MNE側で独自に拡張したFIFFファイルでxxx-inv.fifという名前になっています。 まだ詳細不明な項目が残ったままになっています。 電流源の置き方に、脳表三角メッシュの頂点に置く場合と等間隔で脳を分割した格子点とがあります。 計算量を減らすため、電流源を間引いて電流源推定しています。 脳表メッシュでは 左半球の△310810を△8192にし、電流源を155407点中3732点に減らし、 右半球の△313728を△8192にし、電流源を156866点中3766点に減らしています。 格子点では電流源を13104点中3757点に減らしています。
% 脳表メッシュ filename='sample_audvis-meg-eeg-oct-6-meg-eeg-inv.fif'; F=hns_loadfiff5(filename)% ;はナシ disp('F.mne'); F.mne disp('F.mne.mne_source_space{1} % left hemisphere'); F.mne.mne_source_space{1} disp('F.mne.mne_source_space{2} % right hemisphere'); F.mne.mne_source_space{2} for m=1:2 figure; for n=1:2 S=F.mne.mne_source_space{n}; p=double(S.source_rr);% メッシュ頂点 if m==1 h=trimesh(S.tris',p(1,:),p(2,:),p(3,:)); else h=trimesh(S.use_tris',p(1,:),p(2,:),p(3,:)); end set(h,'edgecolor',[1,1,1]*0.6,'facecolor','none','facealpha',0.5); hold on; q=double(S.source_nn); % 方向 k=find(S.inuse==1);% 使用された格子点 quiver3(p(1,k),p(2,k),p(3,k),q(1,k),q(2,k),q(3,k),'o','markersize',2); end daspect([1,1,1]); axis tight; view([-148,6]); rotate3d on; title('polygonmesh'); end % 格子点 F=hns_loadfiff5('sample_audvis-meg-vol-7-meg-inv.fif') % ;はナシ disp('F.mne'); F.mne disp('F.mne.mne_source_space'); % lattices F.mne.mne_source_space figure; S=F.mne.mne_source_space; p=double(S.source_rr);% 格子点 q=double(S.source_nn);% 方向 k=find(S.inuse==1);% 使用された格子点 plot3(p(1,:),p(2,:),p(3,:),'color',[1,1,1]*0.9,'marker','.'); hold on; quiver3(p(1,k),p(2,k),p(3,k),q(1,k),q(2,k),q(3,k),'ro','markersize',2);% 13104点中3757点使用 daspect([1,1,1]); axis tight; view([-148,6]); rotate3d on; title('lattices');
F =
フィールドをもつ struct:
file_id: [1×1 struct]
dir_pointer: -1
free_list: -1
mne: [1×1 struct]
nop: []
mne_env: [1×1 struct]
filename: 'sample_audvis-meg-eeg-oct-6-meg-eeg-inv.fif'
version: 'Released on 13-Nov-2017↵Since 13-Nov-2017↵by Akira Hashizume'
list: [560×5 int32]
F.mne
ans =
フィールドをもつ struct:
mne_meg_file: [1×1 struct]
mne_trans_coord_meg2mri: [1×1 struct]
xfit_proj: [1×1 struct]
mne_source_space: {[1×1 struct] [1×1 struct]}
mne_inv: [1×1 struct]
F.mne.mne_source_space{1} % left hemisphere
ans =
フィールドをもつ struct:
space_type: 1
space_id: 101
subj_his_id: 'sample'
coord_frame: 4
n_vertices: 155407
source_rr: [3×155407 single]
source_nn: [3×155407 single]
inuse: [1×155407 int32]
n_used: 3732
nrti: 310810
tris: [3×310810 int32]
nuse_tri: 8192
use_tris: [3×8192 int32]
nearest_array: [1×155407 int32]
nearest_dist: [1×155407 single]
source_potential_sol: [155407×155407 double]
dist_limit: -829934272
F.mne.mne_source_space{2} % right hemisphere
ans =
フィールドをもつ struct:
space_type: 1
space_id: 102
subj_his_id: 'sample'
coord_frame: 4
n_vertices: 156866
source_rr: [3×156866 single]
source_nn: [3×156866 single]
inuse: [1×156866 int32]
n_used: 3766
nrti: 313728
tris: [3×313728 int32]
nuse_tri: 8192
use_tris: [3×8192 int32]
nearest_array: [1×156866 int32]
nearest_dist: [1×156866 single]
source_potential_sol: [156866×156866 double]
dist_limit: -829934272
F =
フィールドをもつ struct:
file_id: [1×1 struct]
dir_pointer: -1
free_list: -1
mne: [1×1 struct]
nop: []
mne_env: [1×1 struct]
filename: 'sample_audvis-meg-vol-7-meg-inv.fif'
version: 'Released on 13-Nov-2017↵Since 13-Nov-2017↵by Akira Hashizume'
list: [487×5 int32]
F.mne
ans =
フィールドをもつ struct:
mne_meg_file: [1×1 struct]
mne_trans_coord_meg2mri: {[1×1 struct] [1×1 struct]}
xfit_proj: [1×1 struct]
mne_source_space: [1×1 struct]
mne_inv: [1×1 struct]
F.mne.mne_source_space
ans =
フィールドをもつ struct:
space_type: 2
coord_frame: 4
n_vertices: 13104
source_rr: [3×13104 single]
source_nn: [3×13104 single]
inuse: [1×13104 int32]
n_used: 3757
source_space_nneighbours: [1×13104 int32]
source_space_neighbours: [1×340704 int32]
coord_trans: [1×1 struct]
source_space_voxel_dims: [21 26 24]
mne_trans_coord_meg2mri: [1×1 struct]
STCファイル
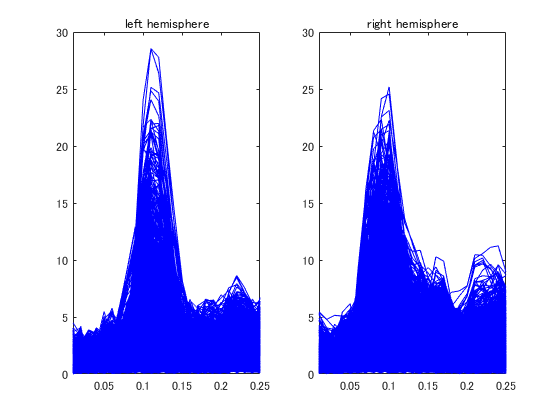
STCファイルはsources time courseの略です。電流源の経時変化情報が記載されています。 電流源の位置は記載されていません。
for n=1:2 if n==1 filename='sample_audvis-meg-eeg-lh.stc';% 左半球 figure; st='left hemisphere'; else filename='sample_audvis-meg-eeg-rh.stc';% 右半球 st='right hemisphere'; end F=hns_loadfiff5(filename) subplot(1,2,n); time=(1:size(F.data,2))'*F.tstep; plot(time,F.data,'b'); set(gca,'xlim',[time(1),time(end)],'ylim',[0,30]); title(st); end
F =
フィールドをもつ struct:
tmin: 0
tstep: 0.0100
vertices: [3732×1 uint32]
data: [3732×25 single]
F =
フィールドをもつ struct:
tmin: 0
tstep: 0.0100
vertices: [3766×1 uint32]
data: [3766×25 single]
Wファイル
WファイルはSTCファイルのある一時点の電流源の番号と電流の大きさを示すデータです。 要するにSTCファイルのverticesとdataの1行分のデータです。 どの時間のデータか?の情報はありません。
filename='sample_audvis-meg-oct-6-fwd-sensmap-lh.w';
F=hns_loadfiff5(filename)
F =
フィールドをもつ struct:
vertices: [3732×1 uint32]
data: [3732×1 single]
labelファイル
これ、実はテキストファイルです。
clear F; filename='lh.cortex.label'; F=hns_loadfiff5(filename)
F =
フィールドをもつ struct:
comment: '#!ascii label , from subject vox2ras=TkReg ↵'
vertices: [147147×1 int32]
pos: [147147×3 double]
values: [147147×1 double]