Pコード化
関数pcodeを使うとMatlabのスクリプト/関数Mファイルを隠蔽化することができます。 Fiffファイルの構造は、一般には公開されていないので、FiffファイルをMatlabで読み込む関数を pcode化することとしました。
hns_loadfiff4.mというファイルをpcode(hns_loadfiff4.m)としてhns_loadfiff4.pというファイルを作成しました。 ファイルは http://meg.aalip.jp/matlab/data/hns_loadfiff4.p から入手してください。
Fiff2MatFileはMatlab Compiler Runtime(MCR)が必要で、いろいろ煩雑なのですが、 hns_loadfiff4.pだと、Matlab上で直接データの読み込みができるので便利だと思います。
Contents
raw_dataで保存した自発波形(てんかん)のFIFFファイルを読む
hns_loadiff4はFiff2MatFileの元になったファイルです。 Fiff2MatFileではMEX化することで高速化していますが、 hns_loadfiff4.pは高速化してないので、処理速度が遅くなっています。 結果はFの構造体としてFiff2MatFileとほぼ同じものが出力されています。
meg='epilepsy1.fif';
tic;
F=hns_loadfiff4(meg)
toc;
F =
ID: [1x1 struct]
file: 'epilepsy1.fif'
size: 245404646
list: [5x1357 int32]
nlist: 1357
ExamDate: '17-Dec-2012 13:34:22'
subject: [1x1 struct]
Examinee: 'case 1328'
KindCoil: [2x336 double]
CalXRnge: [1x337 double]
ChInform: [12x336 double]
ChanName: {1x336 cell}
SSPChanl: {1x306 cell}
PCA_0001: [306x1 double]
PCA_0002: [306x1 double]
PCA_0003: [306x1 double]
PCA_0004: [306x1 double]
PCA_0005: [306x1 double]
PCA_0006: [306x1 double]
PCA_0007: [306x1 double]
PCA_0008: [306x1 double]
PCA_0009: [306x1 double]
PCA_0010: [306x1 double]
bandpass: [0.1000 172.1763]
DigitPts: [247x3 double]
MEG2HEAD: [3x8 single]
sfreq: 600.6150
raw_data: [336x360600 int16]
経過時間は74.927801秒です
TSSSで32bit浮動小数点でencodeした自発波形(てんかん)のFIFFファイルを読む
TSSS処理に伴い、いろいろな変数がでています。 使い方は・・・よくわかりません。
meg='epilepsy1_tsss_float.fif';
tic;
G=hns_loadfiff4(meg)
toc;
G =
ID: [1x1 struct]
file: 'epilepsy1_tsss_float.fif'
size: 487822517
list: [5x1308 int32]
nlist: 1308
ExamDate: '17-Dec-2012 13:34:22'
subject: [1x1 struct]
Examinee: 'case 1328'
KindCoil: [2x336 double]
CalXRnge: [1x337 double]
ChInform: [12x336 double]
ChanName: {1x336 cell}
SSPChanl: {1x306 cell}
bandpass: [0.1000 172.1763]
DigitPts: [247x3 double]
MEG2HEAD: [3x8 single]
sfreq: 600.6150
CrosTalk: [306x306 double]
CTMagSns: [2x306 double]
CTChInfo: [14x306 double]
raw_data: [336x360600 single]
経過時間は131.281215秒です
Magnes 2500 WHの体性感覚誘発磁場のFIFFファイルを読む
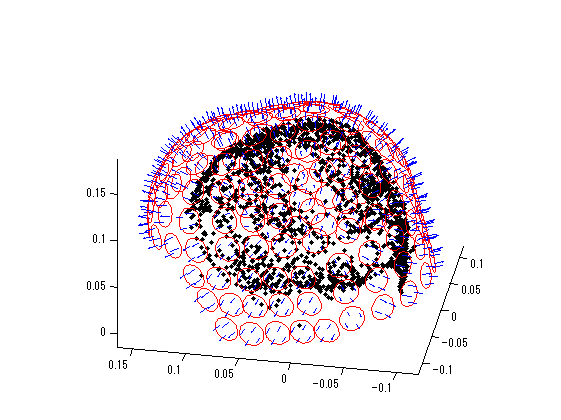
Magnes 2500 WHの体性感覚誘発磁場のデータを岡山理科大学の畑中啓作先生の好意でいただきました。 148chのmagnetometer、11chのreference coilで構成されますが、reference coilの情報はFIFFに含まれていていません。 コイル半径はあてずっぽうで12mmとしました。赤丸がコイル、 青線は磁場計算用のコイル中心から(±5.75,±5.75,0)mmの点の法線成分、 黒の点はPolhemusで記録した頭皮の点です。
clear; meg='MagnesSEF.fif'; % meg='fujii_teruaki_1-2ym-m.fif'; tic; F=hns_loadfiff4(meg) toc
F =
ID: [1x1 struct]
file: 'MagnesSEF.fif'
size: 650561
list: [5x1930 int32]
nlist: 1930
subject: [1x1 struct]
Examinee: 'Hiroki Nakama'
KindCoil: [2x148 double]
CalXRnge: [2x148 double]
ChInform: [12x148 double]
ChanName: {1x148 cell}
bandpass: [0 813.8040]
DigitPts: [1602x3 double]
MEG2HEAD: [3x8 single]
sfreq: 2.0345e+003
ave_0001: [916x149 double]
経過時間は0.694556秒です
Magnes 2500 WHの体性感覚誘発磁場のFIFFファイルを表示
figure('color',[1,1,1]); M=F.MEG2HEAD(1:3,1:4); megch=find(F.KindCoil(1,:)==1); P=M(:,1:3)*F.ChInform(1:3,megch)+M(:,4)*ones(1,length(megch)); X=M(:,1:3)*F.ChInform(4:6,megch); Y=M(:,1:3)*F.ChInform(7:9,megch); Z=M(:,1:3)*F.ChInform(10:12,megch); r=0.012;% コイル半径を12mmとする。 seg=37; t=linspace(0,2*pi,seg); x=r*cos(t); y=r*sin(t); weight=5.75*1e-3; for sns=1:length(megch) coil=P(:,sns)*ones(1,seg)+X(:,sns)*x+Y(:,sns)*y; plot3(coil(1,:),coil(2,:),coil(3,:),'r-'); if sns==1;hold on;end; meas=P(:,sns)*ones(1,4)+weight*X(:,sns)*[1,1,-1,-1]+weight*Y(:,sns)*[1,-1,1,-1]; z=Z(:,sns)*ones(1,4); quiver3(meas(1,:),meas(2,:),meas(3,:),z(1,:),z(2,:),z(3,:),'b-'); end; D=F.DigitPts; plot3(D(:,1),D(:,2),D(:,3),'k.'); view([-80,30]); daspect([1,1,1]);axis tight;rotate3d on; megch=find(F.KindCoil(1,:)==1); P=F.ChInform(1:3,megch)'; [azimuth,elevation]=cart2sph(P(:,1),P(:,2),P(:,3)); k=1; elevation=(1-elevation/pi*2).^k; ChPos(:,1)=cos(azimuth).*elevation; ChPos(:,2)=sin(azimuth).*elevation; smp=size(F.ave_0001,1); time=F.ave_0001(:,end); span=find(time<-5);% 5msより前をbaselineとする MEG=F.ave_0001(:,megch); MEG=MEG-ones(smp,1)*mean(MEG(span,:),1);% baseline補正 xscale=5e-2; yscale=5e-5; X=ChPos(:,1)*ones(1,smp)+xscale*ones(size(ChPos,1),1)*linspace(-1,1,length(time)); Y=ChPos(:,2)*ones(1,smp)+yscale*MEG'; figure('color',[1,1,1]); plot(X',Y','color',[0,0,1]); axis tight;axis off; t=find(time>19,1);% 19msec以上の最初のindex seg=200; xx=linspace(min(ChPos(:,1)),max(ChPos(:,1)),seg); yy=linspace(min(ChPos(:,2)),max(ChPos(:,2)),seg); [xx,yy]=meshgrid(xx,yy); zz=griddata(ChPos(:,1),ChPos(:,2),MEG(t,:)',xx,yy); figure('color',[1,1,1]); step=200; contour(xx,yy,zz,'LevelList',(fix(min(zz(:))/step)*step):step:-step,'color',[0,0,1]);hold on; contour(xx,yy,zz,'LevelList',0,'color',[1,1,1]*0.1); contour(xx,yy,zz,'LevelList',step:step:(fix(max(zz(:))/step)*step),'color',[1,0,0]); plot(ChPos(:,1),ChPos(:,2),'k.'); for sns=1:size(ChPos,1) str=F.ChanName(megch(sns)); str=strrep(str,'MEG ',''); text(ChPos(sns,1),ChPos(sns,2),str); end; axis tight; axis off;

横河の脳磁図データをMatlabで読むPコード
実はPコードの存在を知ったのは、横河の脳磁図データをMatlabで読むためのツールがあるのに気づいてからでした。 で、横河のPコード、横河電機のホームページに登録すると、software downloadのところから入手できるそうです。 宮崎県都城市の藤元総合病院の大坪俊昭先生のご好意で頂いたSEFのデータを使ってみます。 aveは加算データ、rawは元データですが、 conという拡張子のデータがあります。rawより時間が短い元データのようですが、よくわかりません。
clear F; meg='SEF左1_n402.ave'; mrk='SEFマーカ左1.mrk'; mri='subject_mri_volume.mri'; F.data =getYkgwData(meg); % 計測データ F.system =getYkgwHdrSystem(meg);% 横河の脳磁計や脳磁図のデータファイルの情報 F.channel =getYkgwHdrChannel(meg);% チャンネル情報 F.acqcond =getYkgwHdrAcqCond(meg);% 計測条件など F.event =getYkgwHdrEvent(meg);% 加算回数やトリガーチャンネルなど F.coregist=getYkgwHdrCoregist(mrk);% 頭皮上のマーカー座標を抽出 F.digitize=getYkgwHdrDigitize(meg); F.subject =getYkgwHdrSubject(meg);% 患者情報など F.bookmark=getYkgwHdrSource(meg); F.MRI =getYkgwMriHdr(mri);% MRIのヘッダー情報の抽出 F.version =getYkgwVersion(meg); F F.acqcond % 計測条件 F.channel.channel(1).data % ch1の情報 clear G; con='SEF左1.con'; G.data =getYkgwData(con); G.acqcond=getYkgwHdrAcqCond(con); G.channel=getYkgwHdrChannel(con); G.event=getYkgwHdrEvent(con); G G.acqcond G.channel.channel(1).data clear H; raw='SEF左1のエポック.raw'; tic; H.data=getYkgwData(raw); toc; H.acqcond=getYkgwHdrAcqCond(raw); H.channel=getYkgwHdrChannel(raw); H.event=getYkgwHdrEvent(raw); H H.acqcond H.channel.channel(1).data
F =
data: [224x2000 double]
system: [1x1 struct]
channel: [1x1 struct]
acqcond: [1x1 struct]
event: [1x1 struct]
coregist: [1x1 struct]
digitize: [1x1 struct]
subject: [1x1 struct]
bookmark: []
MRI: [1x1 struct]
version: [1x1 struct]
ans =
acq_type: 2
sample_rate: 2000
frame_length: 2000
pretrigger_length: 1000
average_count: 402
specified_average_count: 450
multi_trigger: [1x1 struct]
ans =
x: 0.1162
y: 0.0225
z: 0.0433
zdir: 70.9840
xdir: 15.3399
baseline: 0.0500
size: 0.0155
name: ''
G =
data: [224x820000 double]
acqcond: [1x1 struct]
channel: [1x1 struct]
event: []
ans =
acq_type: 1
sample_rate: 2000
sample_count: 820000
specified_sample_count: 820000
ans =
x: 0.1162
y: 0.0225
z: 0.0433
zdir: 70.9840
xdir: 15.3399
baseline: 0.0500
size: 0.0155
name: ''
経過時間は20.017364秒です
H =
data: [224x804000 double]
acqcond: [1x1 struct]
channel: [1x1 struct]
event: [1x402 struct]
ans =
acq_type: 3
sample_rate: 2000
frame_length: 2000
pretrigger_length: 1000
average_count: 402
specified_average_count: 450
multi_trigger: [1x1 struct]
ans =
x: 0.1162
y: 0.0225
z: 0.0433
zdir: 70.9840
xdir: 15.3399
baseline: 0.0500
size: 0.0155
name: ''
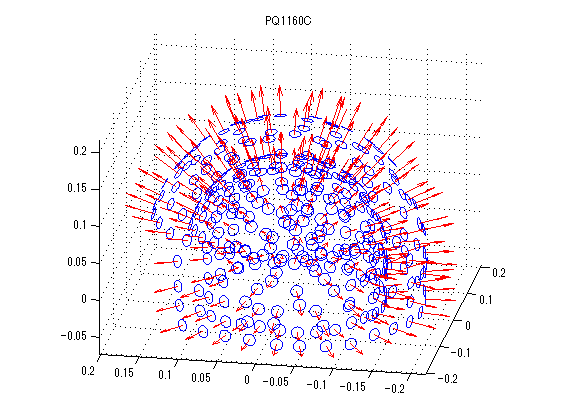
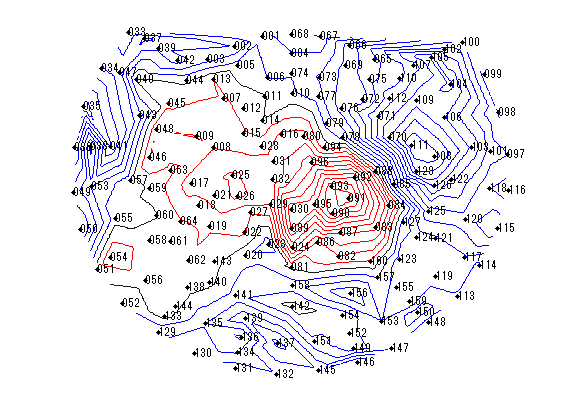
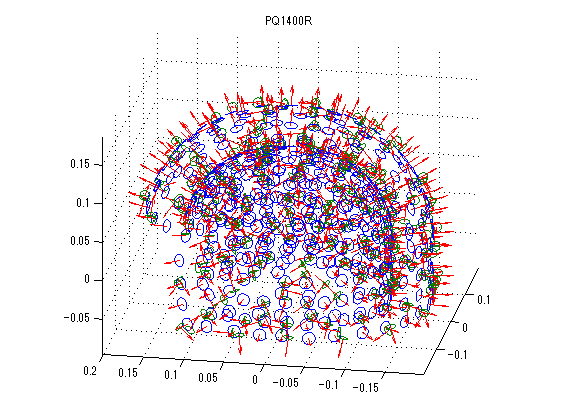
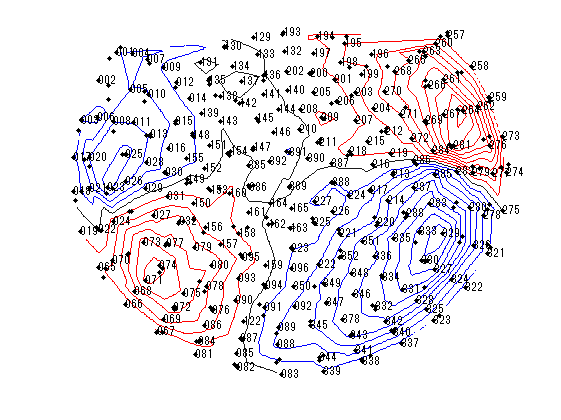
横河電機社製脳磁計 PQ1160Cの体性感覚誘発磁場とPQ1400Rの聴覚誘発磁場ファイルを表示
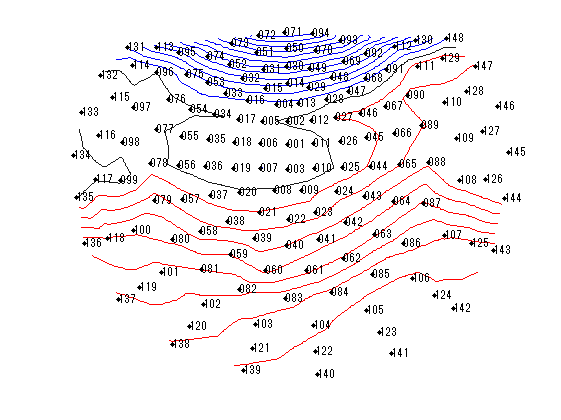
変数をなるべくhns_loadfiffのものになるように書き換えています。 横河電機には脳磁図番号がないみたいなので適当につけてます。 等磁場線図はaxial gradiometerの成分のみを用いています。
for nmeg=1:2 clear F; switch nmeg case 1;meg='SEF左1_n402.ave'; case 2;meg='A008a_BC_0.5HPF_100LPF_trig437_label4.ave'; end; F.channel =getYkgwHdrChannel(meg); F.system =getYkgwHdrSystem(meg); F.ave_0001=getYkgwData(meg)';% 行・列をMatlab仕様に F.acqcond =getYkgwHdrAcqCond(meg); sfreq=F.acqcond.sample_rate; time=((1:size(F.ave_0001,1))-F.acqcond.pretrigger_length)/sfreq*1000;% msec F.ave_0001(:,end+1)=time(:); nch=F.channel.channel_count; F.KindCoil=zeros(1,nch); F.ChInform=zeros(9,nch); str=F.system.model_name; megno=1; nullno=1; refno=1; stimno=1; eegno=1; for n=1:nch sns=F.channel.channel(n).type; F.KindCoil(n)=sns; ch=F.channel.channel(n).data; if ismember(sns,[1,2,3])% 1 magnetometer 2 axial 3 planar gradiometer F.ChInform([1:3,9],n)=[ch.x,ch.y,ch.z,ch.size]; F.ChanName{n}=sprintf('MEG %03.0f',megno);megno=megno+1; end; switch sns case 2; F.ChInform(4:8,n)=[ch.zdir,ch.xdir,ch.zdir,ch.xdir,ch.baseline]; case 3; F.ChInform(4:8,n)=[ch.zdir1,ch.xdir1,ch.zdir2,ch.xdir2,ch.baseline]; case 0; F.ChanName{n}=sprintf('NULL %03.0f',nullno);nullno=nullno+1; case {257,258,259} F.ChanName{n}=sprintf('REF %03.0f',refno);refno=refno+1; case -1; F.ChanName{n}=sprintf('STI %03.0f',stimno);stimno=stimno+1; case {-2,-3,-4} name=F.channel.channel(n).data.name; F.ChanName{n}=[sprintf('EEG %03.0f ',eegno),name]; eegno=eegno+1; end; end; megch=find(F.KindCoil>0 & F.KindCoil<4); axch=find(F.KindCoil(megch)==2); plch=find(F.KindCoil(megch)==3); P0=F.ChInform(1:3,megch); phi=F.ChInform(6,megch)/180*pi; theta=F.ChInform(7,megch)/180*pi; Z=[cos(theta).*sin(phi);sin(theta).*sin(phi);cos(phi)]; P1=P0+(ones(3,1)*F.ChInform(8,megch)).*Z;% baseline phi=F.ChInform(4,megch)/180*pi; theta=F.ChInform(5,megch)/180*pi; Z=[cos(theta).*sin(phi);sin(theta).*sin(phi);cos(phi)]; X=[cos(theta).*cos(phi);sin(theta).*cos(phi);-sin(phi)]; Y=[-sin(theta);cos(theta);0*phi]; % Neuromagと同じ座標系にする P0(1:2,:)=[-P0(2,:);P0(1,:)]; P1(1:2,:)=[-P1(2,:);P1(1,:)]; Z(1:2,:)=[-Z(2,:);Z(1,:)]; X(1:2,:)=[-X(2,:);X(1,:)]; Y(1:2,:)=[-Y(2,:);Y(1,:)]; seg=37; t=linspace(0,2*pi,seg); figure('color',[1,1,1]); quiver3(P0(1,:),P0(2,:),P0(3,:),Z(1,:),Z(2,:),Z(3,:),'r-');hold on; quiver3(P1(1,:),P1(2,:),P1(3,:),Z(1,:),Z(2,:),Z(3,:),'r-'); for sns=1:length(megch) r=F.ChInform(9,megch(sns))/2;% コイルの内径/2 x=r*cos(t); y=r*sin(t); if F.KindCoil(megch(sns))==2;color=[0,0,1];else color=[0,0.5,0];end; for nn=0:1 switch nn case 0;P=P0; case 1;P=P1; end; coil=P(:,sns)*ones(1,seg)+X(:,sns)*x+Y(:,sns)*y; plot3(coil(1,:),coil(2,:),coil(3,:),'color',color); end; end; axis tight;daspect([1,1,1]);view([-80,20]);rotate3d on; title(str); [azimuth,elevation]=cart2sph(P1(1,:),P1(2,:),P1(3,:));% 少し離れてる方がch分離しやすい k=1; elevation=(1-elevation/pi*2).^k; clear ChPos; ChPos(:,1)=cos(azimuth).*elevation; ChPos(:,2)=sin(azimuth).*elevation; smp=size(F.ave_0001,1); time=F.ave_0001(:,end); span=find(time<-5);% 5msより前をbaselineとする MEG=F.ave_0001(:,megch)*1e+15;% fTに MEG=MEG-ones(smp,1)*mean(MEG(span,:),1);% baseline補正 xscale=5e-2; yscale=2e-4; X=ChPos(:,1)*ones(1,smp)+xscale*ones(size(ChPos,1),1)*linspace(-1,1,length(time)); Y=ChPos(:,2)*ones(1,smp)+yscale*MEG'; figure('color',[1,1,1]); plot(X(axch,:)',Y(axch,:)','color',[0,0,1]);hold on; if ~isempty(plch) plot(X(plch,:)',Y(plch,:)','color',[0,0.5,0]); end; axis tight;axis off; title(str); if nmeg==1 t=find(time>18,1);% 19msec以上の最初のindex else t=find(time>100,1);% 100msec以上の最初のindex end; seg=200; xx=linspace(min(ChPos(axch,1)),max(ChPos(axch,1)),seg); yy=linspace(min(ChPos(axch,2)),max(ChPos(axch,2)),seg); [xx,yy]=meshgrid(xx,yy); zz=griddata(ChPos(axch,1),ChPos(axch,2),MEG(t,axch)',xx,yy); figure('color',[1,1,1]); if nmeg==1;step=10;else step=25;end; contour(xx,yy,zz,'LevelList',(fix(min(zz(:))/step)*step):step:-step,'color',[0,0,1]);hold on; contour(xx,yy,zz,'LevelList',0,'color',[1,1,1]*0.1); contour(xx,yy,zz,'LevelList',step:step:(fix(max(zz(:))/step)*step),'color',[1,0,0]); plot(ChPos(:,1),ChPos(:,2),'k.'); for sns=1:size(ChPos(axch,:),1) str=F.ChanName(megch(axch(sns))); str=strrep(str,'MEG ',''); text(ChPos(axch(sns),1),ChPos(axch(sns),2),str); end; axis tight; axis off; % title([str,sprintf(' %d fT/step',step)]); end;