12. dipole推定
FreeSurferの構造画像のデータを読み込んで、脳磁図データも読み込み、位置合わせが終わったとします。
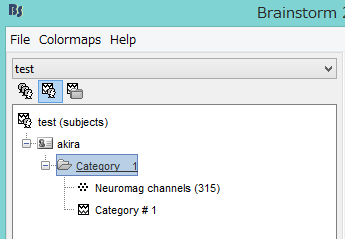
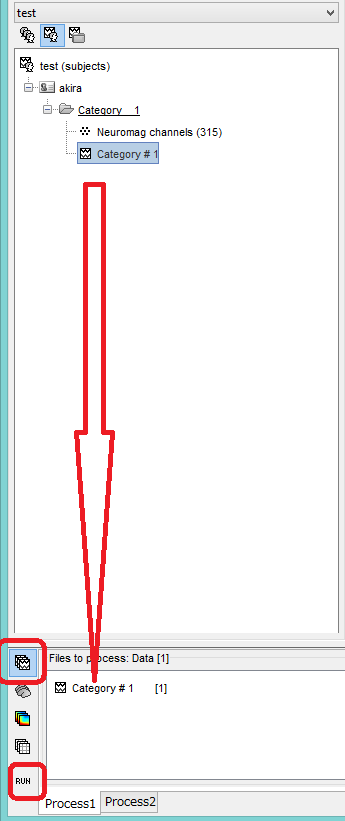
脳磁図データを下の欄にドラッグし、ファイルマークをクリックしたのちRUNをクリックします。
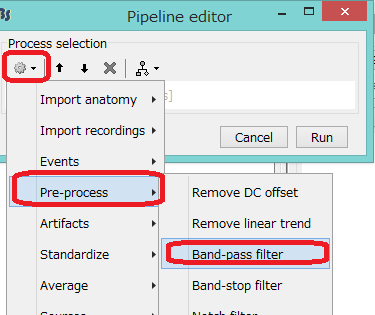
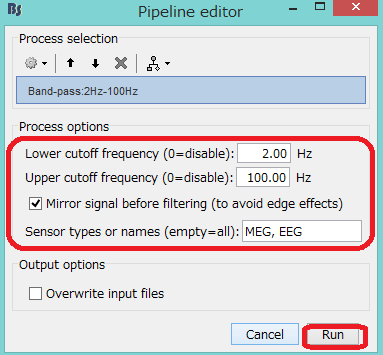
歯車をクリックし、pre-processをクリックして、band-pass
filterを選択します。
周波数フィルタをかけて、RUNをクリックします。
フィルタ処理後のファイルを右クリックし、Compute head modelを選択します。
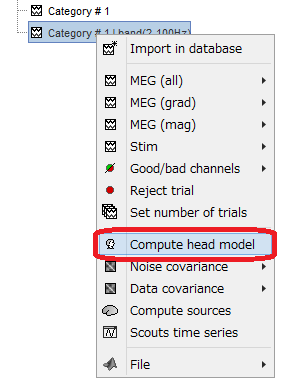
dipoleの場所は皮質限定Cortex surfaceでもいいのですが、MRI
volumeを選択しOKを押します。
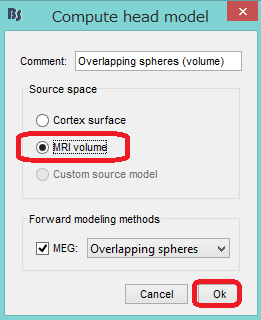
MRI volumeといっても、皮質面から深部に17層のメッシュを想定し、約20606個のgrid点の中から単一双極子推定法で最適の双極子を選ぶというアルゴリズムです。Previewを押すと、grid点が表示されます。OKを押します。
Overlapping spheres (volume)が作成されました。
フィルタ処理後の波形を右クリックしてNoise covarianceを選択し、Compute from recordingsを選択します。
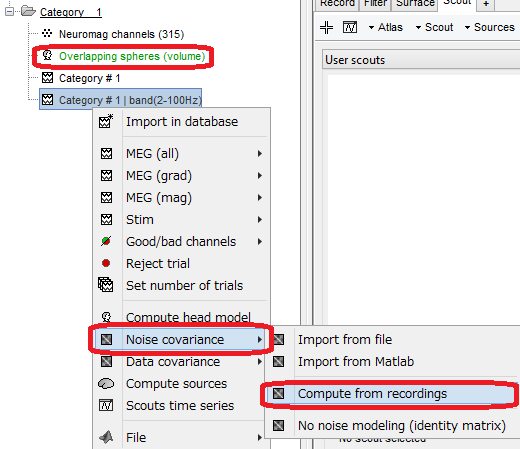
Baselineを設定し、OKを押します。
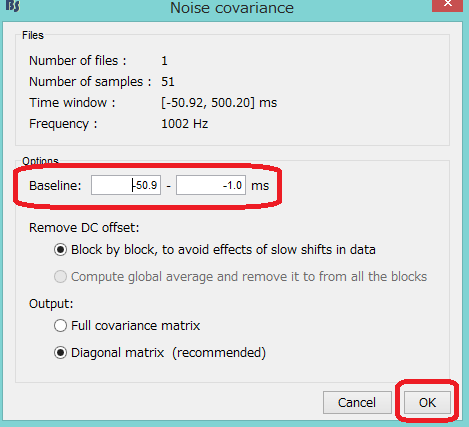
Noise covarianceが作成されました。
フィルタ処理後の波形を右クリックし、Compute sourcesをクリックします。
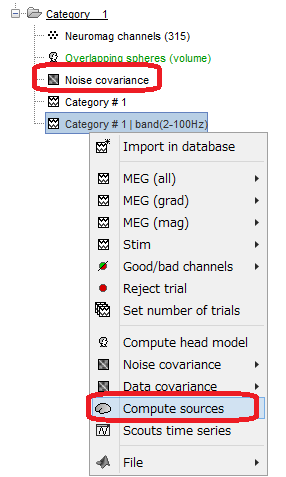
dSPMを選択し、OKを押します。
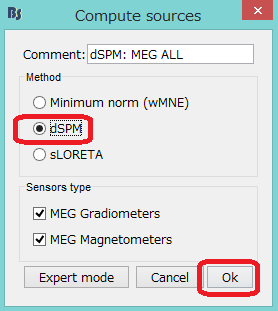
dSPM: MEG ALLを下の欄にドラッグし、RUNをクリックします。
d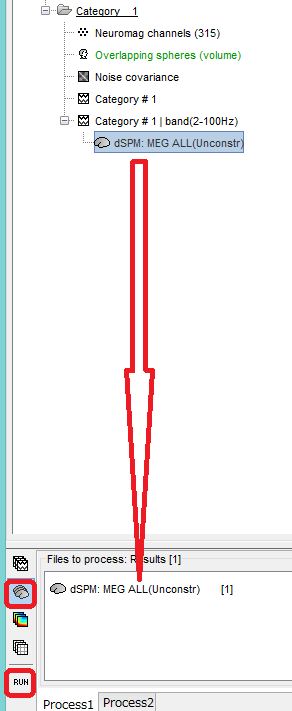
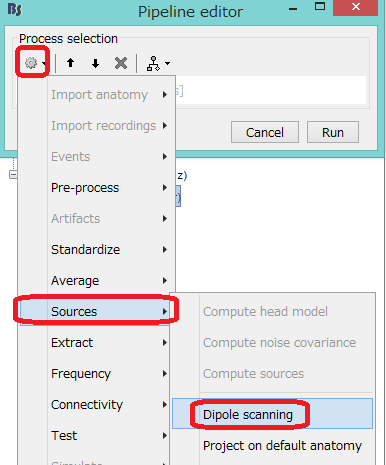
歯車をクリックし、Sourcesを選択し、Dipole
scanningを選択します。
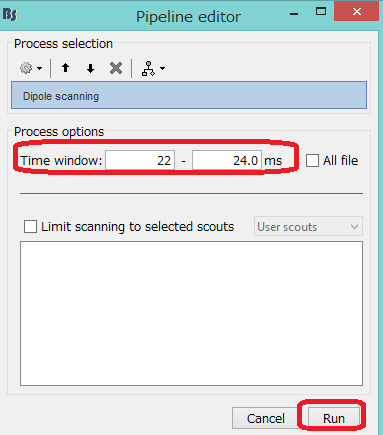
時間を設定し、Runを押します。
Dipoleのマークができます。Display on cortexやDisplay
on MRI(3D)を選択します。
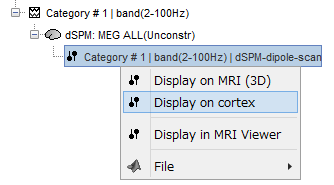
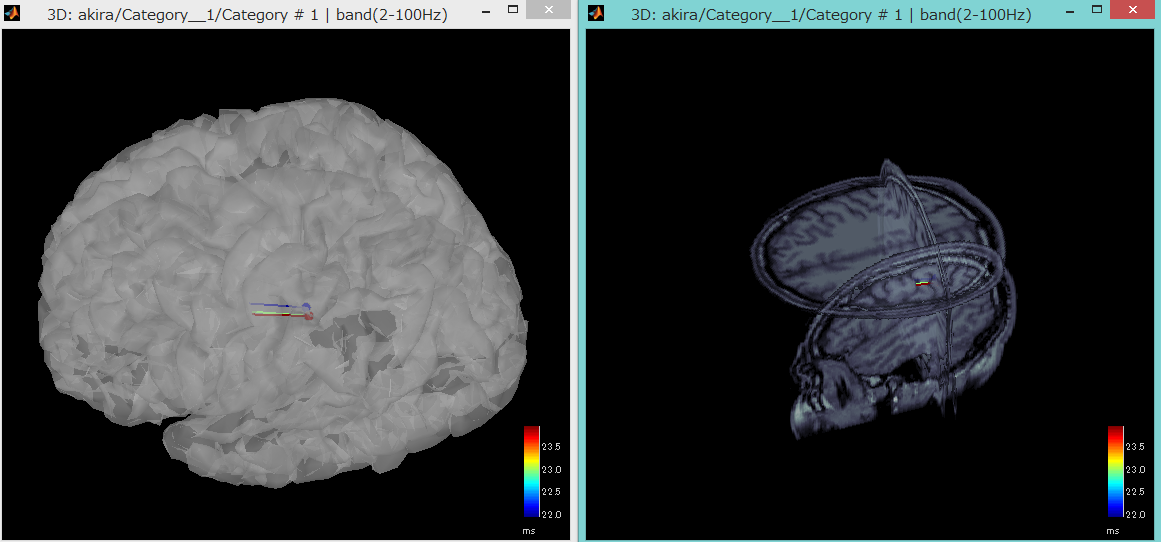
Display in MRI viewerを選択してみました。
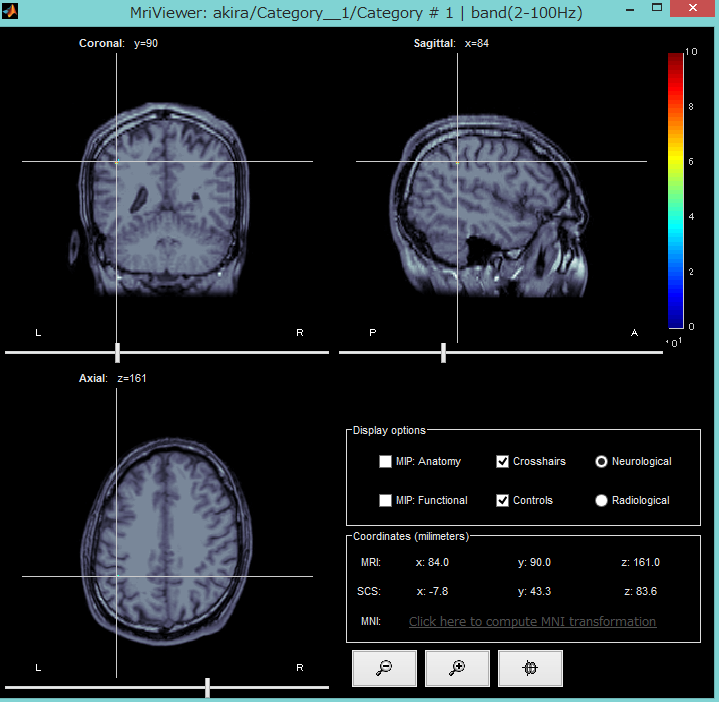
結果は
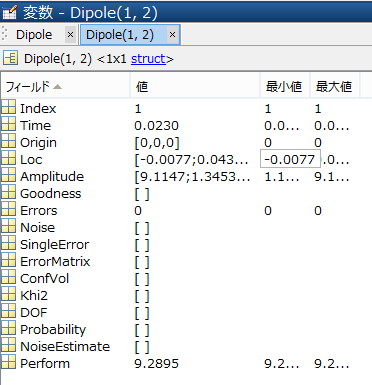
dipoles_fit.matというMATファイルに保存されています。
MATLABで読み込むと、Dipoleという変数があり、時間ごとセル形式で推定結果の情報が保存されています。