MRI画像とCT画像の融合
MATLAB8以降、R2012b以降からImage Processing Toolboxに2つの三次元配列データの位置を自動的にそろえる機能が整備されました。 脳外科ではよく使う機能なので試しに使ってみたのですが、初期設定のままでは残念ながら実用的ではありませんでした。 もう少しparameterのtuningが必要なんだだと思います。
Contents
MRIとCTファイルの読み込み
Image Processing ToolboxのDICOM読み込み関数を使ってDICOMのヘッダー部分とデータ部分を読み込みます。 path1とpath2はそれぞれMRIとCTのDICOMファイルがあるfolderです。
for nn=1:2 eval(sprintf('imagepath=path%d;',nn));%1 MRI 2 CT files=ls(imagepath);% ファイル数×最長ファイル名のcharの配列 if ispc % windows用 nfiles=size(files,1); else % mac/linux用 k=strfind(files,char(10)); nfiles=length(k); files=[char(10),files]; k=[1,k+1]; end; data=[]; info=[]; num=1; for n=1:nfiles if ispc loadfile=strrep(files(n,:),' ','');% 空白文字を削除 else loadfile=files((k(n)+1):(k(n+1)-1)); end; loadfile=[imagepath,loadfile]; try info{num}=dicominfo(loadfile); data(:,:,num)=int16(dicomread(loadfile));% double→int16 num=num+1; catch continue; end; end; num=num-1; SliceLocation=zeros(1,num); for n=1:num SliceLocation(n)=info{n}.SliceLocation; end; [x,y]=sort(SliceLocation);% スライス順に並び替える eval(sprintf('info%d=info(y);',nn)); eval(sprintf('data%d=data(:,:,y);',nn)); end;
画像表示
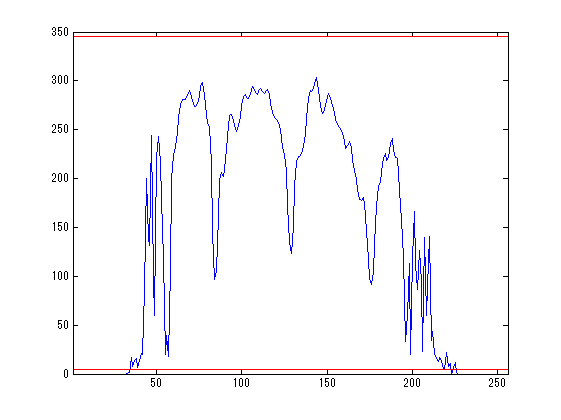
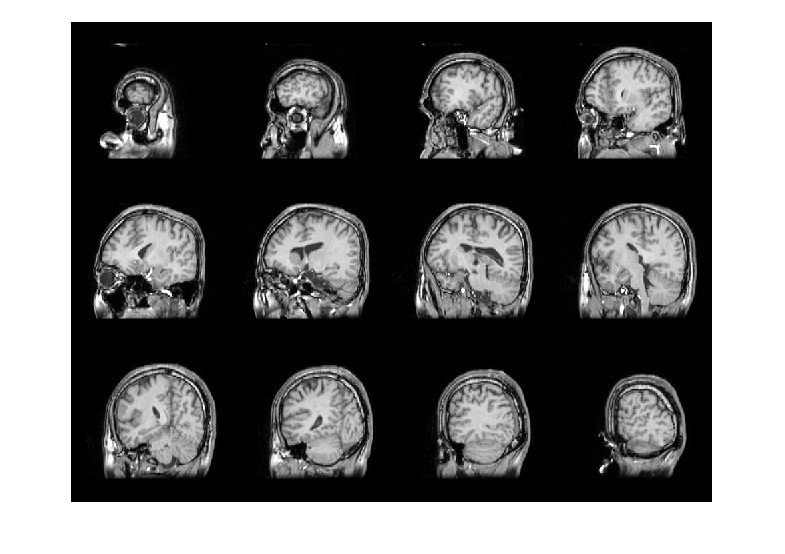
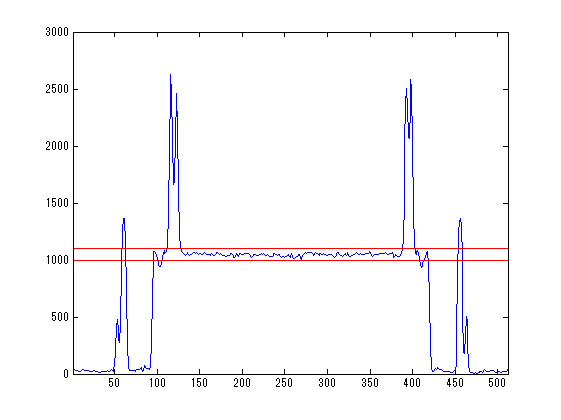
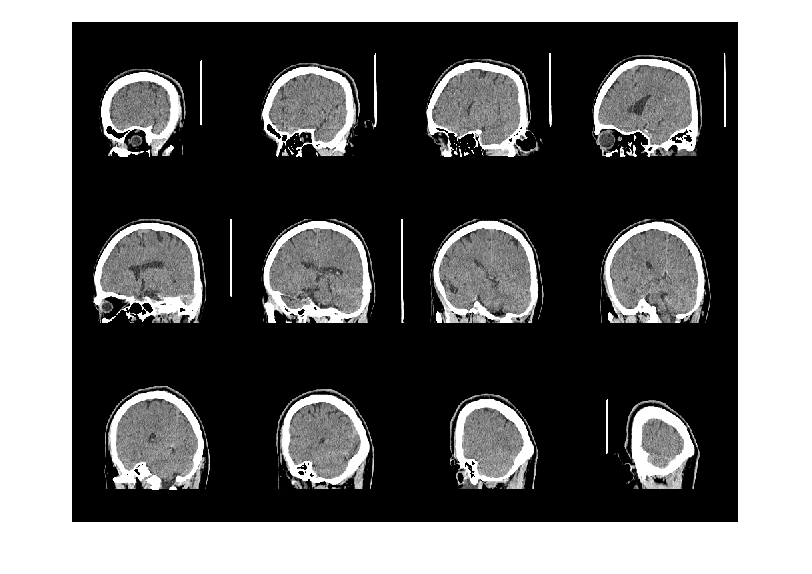
Image Processing Toolboxのmaketform関数、tformarray関数、montage関数を使って画像を表示します。 tformarray関数の出力の1,2,3軸はY軸、X軸、Z軸に対応するので[2,1,3]にしています。 montage関数はint16は使えないのでuint8に変換して表示させます。 方位角45度、天頂角90度、回転角0度の画像をmontage関数で表示してます。 輝度は側面中心点を通る線のDICOM値から脳実質に適当な値を探しました。
for nn=1:2 clear T; eval(sprintf('info=info%d;',nn)); eval(sprintf('data=data%d;',nn)); Dsize=size(data)/2; dicomvalue=squeeze(data(round(Dsize(1)),:,round(Dsize(3)))); scale=[info{1}.PixelSpacing;info{1}.SliceThickness]'; figure; plot(dicomvalue);hold on; set(gca,'xlim',[1,Dsize(2)*2]); xx=[1;Dsize(2)*2]*ones(1,2); if nn==1 clim=[5,345]; else clim=[1000,1100]; end; plot(xx,ones(2,1)*clim,'r'); old_shift=-Dsize; new_shift=round(Dsize.*scale); ang1=45;% 方位角 ang2=90;% 天頂角 ang3=0;% 回転角 ang1=ang1/180*pi; ang2=ang2/180*pi; ang3=ang3/180*pi; Phi=[cos(ang1),-sin(ang1),0;sin(ang1),cos(ang1),0;0,0,1]; Theta=[1,0,0;0,cos(ang2),-sin(ang2);0,sin(ang2),cos(ang2)]; Lambda=[cos(ang3),-sin(ang3),0;sin(ang3),cos(ang3),0;0,0,1]; T=Lambda*Theta*Phi*diag(scale); T1=[eye(4,3),[new_shift';1]]; T2=[[T,zeros(3,1)];[0,0,0,1]]; T3=[eye(4,3),[old_shift';1]]; TT=T1*T2*T3; T=maketform('affine',TT'); resample=makeresampler({'linear','linear','linear'},'fill'); tic; D=tformarray(data,T,resample,[1,2,3],[2,1,3],new_shift*2,[],0); toc clear DD; DD(:,:,1,:)=D; DD=(DD-clim(1))/(clim(2)-clim(1))*255; DD(DD<0)=0;DD(DD>255)=255; DD=uint8(DD); index=round(linspace(1,size(D,3),14)); index([1,end])=[]; figure;montage(DD,'indices',index); end;
経過時間は 7.016832 秒です。 警告: イメージが大きすぎて画面に収まりません。67% で表示します。 経過時間は 7.014341 秒です。 警告: イメージが大きすぎて画面に収まりません。67% で表示します。
RIPEデータの形式に変換
上記のデータでMATLABのImage Processing Toolboxの例「マルチモーダルな3次元医療画像のレジストレーション」のコードを実行します。 The Restrospective Image Registration Evaluation (RIRE)形式に変換します。 helperVolumeRegistration関数は対話方式で回転ができ、両軸は同期して動きます。
for nn=1:2 eval(sprintf('info=info%d;',nn)); eval(sprintf('data=data%d;',nn)); R.Modality=info{1}.Modality; R.SliceThickness=info{1}.SliceThickness; R.Rows=info{1}.Rows; R.Columns=info{1}.Columns; R.PixelSize=info{1}.PixelSpacing'; R.Slices=size(data,3); if nn==1; fixedHeader=R; fixedVolume=single(data); else movingHeader=R; movingVolume=single(data); end; end; helperVolumeRegistration(fixedVolume,movingVolume);
imshowpair関数でMRIとCTを比較
imshowpair関数を使ってMRIとCTの上下中間の面で比較しました。
centerFixed =size(fixedVolume)/2;
centerMoving=size(movingVolume)/2;
centerFixed =round(centerFixed);
centerMoving=round(centerMoving);
figure;title('位置合わせ前のMRIとCT');
imshowpair(movingVolume(:,:,centerMoving(3)), fixedVolume(:,:,centerFixed(3)));

imregister関数を用いて位置合わせの
Image Processing Toolboxの以下の特殊な仕組みを使います。 1+1の進化の最適化構成のためのregistration.optimizer.OnePlusOneEvolutionaryクラス、 相対輝度が同じである一致ピクセル数を最大にするためのregistration.metric.MattesMutualInformationクラス、 ワールド座標用のimref3dクラスです。 尚、「マルチモーダルな3次元医療画像のレジストレーション」では
警告: 最適化が分岐処理されたため、登録に失敗しました。OnePlusOneEvolutionaryOptimizer 最適子の InitialRadius プロパティを減らしてください。
と表示され
optimizer.InitialRadius=6e-3;
としないと、ちゃんと位置合わせできませんでした。
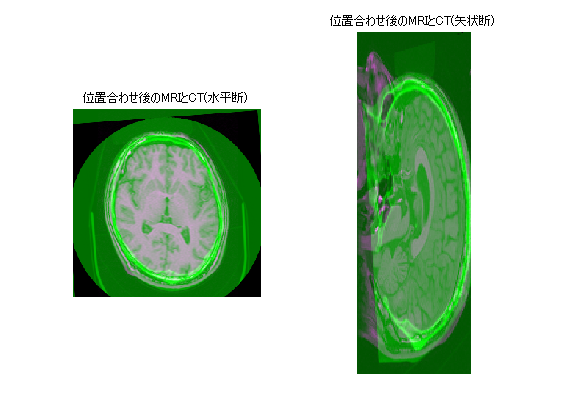
[optimizer,metric]=imregconfig('multimodal'); % 次の2つのコード実行と等価 % optimizer=registration.optimizer.OnePlusOneEvolutionary(); % metric =registration.metric.MattesMutualInformation(); Rfixed =imref3d(size(fixedVolume),fixedHeader.PixelSize(2),fixedHeader.PixelSize(1),fixedHeader.SliceThickness); Rmoving=imref3d(size(movingVolume),movingHeader.PixelSize(2),movingHeader.PixelSize(1),movingHeader.SliceThickness); tic; movingRegisteredVolume=imregister(movingVolume,Rmoving,fixedVolume,Rfixed,'rigid',optimizer, metric); toc; figure; subplot(121); imshowpair(movingRegisteredVolume(:,:,centerFixed(3)),fixedVolume(:,:,centerFixed(3))); title('位置合わせ後のMRIとCT(水平断)'); subplot(122); imshowpair(squeeze(movingRegisteredVolume(:,centerFixed(2),:)),squeeze(fixedVolume(:,centerFixed(2),:))); title('位置合わせ後のMRIとCT(矢状断)'); helperVolumeRegistration(fixedVolume,movingRegisteredVolume);
経過時間は 36.106146 秒です。
imregtform関数とimwarp関数を用いて位置合わせ
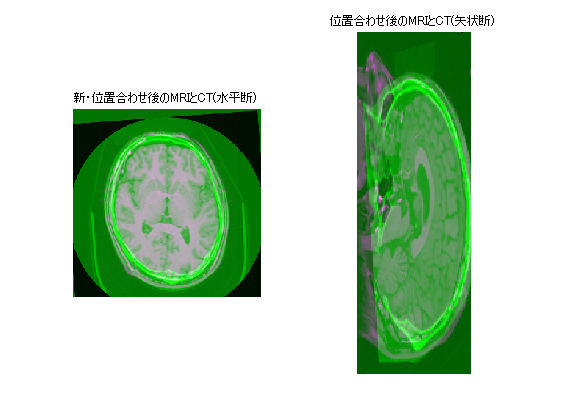
imregtform関数は幾何学的変換を推定し、affine3dクラスを出力します。 transformPointsForward関数はaffine3dクラスの変換行列を用いて座標を変換します。 worldToSubscript関数はワールド座標から行と列の数値ベクトルへ変換します。 imregisterよりも位置合わせの精度がよくなるのではと期待したのですが、あんまり変化ありませんでした。 もう少しtuningが要るみたいです。
tic; geomtform=imregtform(movingVolume,Rmoving,fixedVolume,Rfixed,'rigid',optimizer,metric) toc; centerXWorld=mean(Rmoving.XWorldLimits); centerYWorld=mean(Rmoving.YWorldLimits); centerZWorld=mean(Rmoving.ZWorldLimits); [xWorld,yWorld,zWorld]=transformPointsForward(geomtform,centerXWorld,centerYWorld,centerZWorld); [r,c,p]=worldToSubscript(Rfixed,xWorld,yWorld,zWorld); tic; movingRegisteredVolume2=imwarp(movingVolume,Rmoving,geomtform,'bicubic','OutputView',Rfixed); toc; figure; subplot(121);imshowpair(movingRegisteredVolume2(:,:,centerFixed(3)), fixedVolume(:,:,centerFixed(3))); title('新・位置合わせ後のMRIとCT(水平断)'); subplot(122);imshowpair(squeeze(movingRegisteredVolume2(:,centerFixed(2),:)),squeeze(fixedVolume(:,centerFixed(2),:))); title('位置合わせ後のMRIとCT(矢状断)');
geomtform =
affine3d のプロパティ:
T: [4x4 double]
Dimensionality: 3
経過時間は 37.126211 秒です。
経過時間は 2.577665 秒です。