NeuromagのMRI断面・脳表画像との位置合わせ
Neuromagでは以下の4つの座標系が使われています。 (1).脳磁図センサー(device)座標系、 (2).Polhemusで取得した左耳介、鼻根点、右耳介の3つ点を基準点とする頭(head)座標系、 (3).構造画像であるDICOMデータを計測する際に使われた(MRI)座標系、 (4).画像上(slice)の座標系。
センサー・頭座標間の変換行列は、脳磁場の計測直前にHead Position Indicator コイルに電流を流し、計測用ワークステーションが自動で4つの等価電流双極子推定を行うことで確立されます。 頭・MRI座標間の変換行列は、解析者がMRI Labを起動して、MRI画像上で上記3つの基準点を選ぶことで確立されます。 MRI・画像間の変換行列は、CT・MRIの元画像の場合は、DICOMファイルに書き込まれていますが、 脳表画像の場合はViewBrainのrendererツールが自動で確立していきます。
Contents
FIFFファイルの読み込み
ここでは各種FIFFファイルを読み込み、とりあえずDICOM ACCESSで作成されたsets以下のFIFファイルと、 それをもとにSEG LABで作成した3D.FIFファイルをhelperVolumeRegistration関数で比較してみました。 尚、前者はint16、後者はuint8で、両者とも位置合わせ後です。 前者ではDICOMファイルの並び方によっては、画像の順番が狂っていることが有るので注意が必要です
for nn=1:4 switch nn case 1;filename='hasizume_akira_coregistered_with_RSEF.fif';% DICOM Access後MriLabで位置合わせ case 2;filename='hasizume_akira_coregistered_with_RSEF_3D.fif';% SegLabで3D化 case 3;filename='bhasizume_akira_coregistered_with_RSEF_3D.scenes.fif';% 脳表画像 case 4;filename='SEF_median_ave.fif';%脳磁図データ end; F=hns_loadfiff4(filename); if isfield(F,'dcmfiles') nf=size(F.dcmfiles,2); slice2MR=zeros(3,8,nf); for n=1:nf [~,name,ext]=fileparts(F.dcmfiles{n}); loadfile=[name,ext]; info=dicominfo(loadfile); e0=info.ImagePositionPatient/1000;% mmからmに ex=info.ImageOrientationPatient(1:3)'; ey=info.ImageOrientationPatient(4:6)'; e0(1)=-e0(1);ex=-ex;% 右手系と左手系の反転 ez=cross(ex,ey); M=[[[ex;ey;ez],e0];0 0 0 1]; M=[inv(M),M]; slice2MR(:,:,n)=M(1:3,:); F.Volume3D(:,:,n)=int16(dicomread(loadfile)); end; row=double(info.Rows); column=double(info.Columns); F.scale=[row,column,[row,column].*info.PixelSpacing'/1000,nf]; end; eval(sprintf('F%d=F;',nn)); end; clear F; helperVolumeRegistration(F1.Volume3D,F2.Volume3D); view([-80,10]);
警告: WAVPLAY は将来のリリースで削除される予定です。代わりに AUDIOPLAYER を使用してください。 警告: WAVPLAY は将来のリリースで削除される予定です。代わりに AUDIOPLAYER を使用してください。 警告: WAVPLAY は将来のリリースで削除される予定です。代わりに AUDIOPLAYER を使用してください。 警告: WAVPLAY は将来のリリースで削除される予定です。代わりに AUDIOPLAYER を使用してください。

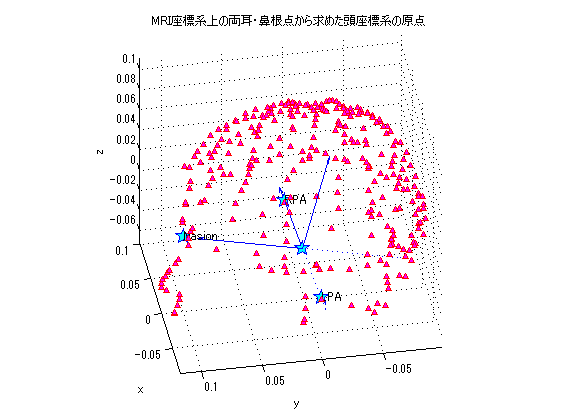
Symbolic Math Toolboxを使って両耳・鼻根点から頭座標系を構築
左耳がP1[p1x,p1y,p1z],右耳がP2[p2x,p2y,p2z],鼻根点がP3[p3x,p3y,p3z], 頭座標の原点がP0[p0x,p0y,p0z]とします。 P0はP1とP2を結ぶ線にP3からおろした垂線の足です。 頭座標系を作成した後、頭座標系とMRI座標系間の変換行列を求め、MRI LABの結果と比較しました。 微妙な誤差がありますが、singleとdoubleの計算結果の違いだと思います。
syms p0x p0y p0z p1x p1y p1z p2x p2y p2z p3x p3y p3z real; [sol_p0x,sol_p0y,sol_p0z]=solve(... (p2x-p1x)/(p2x-p0x)==(p2y-p1y)/(p2y-p0y),... (p2x-p1x)/(p2x-p0x)==(p2z-p1z)/(p2z-p0z),... (p3x-p0x)*(p2x-p1x)+(p3y-p0y)*(p2y-p1y)+(p3z-p0z)*(p2z-p1z)==0.... ); sol_p0x sol_p0y sol_p0z P=F2.DigitPts; P0=[... double(subs(sol_p0x,[p1x,p1y,p1z,p3x,p3y,p3z,p2x,p2y,p2z],reshape(F2.DigitPts',[1,9])));... double(subs(sol_p0y,[p1x,p1y,p1z,p3x,p3y,p3z,p2x,p2y,p2z],reshape(F2.DigitPts',[1,9])));... double(subs(sol_p0z,[p1x,p1y,p1z,p3x,p3y,p3z,p2x,p2y,p2z],reshape(F2.DigitPts',[1,9])))]; P0=P0'; figure('color',[1,1,1]); h=plot3(P(:,1),P(:,2),P(:,3),'bo');hold on; text(P(1,1),P(1,2),P(1,3),'LPA'); text(P(2,1),P(2,2),P(2,3),'Nasion'); text(P(3,1),P(3,2),P(3,3),'RPA'); h=[h;plot3(P0(1),P0(2),P0(3),'bo')]; set(h,'Marker','pentagram','MarkerSize',12,'MarkerFaceColor',[0,1,1]); xlabel('x');ylabel('y');zlabel('z');grid on; daspect([1,1,1]); title('MRI座標系上の両耳・鼻根点から求めた頭座標系の原点'); % 頭座標系のx方向 y方向 z方向の単位ベクトルを求める vec=P-ones(3,1)*P0; vec=vec./(sum(vec.^2,2).^0.5*ones(1,3)); vec(1,:)=vec(3,:);% 右耳介方向がX軸の正の方向 vec(4,:)=-vec(1,:);% 作図用 vec(5,:)=-vec(2,:);% 作図用 vec(3,:)=cross(vec(1,:),vec(2,:));% HEAD2MRIの回転部分 拡大縮小・平行移動はまだ scale=0.1;% 適当に縮小して表示 for n=1:5 h=quiver3(P0(1),P0(2),P0(3),vec(n,1)*scale,vec(n,2)*scale,vec(n,3)*scale,'b'); if n>3;set(h,'linestyle',':');end; end; % 基準点をまとめる P0=[P(1,:);P(3,:);P0;P(2,:)]'; P0(4,:)=1; vec(4:5,:)=[]; % digitized points P=F4.DigitPts'; P(4,:)=1; MTX=F2.HEAD2MRI(:,1:4); MTX(4,:)=[0,0,0,1]; P=MTX*P; plot3(P(1,:),P(2,:),P(3,:),'r^','MarkerSize',5,'MarkerFaceColor',[1,0,1]); axis tight; view([-100,35]); rotate3d on; % 検算 HEAD2MRI=[inv(vec),P0(1:3,3)];% 順番をずらした。 k=F2.HEAD2MRI(:,1:4)-HEAD2MRI; max(abs(k(:)))
sol_p0x = (p3x*p1x^2 - 2*p3x*p1x*p2x - p1x*p1y*p2y + p3y*p1x*p1y + p1x*p2y^2 - p3y*p1x*p2y - p1x*p1z*p2z + p3z*p1x*p1z + p1x*p2z^2 - p3z*p1x*p2z + p3x*p2x^2 + p2x*p1y^2 - p2x*p1y*p2y - p3y*p2x*p1y + p3y*p2x*p2y + p2x*p1z^2 - p2x*p1z*p2z - p3z*p2x*p1z + p3z*p2x*p2z)/(p1x^2 - 2*p1x*p2x + p2x^2 + p1y^2 - 2*p1y*p2y + p2y^2 + p1z^2 - 2*p1z*p2z + p2z^2) sol_p0y = (p1x^2*p2y - p1x*p2x*p1y - p1x*p2x*p2y + p3x*p1x*p1y - p3x*p1x*p2y + p2x^2*p1y - p3x*p2x*p1y + p3x*p2x*p2y + p3y*p1y^2 - 2*p3y*p1y*p2y - p1y*p1z*p2z + p3z*p1y*p1z + p1y*p2z^2 - p3z*p1y*p2z + p3y*p2y^2 + p2y*p1z^2 - p2y*p1z*p2z - p3z*p2y*p1z + p3z*p2y*p2z)/(p1x^2 - 2*p1x*p2x + p2x^2 + p1y^2 - 2*p1y*p2y + p2y^2 + p1z^2 - 2*p1z*p2z + p2z^2) sol_p0z = (p1x^2*p2z - p1x*p2x*p1z - p1x*p2x*p2z + p3x*p1x*p1z - p3x*p1x*p2z + p2x^2*p1z - p3x*p2x*p1z + p3x*p2x*p2z + p1y^2*p2z - p1y*p2y*p1z - p1y*p2y*p2z + p3y*p1y*p1z - p3y*p1y*p2z + p2y^2*p1z - p3y*p2y*p1z + p3y*p2y*p2z + p3z*p1z^2 - 2*p3z*p1z*p2z + p3z*p2z^2)/(p1x^2 - 2*p1x*p2x + p2x^2 + p1y^2 - 2*p1y*p2y + p2y^2 + p1z^2 - 2*p1z*p2z + p2z^2) ans = 7.4457e-08
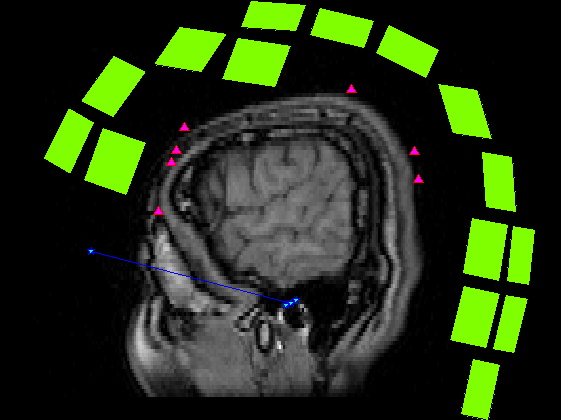
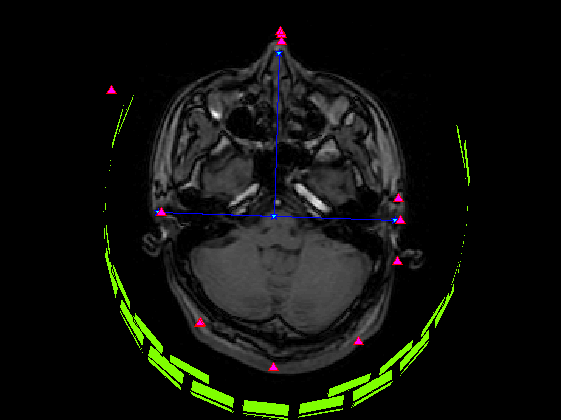
基準点・頭皮プロット点・センサーを脳断面画像に重畳
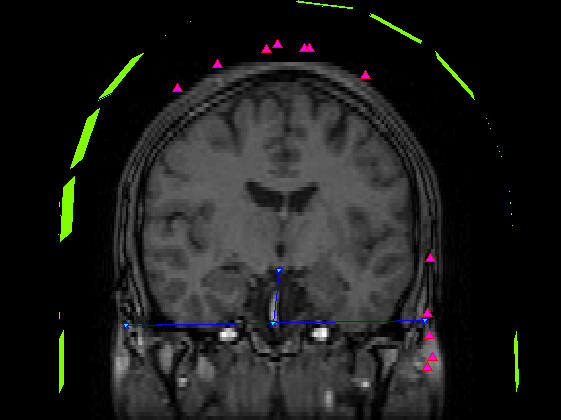
MRI座標上のモノを実際の脳断面画像上に表示してみます。 尚、MATLABの表示の問題でY軸方向にマイナスをかけています。
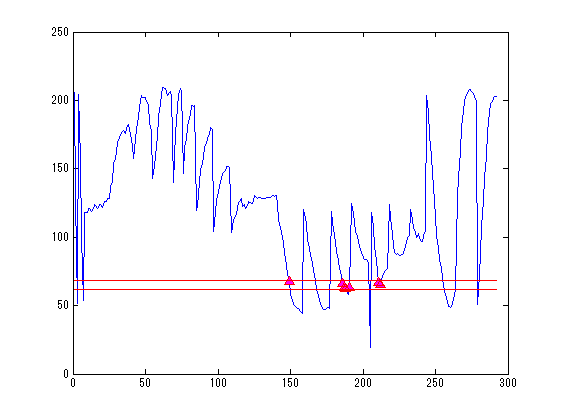
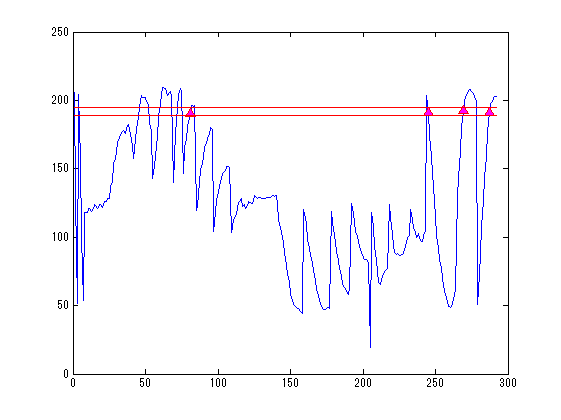
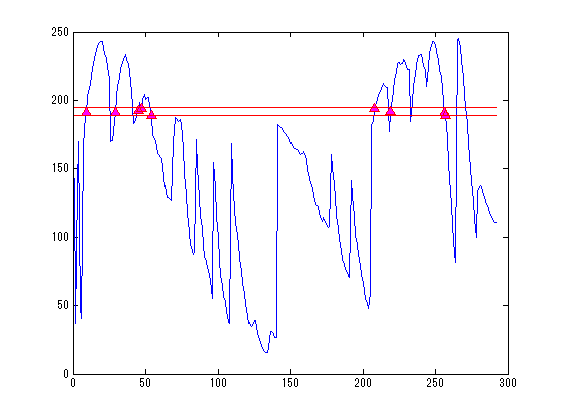
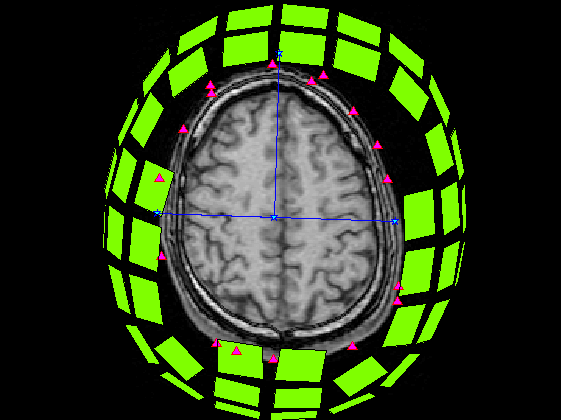
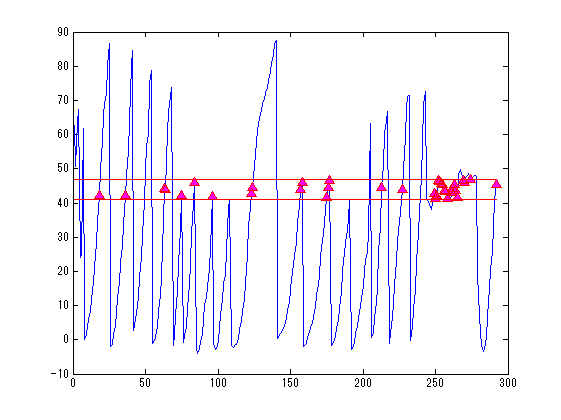
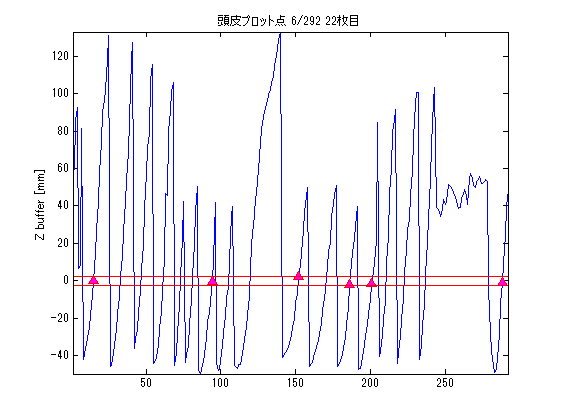
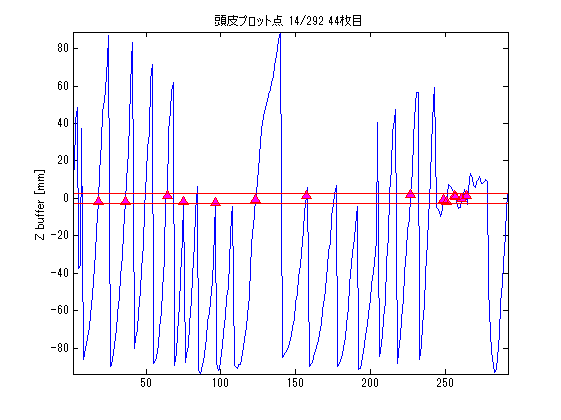
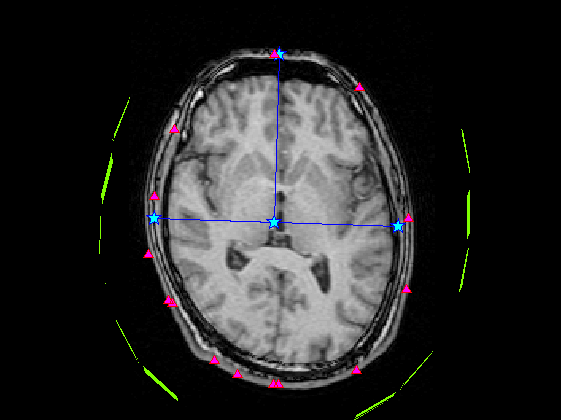
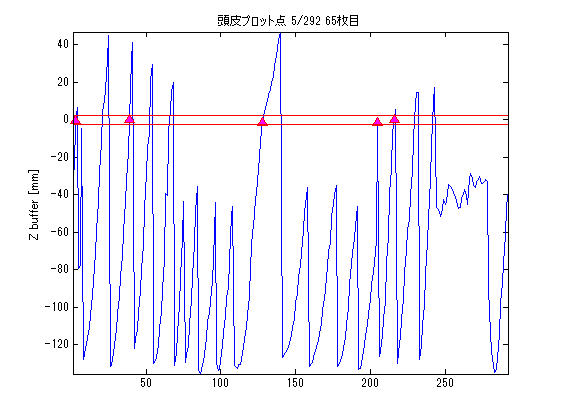
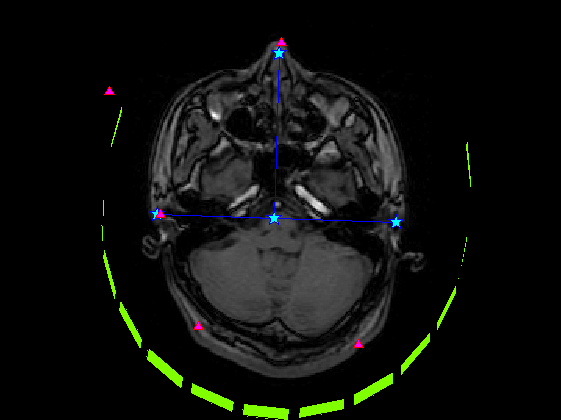
SliceNo=round(linspace(1,F2.scale(5),5)); SliceNo([1,end])=[]; scale=diag([F2.scale(1:2)./F2.scale(3:4),1000,1]);% zubbfer方向はmをmmに np=size(P,2); lim=2.5;% 厚さ±2.5mmを表示 lim2=15;% 厚さ±15mmを表示 S0=F4.ChInform(1:3,1:3:306);% センサー座標系のセンサー位置 Sx=F4.ChInform(4:6,1:3:306);% センサー座標系の接平面方向 Sy=F4.ChInform(7:9,1:3:306);% センサー座標系の接平面方向 Sz=F4.ChInform(10:12,1:3:306);% センサー座標系の法線方向 S0(4,:)=1; MTX4=[F4.MEG2HEAD(:,1:4);0 0 0 1];% センサー座標・頭座標間の変換行列 MTX2=[F3.HEAD2MRI(:,1:4);0 0 0 1]*MTX4;% センサー座標・MRI座標感の変換行列 % MTX3=[F3.HEAD2MRI(:,1:4);0 0 0 1]*MtX4;% 同上(異なるFIFFファイル) S0=MTX2*S0; Sx=MTX2(1:3,1:3)*Sx; Sy=MTX2(1:3,1:3)*Sy; Sz=MTX2(1:3,1:3)*Sz; wx=0.014;wz=0.0003; S=[S0(1:3,:)+wz*Sz;...% MRI座標系上のセンサーの中心点の位置 S0(1:3,:)+wx*Sx+wx*Sy+wz*Sz;... S0(1:3,:)+wx*Sx-wx*Sy+wz*Sz;... S0(1:3,:)-wx*Sx-wx*Sy+wz*Sz;... S0(1:3,:)-wx*Sx+wx*Sy+wz*Sz]; S=reshape(S,[3,5*102]); S(4,:)=1; for n=1:length(SliceNo) MTX=[squeeze(F2.slice2MR(:,5:8,SliceNo(n)));0 0 0 1]; p0=scale*MTX*P0; p=scale*MTX*P; figure('color',[1,1,1]); plot(1:np,p(3,:));hold on; x=find(p(3,:)<lim & p(3,:)>-lim); plot(x,p(3,x),'r^','MarkerFaceColor',[1,0,1]); plot([1;np]*ones(1,2),lim*ones(2,1)*[-1,1],'r'); ylabel('Z buffer [mm]'); axis tight; title(sprintf('頭皮プロット点 %d/%d %d枚目',length(x),np,SliceNo(n))); s=scale*MTX*S; s=reshape(s(1:3,:),[3*5,102]); z=find(s(3,:)<lim2 & s(3,:)>-lim2); figure('color',[1,1,1]*0,'InvertHardCopy','off'); imagesc(squeeze(F2.Volume3D(:,:,SliceNo(n)))); colormap(gray);daspect([1,1,1]);hold on; h=plot(p0(1,:),-p0(2,:),'b','Marker','pentagram'); set(h,'MarkerSize',12,'MarkerFaceColor',[0,1,1]); plot(p(1,x),-p(2,x),'r^','MarkerFaceColor',[1,0,1]); if isempty(z);continue;end; h=fill(s(4:3:15,z),-s(5:3:15,z),'g','FaceColor',[0.5,1,0]); set(gca,'position',[0,0,1,1]); axis off;axis tight; end;
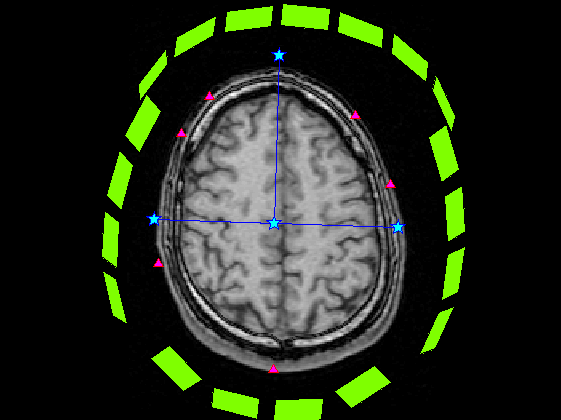
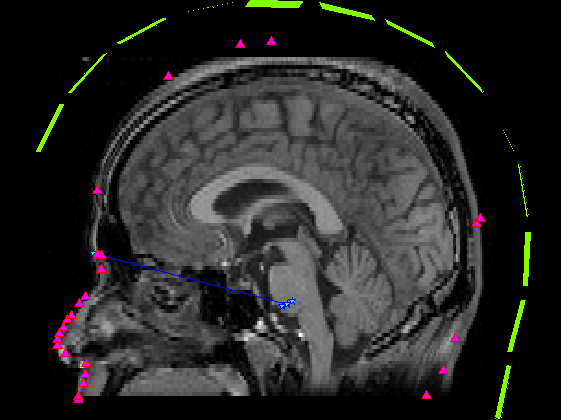
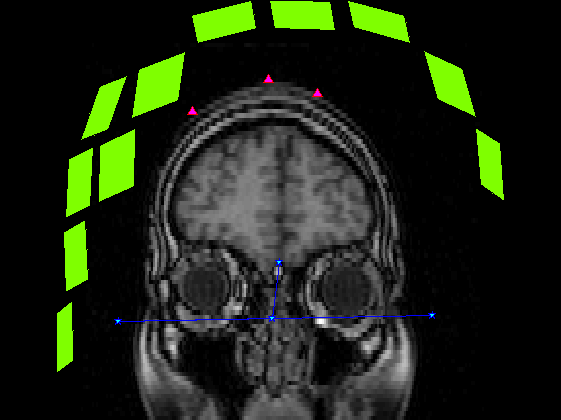
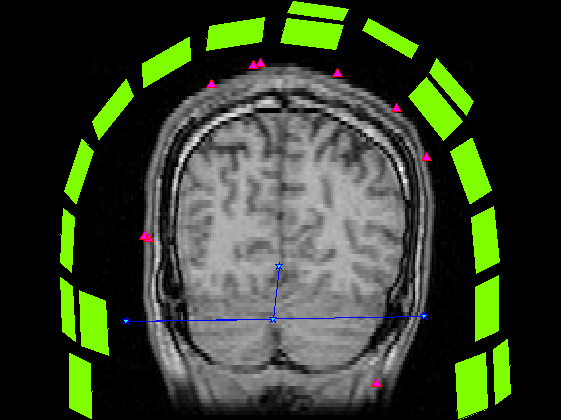
基準点・頭皮プロット点・センサーを脳表画像に重畳
頭・MRI座標感の変換行列が同一として、脳断面画像に重畳した際のでMRI座標上のデータはそのまま使います。 奥行き方向であるZ buffer情報はありません。そこでセンサーの法線方向を計算し、適当に間引いています。 ViewBrainもこれくらいできたらいいんだけど・・・。
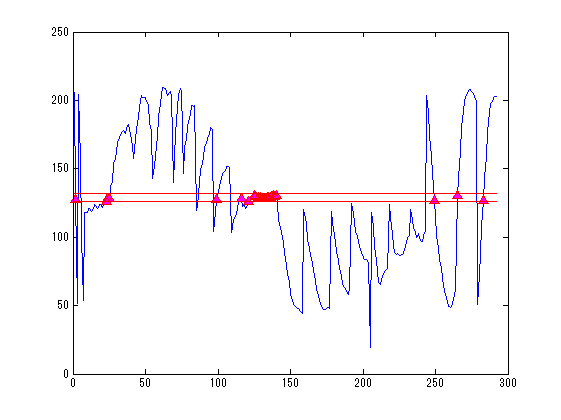
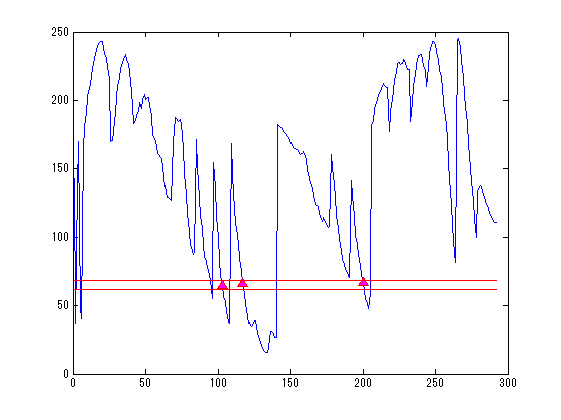
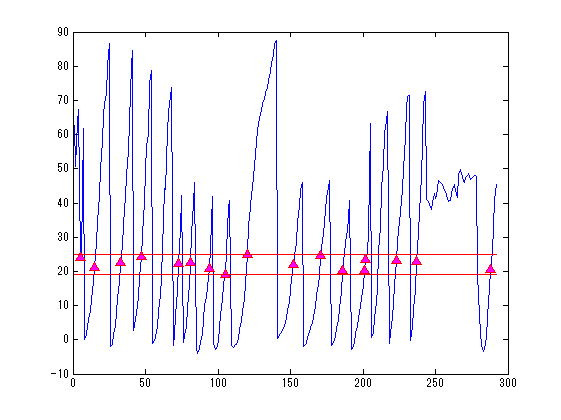
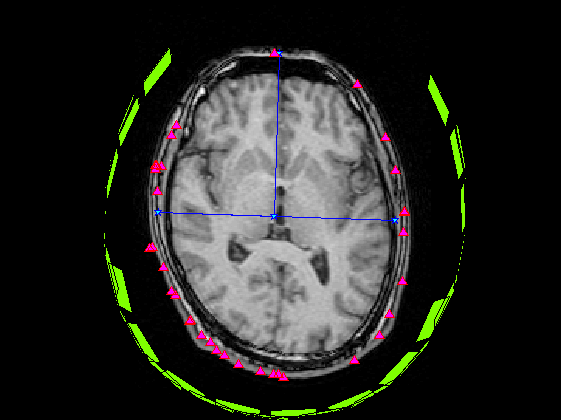
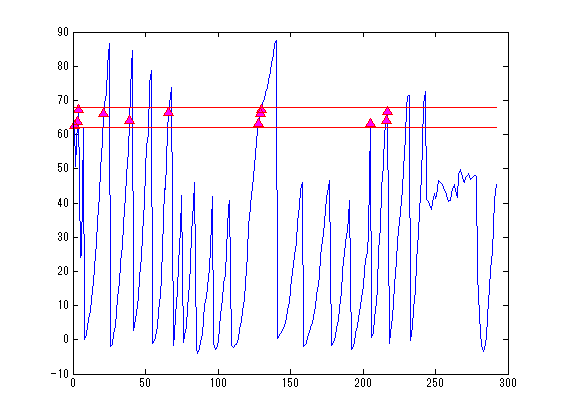
SliceNo=round(linspace(1,F3.scale(5),5)); SliceNo([1,end])=[]; scale=diag([F3.scale(1:2)./F3.scale(3:4),1000,1]);% zubbfer方向はmをmmに Sz(4,:)=1; for n=1:length(SliceNo) MTX=[squeeze(F3.slice2MR(:,5:8,SliceNo(n)));0 0 0 1]; p0=scale*MTX*P0; p=scale*MTX*P; s=scale*MTX*S; s=reshape(s(1:3,:),[3*5,102]); sz=MTX*Sz; z=find(sz(3,:)>-0.5); figure('color',[1,1,1]*0,'InvertHardCopy','off'); imagesc(squeeze(F3.VolImage(:,:,SliceNo(n)))); colormap(gray);daspect([1,1,1]);hold on; h=plot(p0(1,:),-p0(2,:),'b','Marker','pentagram'); set(h,'MarkerSize',12,'MarkerFaceColor',[0,1,1]); plot(p(1,:),-p(2,:),'r^','MarkerFaceColor',[1,0,1]); if isempty(z);continue;end; h=fill(s(4:3:15,z),-s(5:3:15,z),'g','FaceColor',[0.5,1,0],'FaceAlpha',0.2); set(h,'LineWidth',1,'EdgeColor',[0.5,1,0]); set(gca,'position',[0,0,1,1]); axis off;axis tight; end;
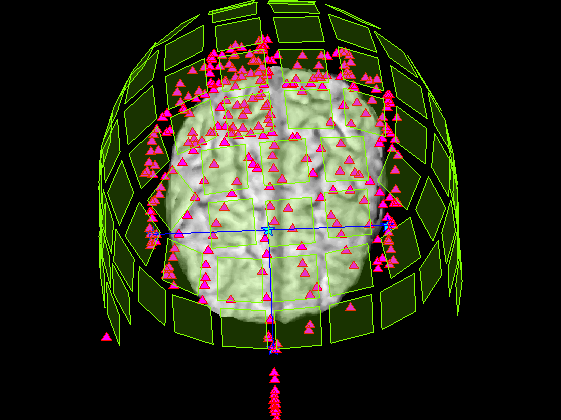
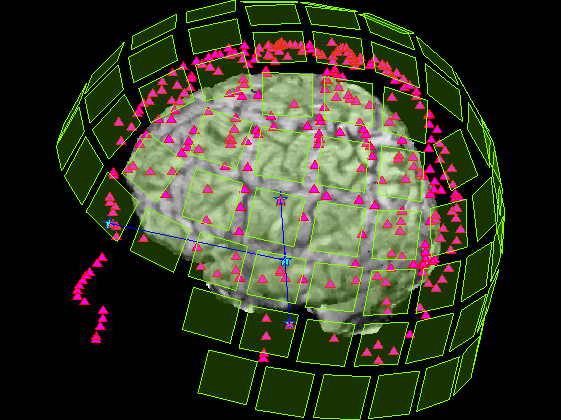
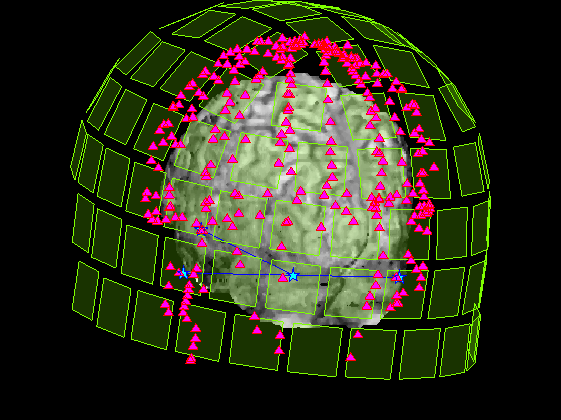
基準点・頭皮プロット点・センサーをボリュームデータ上に重畳
MRI座標系と画像座標系間の変換行列は有るのですが、MRI座標系とデータボリューム間での変換行列はありません。 なので、作ります、といってもscaleのところをmmから枚に返るだけです。 slice2MR=squeeze(F2.slice2MR(:,1:3,1)); scale=[F2.scale(1:2)./F2.scale(3:4),F2.scale(5),1]; T=squeeze(F2.slice2MR(:,4,:));
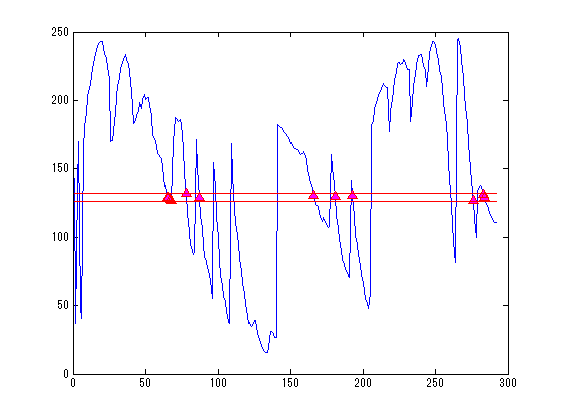
zscale=F2.slice2MR(:,4,end)-F2.slice2MR(:,4,1); zscale=sqrt(sum(squeeze(zscale).^2)); scale=[F2.scale(1:2)./F2.scale(3:4),(F2.scale(5)-1)/zscale]; MTX=diag([scale,1])*[squeeze(F2.slice2MR(:,5:8,1));0 0 0 1]; MTX=diag([1,-1,1,1])*MTX; p0=MTX*P0; p=MTX*P;np=size(p,2); s=MTX*S; s=reshape(s(1:3,:),[3*5,102]); lim=3;% 頭皮プロット転用スライス厚±3枚 lim2=20;% センサー用スライス厚±20枚 for ax=1:3 SliceNo=round(linspace(1,size(F2.Volume3D,ax),5)); SliceNo([1,end])=[]; for n=1:length(SliceNo); switch ax case 2;V=F2.Volume3D(SliceNo(n),:,:);V=squeeze(V)';ax1=1;ax2=3; case 1;V=F2.Volume3D(:,SliceNo(n),:);V=squeeze(V)';ax1=2;ax2=3; case 3;V=F2.Volume3D(:,:,SliceNo(n));V=squeeze(V); ax1=1;ax2=2; end; x=find(p(ax,:)>=SliceNo(n)-lim & p(ax,:)<=SliceNo(n)+lim); figure('color',[1,1,1]);plot(1:np,p(ax,:));hold on; plot(x,p(ax,x),'r^','MarkerFaceColor',[1,0,1]); plot([1;np]*ones(1,2),SliceNo(n)+lim*ones(2,1)*[-1,1],'r'); z=find(s(ax,:)>=SliceNo(n)-lim2 & s(ax,:)<=SliceNo(n)+lim2); figure('color',[1,1,1]*0,'InvertHardCopy','off'); imagesc(V);hold on; colormap(gray); h=plot(p0(ax1,:),p0(ax2,:),'b','Marker','pentagram','MarkerFaceColor',[0,1,1]); plot(p(ax1,x),p(ax2,x),'r^','MarKerFaceColor',[1,0,1]); daspect([scale(ax1),scale(ax2),scale(ax)]); if isempty(z);continue;end; h=fill(s((3+ax1):3:15,z),s((3+ax2):3:15,z),'g','FaceColor',[0.5,1,0]); axis tight;axis off;set(gca,'position',[0,0,1,1]); end; end;