BrainStormやMNE-Cで使えるようにする
BrainSuiteの皮髄境界抽出画像と左右半球ラベル画像から皮髄境界メッシュを作成
FreeSurferのMGZやSURFファイルを読む関数Mファイル
BrainVISAやBrainSuiteのファイルをFreeSurferのMGZファイルやSURFファイルに変換する関数Mファイル
BrainSuiteのDFSファイルを読み込む関数Mファイル
はじめに
BrainStormやMNE-Cは通常ではFreeSurferで作成した脳ボクセル抽出データや、大脳皮質メッシュデータなどを用いて電流源推定しています。
ただし、FreeSurferは計算処理に健常者であっても10時間程度かかり、気軽に電流源推定を、というわけにはいきません。
BrainVISAやBrainSuiteでは計算時間がかなり高速化されています。
そこでBrainVISAやBrainSuiteで作成した脳ボクセル抽出データ、大脳皮質メッシュデータをBrainStormやMNE-Cで使えるかどうか試しました。
BrainStormで使えるようにする
BrainsStormは優秀です。結論から先に言うと、そのまま使えました。BSはBrainSuite、BVはBrainVISA、FSはFreeSurferの略です。
MRIデータ
構造画像であるMRIデータは以下の通りです。
BrainSuiteとBrainVISAのakira.niiは水平断のDICOMファイル×上下方向のスライスを1つのファイルにしたものです。0.9375×0.9375×2mm3のボクセルからなる256×256×86のNIfTIファイルです。軸は左右、前後、上下方向の順になっています。
BrainSuiteのファイルは〜/どっか/名前.nii.gzです。
BrainVISAのファイルは〜/名前/t1mri/default_acquisition/名前.nii.gzです。
FreeSurferはいろいろ処理して1mm3のボクセルからなる256×256×256の独自のMGZファイルです。軸は左右、上下、前後の順になっています。
FreeSurferのファイルは〜/名前/mri/T1.mgzです。
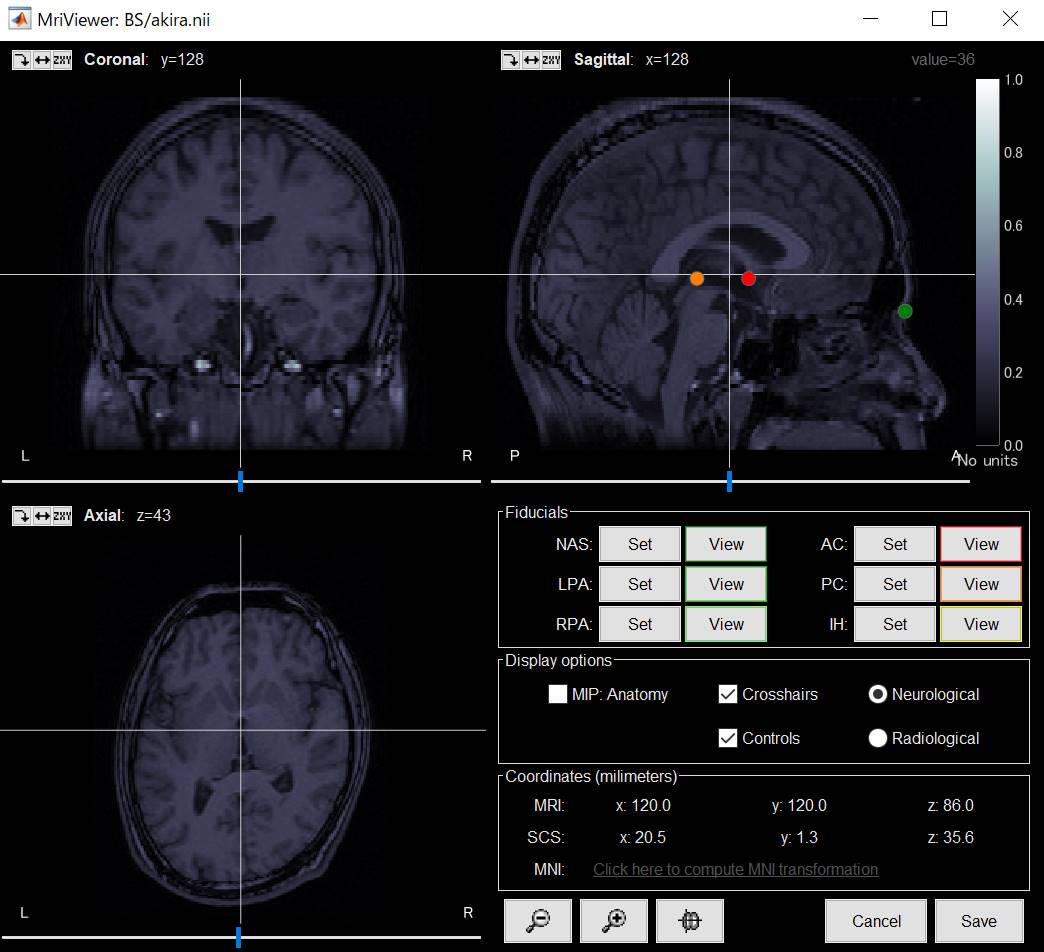
BrainStormは鼻根点・両耳珠、前後の交連、大脳半球間を手動で定義すること軸方向を決定しています。
メッシュデータ
BrainStormでは座標合わせ用に頭皮メッシュファイルが、電流源推定用に皮質表面のメッシュファイルを使います。
頭皮メッシュファイルはMRIデータからGenerate head surfaceを使うことで作成できます。
皮質メッシュファイルは計算量を減らすためメッシュ数は15000程度に減らします。
BrainStormは優秀でメッシュファイルから皮質の表面・皺部分を自動的に計算して色分けする機能があります。
import surfacesで読み込むとき
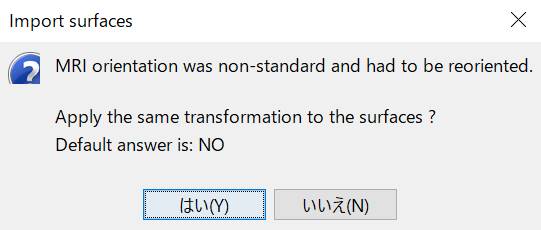
と出ますが、「はい」を押せばたぶんなんとかいけます。
対応するメッシュファイルは以下の通りです。
BrainSuiteは独自のDFSファイルです。
import surfacesで対応する拡張子にDFSファイルはないのですが、大丈夫です。読めます。
〜/どっか/名前.scalp.dfsファイルと
〜/どっか/名前.pial.cortex.dfsファイルをimport surfacesで読み込みます。
左右半球に分ける前です。左右半球に分けた名前.left.pial.cortex.dfsと名前.right.pial.cortex.dfsを読み込んで一つにしてもいいのですが、手間なだけなのでしません。
BrainVISAはGIfTIファイルです。
〜/名前/t1mri/default_acquisition/default_analysis/segmentation/mesh/名前_head.giiファイルと
〜/名前/t1mri/default_acquisition/default_analysis/segmentation/mesh/名前_Lhemi.giiと名前_Rhemi.giiファイルです。
FreeSurferはsurfファイルです。
〜/名前/surf/lh.pialとrh.pialです。
頭皮メッシュファイルはないのでT1.mgzからgenerate head surfaceで作成します。
電流源推定(SEF N20m)の比較
右正中神経電気刺激の体性感覚誘発磁場(Somatosensory Evoked Field SEF)のデータを読み込みます。
いきなりImport MEG/EEGを選択すると、Signal Space Projection (SSP)が無効にされるので、一旦Review raw fileで読み込みます。
![]()
するとSSPをかけるかどうか聞いてきますので全部にcheckを入れ、Saveを押します。
MRI registration→Editで座標合わせをします。
Compute head modelを選択します。
OKを押します。
Link to raw fileからNoise covariance→Compute from recordingsを選択します。
OKを押します。
Compute sources [2018]を選択します。
OKを押します。
Cortical activations→Display on cortexを選択します。
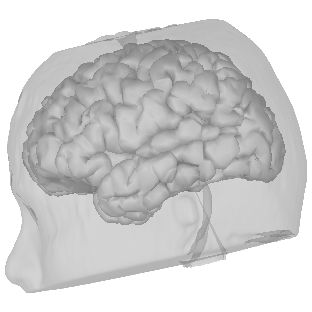
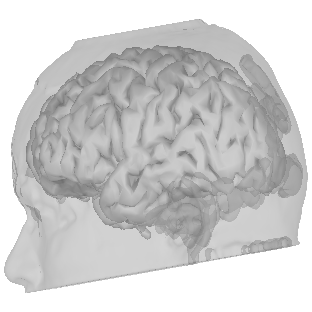
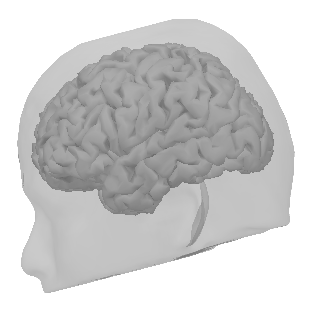
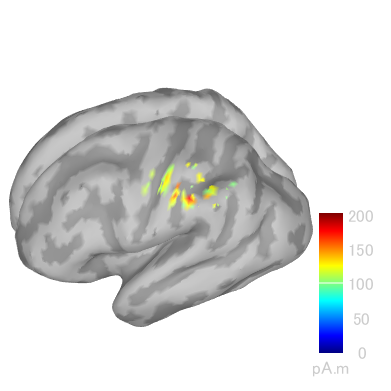
BrainSuiteで22msec時の結果です。
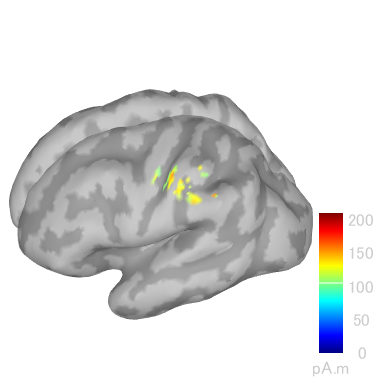
BrainVISAで22msec時の結果です。
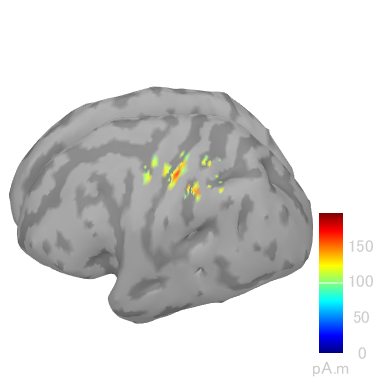
FreeSurferで22msec時の結果です。
MNE-Cで使えるようにする
MNE-CはFreeSurferの決まったフォルダの決まったファイルを使いますので、BrainSuiteとBrainVISA側でFreeSurferに準拠したフォルダとファイルを用意する必要があります。またBrainSuiteは皮質を膨らましたinflated cortexや脳表の曲率を示すファイルを作成する機能がありません。結果としてBrainSuiteで処理したファイルをBrainVISAで不足分を補充し、FreeSurfer用のファイルに変換するという手順となります。
MRIデータ
MNE-CはFreeSurferの
〜/mri/T1.mgz
〜/mri/brain.mgz
の2つのファイルが必要です。以下を実行します。なおhns_loadfreesurferはこのHome Pageの最後に公開しています。
clear;
filename='〜/mri/T1.mgz';
F1=hns_loadfreesurfer(filename);
filename='〜/mri/brain.mgz';
F2=hns_loadfreesurfer(filename);
figure;
m=[F1.width,F1.height,F1.depth]/2;
p=[F1.spacingx,F1.spacingy,F1.spacingz];
for n=1:6
switch n
case
1;data=F1.data(:,:,m(3));ds=[p(1),p(2),p(3)];
case
2;data=F1.data(:,m(2),:);ds=[p(1),p(3),p(2)];
case 3;data=F1.data(m(1),:,:);ds=[p(2),p(3),p(1)];
case
4;data=F2.data(:,:,m(3));ds=[p(1),p(2),p(3)];
case
5;data=F2.data(:,m(2),:);ds=[p(1),p(3),p(2)];
case
6;data=F2.data(m(1),:,:);ds=[p(2),p(3),p(1)];
end
subplot(2,3,n);
imagesc(squeeze(data));
daspect(ds);
end
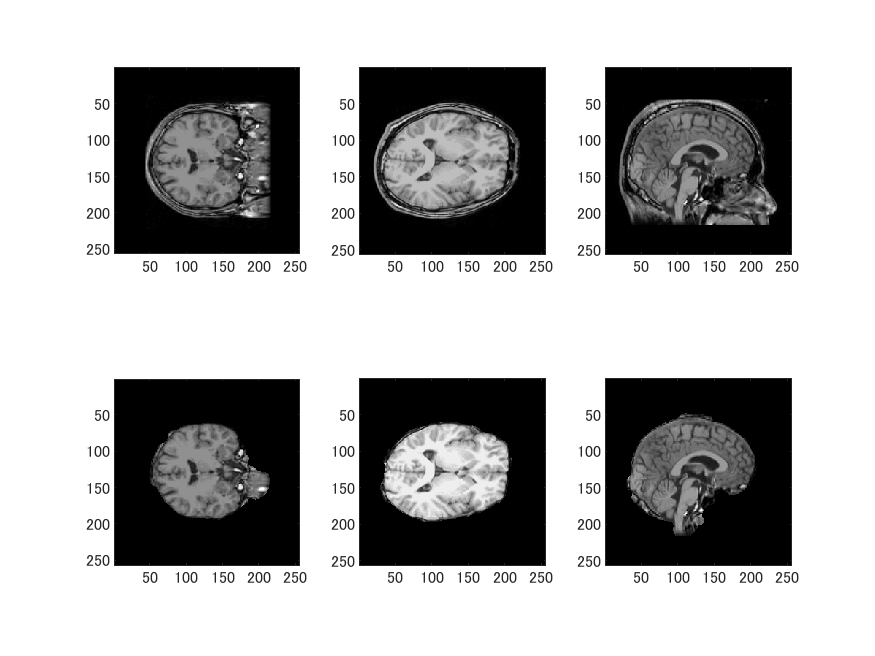
colormap(gray(256));
上段がT1.mgz、下段がbrain.mgzです。
BrainVISAで該当するファイルは以下の通りです。
〜/名前/t1mri/default_acquisition/名前.nii.gzファイルと
〜/名前/t1mri\default_acquisition/default_analysis/segmentation/brain_名前.nii.gzファイルです。
brain_名前.nii.gはマスクデータですので元の名前.nii.gzと掛け算してFreeSurferのbrain.mgz風に表示しています。
clear;
filename='/名前/t1mri/default_acquisition/akira.nii.gz';
F1.data=niftiread(filename);
F1.info=niftiinfo(filename);
filename='/名前/t1mri/default_acquisition/default_analysis/segmentation/brain_akira.nii.gz';
F2.data=niftiread(filename);
F2.info=niftiinfo(filename);
F2.data(F2.data>0)=1;
F2.data(F2.data<1)=0;
F2.data=F1.data.*F2.data;
figure;
m=F1.info.ImageSize/2;
p=F1.info.PixelDimensions;
for n=1:6
switch n
case 1;data=F1.data(:,:,m(3));ds=[p(1),p(2),p(3)];
case
2;data=F1.data(:,m(2),:);ds=[p(1),p(3),p(2)];
case
3;data=F1.data(m(1),:,:);ds=[p(2),p(3),p(1)];
case
4;data=F2.data(:,:,m(3));ds=[p(1),p(2),p(3)];
case
5;data=F2.data(:,m(2),:);ds=[p(1),p(3),p(2)];
case
6;data=F2.data(m(1),:,:);ds=[p(2),p(3),p(1)];
end
subplot(2,3,n);
imagesc(squeeze(data));
daspect(ds);
end
colormap(gray(256));
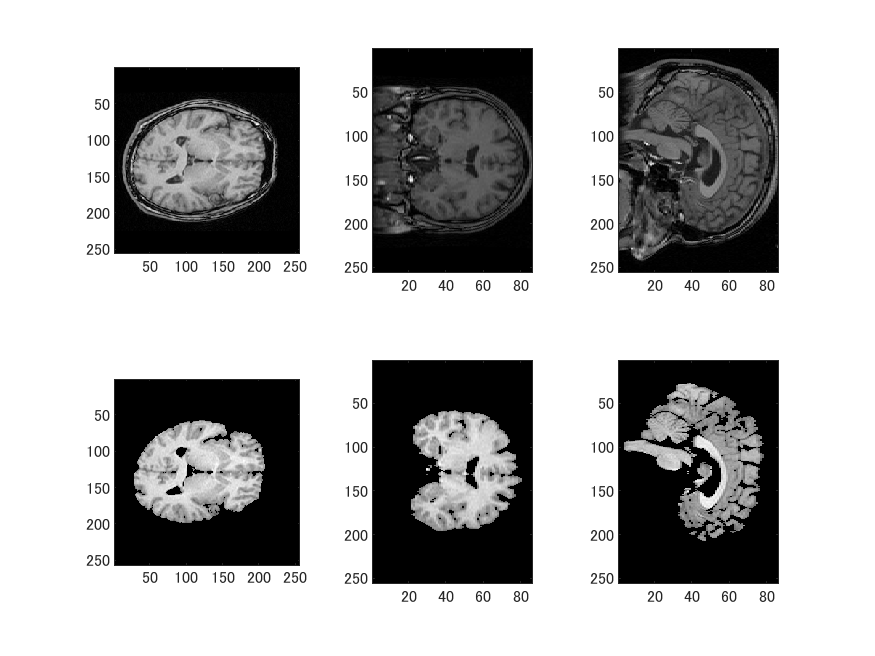
一旦NIfTIファイルとして保存後、読み込んだ後座標変換してMGZファイルで保存します。
hns_savefreesurferはこのHome Pageの最後に公開しています。hns_savefreesurferを使ってメッシュファイルをGIfTIからFreeSurferのSURFファイルに変換するとき、元の三次元配列の実際の大きさがが必要になります。
R=hns_savefreesurfer・・・でその情報を出力しています。
話の流れをわかりやすくするため、1つずつNIfTIで読んで、NIfTIで保存したものをMGZで保存して・・・となっていますが、hns_savefreesurfer自体は、2つのNIfTIファイルを同時にMGZファイルに変換する機能を有しています。
mkdir('~/名前/mri'); %予めフォルダを作成しておくこと
savename='〜/名前/mri/T1.nii';
niftiwrite(F1.data,savename,F1.info);
gzip(savename);
R=hns_savefreesurfer([savename,'.gz']);
delete(savename);
delete([savename,'.gz']);
savename='〜/名前/mri/brain.nii';
niftiwrite(F2.data,savename,F2.info);
gzip(savename);
hns_savefreesurfer([savename,'.gz']);
delete(savename);
delete([savename,'.gz']);
作成したT1.mgzとbrain.mgzです。
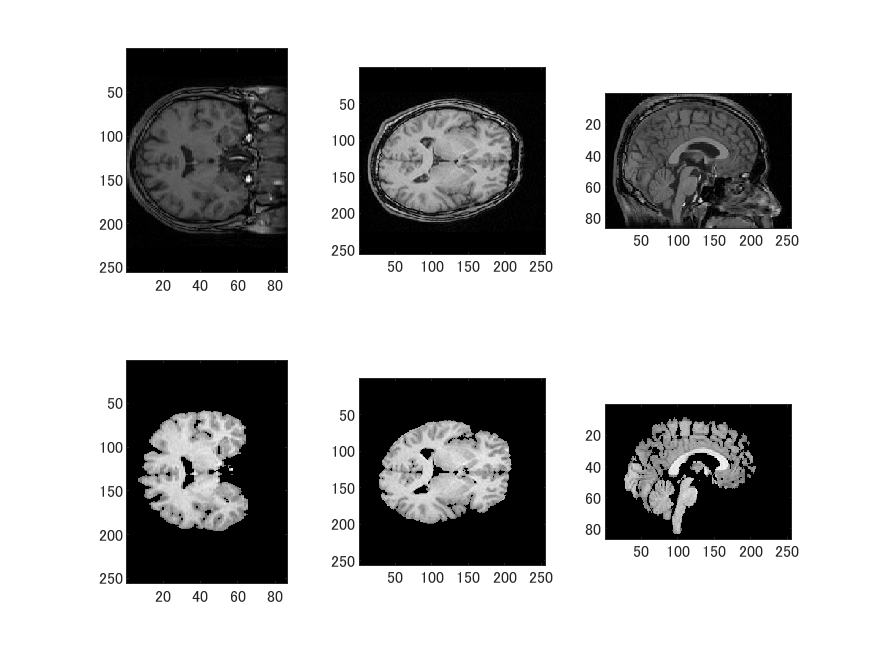
BrainSuiteで該当するファイルは以下の通りです。
〜/どっか/名前.nii.gzファイルと
〜/どっか/名前.mask.nii.gzファイルです。
BrainVISAと同じくbrain_名前.nii.gはマスクデータですので元の名前.nii.gzと掛け算してFreeSurferのbrain.mgz風に表示しています。
BrainVISAと違ってマスクデータのDatatypeがuint8になっていますので、int16にし、BitsPerPixelを8から16に書き直しています。
clear;
filename='〜/akira.nii.gz';
F1.data=niftiread(filename);
F1.info=niftiinfo(filename);
filename='〜/akira.mask.nii.gz';
F2.data=niftiread(filename);
F2.info=niftiinfo(filename);
F2.data(F2.data>0)=1;
F2.data(F2.data<1)=0;
F2.data=F1.data.*int16(F2.data);%
BrainVISAと違うところ
F2.info.Datatype='int16';%
BrainVISAと違うところ
F2.info.BitsPerPixel=16;%
BrainVISAと違うところ
figure;
m=F1.info.ImageSize/2;
p=F1.info.PixelDimensions;
for n=1:6
switch n
case
1;data=F1.data(:,:,m(3));ds=[p(1),p(2),p(3)];
case
2;data=F1.data(:,m(2),:);ds=[p(1),p(3),p(2)];
case
3;data=F1.data(m(1),:,:);ds=[p(2),p(3),p(1)];
case
4;data=F2.data(:,:,m(3));ds=[p(1),p(2),p(3)];
case
5;data=F2.data(:,m(2),:);ds=[p(1),p(3),p(2)];
case
6;data=F2.data(m(1),:,:);ds=[p(2),p(3),p(1)];
end
subplot(2,3,n);
imagesc(squeeze(data));
daspect(ds);
end
colormap(gray(256));
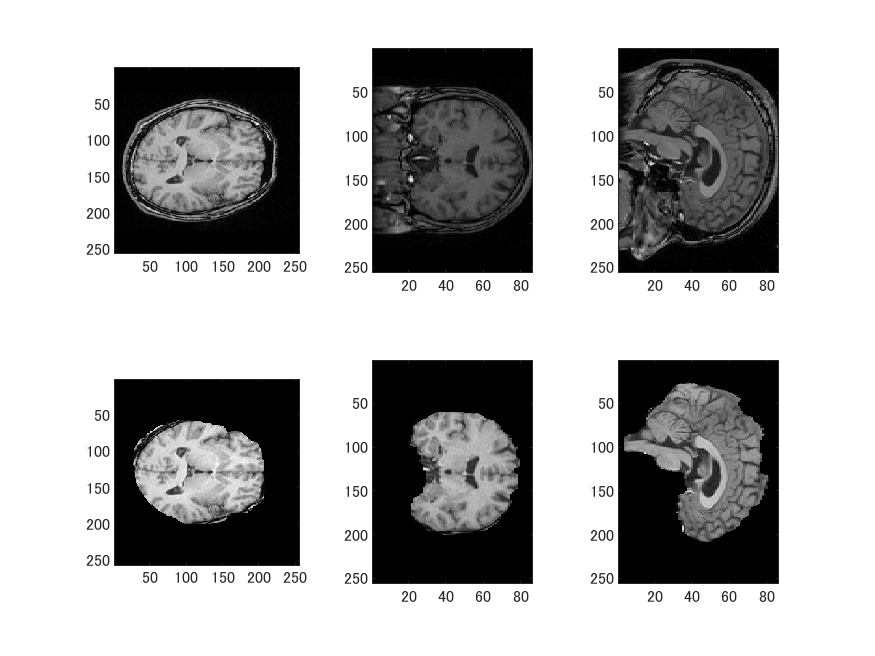
BrainVISAと同じようにNIfTIで保存してhns_savefreeserverでMGZに変換します。
mkdir('〜/名前//mri');%予めフォルダを作成しておくこと
savename='〜/名前/mri/T1.nii';
niftiwrite(F1.data,savename,F1.info);
gzip(savename);
R=hns_savefreesurfer([savename,'.gz']);
delete(savename);
delete([savename,'.gz']);
savename='〜/名前/mri/brain.nii';
niftiwrite(F2.data,savename,F2.info);
gzip(savename);
hns_savefreesurfer([savename,'.gz']);
delete(savename);
delete([savename,'.gz']);
作成したT1.mgzとbrain.mgzです。
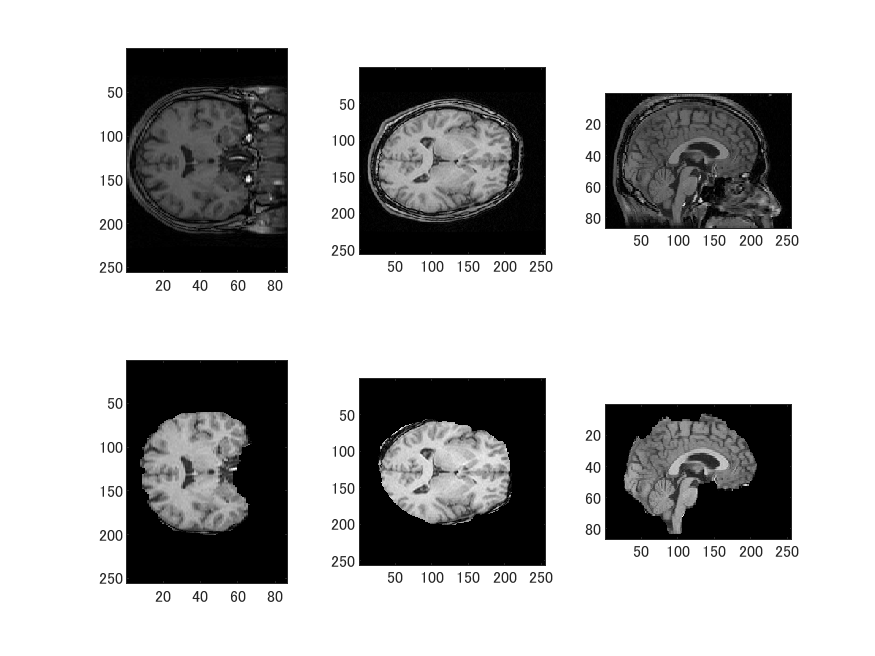
メッシュデータ
MNE-CはFreeSurferの/名前/surfフォルダの皮髄境界を示すlh.whiteとrh.whiteを使って電流源推定します。
表示はlh.whiteとrh.white、lh.inflatedとrh.inflated、lh.pialとrh.pialを使います。
また皮質表面と脳溝部分の色分けにwhiteとinflatedではlh.curvとrh.curvを、pialではlh.curv.pialとrh.curv.pialを使います。
なお、facesの番号は0番から始まるので、MATLABでは認識しないので、1を足して、1番から始まるようにしています。
clear;
filename='lh.white';
F1=hns_loadfreesurfer(filename);
filename='lh.curv';
C1=hns_loadfreesurfer(filename);
filename='rh.white';
F2=hns_loadfreesurfer(filename);
filename='rh.curv';
C2=hns_loadfreesurfer(filename);
figure;
subplot(131);
h1=trimesh(F1.faces'+1,F1.vertices(1,:)',F1.vertices(2,:)',F1.vertices(3,:)',C1.curv);
hold on;
h2=trimesh(F2.faces'+1,F2.vertices(1,:)',F2.vertices(2,:)',F2.vertices(3,:)',C2.curv);
set([h1;h2],'edgecolor','none','facecolor','interp');
set(gca,'clim',[-1,1]);
daspect([1,1,1]);
view([0,0]);
for n=2:3
ha=subplot(1,3,n);
copyobj([h1;h2],ha);
daspect([1,1,1]);
set(gca,'clim',[-1,1]);
if n==2
view([0,90]);
elseif n==3
view([-90,0]);
end
end
map=-gray(2)*0.5+0.75;
colormap(map);
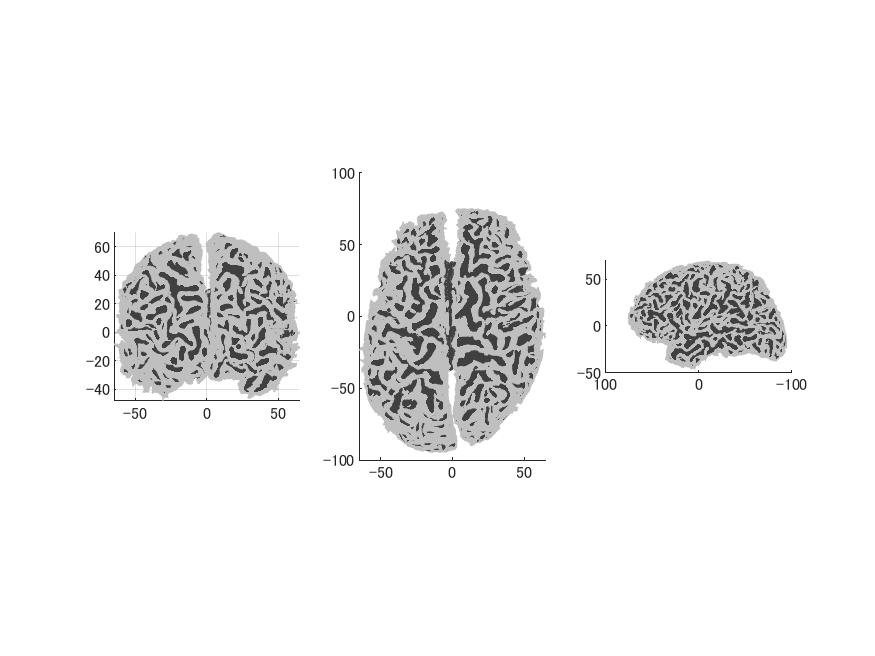
BrainVISAでは
lh/rh.whiteに相当するのが〜/名前/t1mri/default_acquisition/default_analysis/segmentation/meshの名前_Lwhite.giiと名前_Rwhite.gii
lh/rh.inflatedに相当するのが上記フォルダ内の名前_Lwhite_inflated.giiと名前_Rwhite_inflated.gii
lh/rh.curvに相当するのが〜/名前/t1mri/default_acquisition/default_analysis/segmentationの名前_Lwhite_curv.giiと名前_Rwhite_curv.giiです。
拡張子giiはGIfTIファイルを意味します。
MATLABでGIfTIファイルを読み書きするにはA MATLAB GIfTI libraryを利用します。
ポリゴンメッシュ頂点の曲率の正負はBrainVISAとFreeSurferでは逆になっていますのでマイナスをつけています。
addpath('/どっか/gifti-1.6');% A MATLAB
GIfTI libraryにPATHを通しておくこと
filename='/どっか/名前/t1mri/default_acquisition/default_analysis/segmentation/mesh/akira_Lwhite.gii';
F1=gifti(filename);
filename='/どっか/名前/t1mri/default_acquisition/default_analysis/segmentation/akira_Lwhite_curv.gii';
C1=gifti(filename);
filename='/どっか/名前/t1mri/default_acquisition/default_analysis/segmentation/mesh/akira_Rwhite.gii';
F2=gifti(filename);
filename='/どっか/名前/t1mri/default_acquisition/default_analysis/segmentation/akira_Rwhite_curv.gii';
C2=gifti(filename);
figure;
subplot(1,3,1);
h1=trimesh(F1.faces,F1.vertices(:,1),F1.vertices(:,2),F1.vertices(:,3),-C1.cdata);
hold on;
h2=trimesh(F2.faces,F2.vertices(:,1),F2.vertices(:,2),F2.vertices(:,3),-C2.cdata);
set([h1;h2],'edgecolor','none','facecolor','interp');
daspect([1,1,1]);
set(gca,'clim',[-1,1]);
view([0,0]);
for n=2:3
ha=subplot(1,3,n);
copyobj([h1;h2],ha);
daspect([1,1,1]);
set(gca,'clim',[-1,1]);
if n==2
view([0,90]);
elseif n==3
view([-90,0]);
end
end
map=-gray(2)*0.5+0.75;
colormap(map);
rotate3d on;
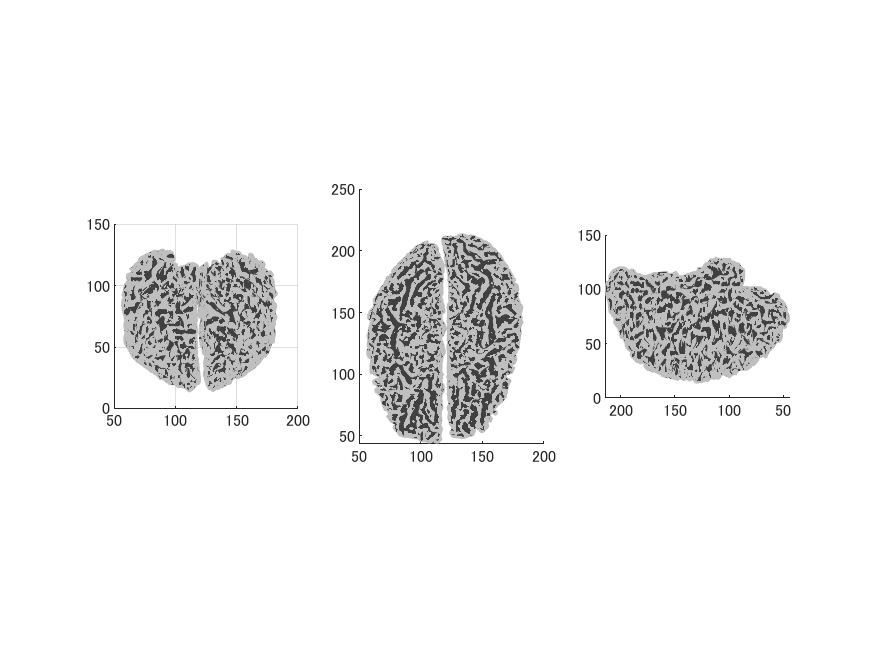
座標変換してFreeSurferのSURF形式で保存します。lh/rh.inflatedやlh/rh.pical、lh/rh.curv.pialも以下の方法に準拠した方法でSURF形式で保存できます。
% 【1段階】FreeSurfer用のフォルダ作成
mkdir('/どっかそのへん/名前/mri');
mkdir('/どっかそのへん/名前/surf');
% 【2段階】名前.nii.gzとvoronoi_名前.nii.gzからT1.mgzとbrain.mgzを作成.
filename='/どっか/名前/t1mri/default_acquisition/akira.nii.gz';
filename1='/どっかそのへん/名前/mri/T1.nii.gz';
copyfile(filename,filename1);
filename='/どっか/名前/t1mri/default_acquisition/default_analysis/segmentation/voronoi_a.nii.gz';
filename2='/どっかそのへん/名前/mri/brain.nii.gz';
copyfile(filename,filename2);
dsize=hns_savefreesurfer(filename1,filename2);
% 【3段階】名前_Lwhite.giiと名前_Rwhite.giiからlh.whiteとrh.whiteを作成
filename='/どっか/名前/t1mri/default_acquisition/default_analysis/segmentation/mesh/akira_Lwhite.gii';
filename1='/どっかそのへん/名前/surf/akira_Lwhite.gii';
copyfile(filename,filename1);
filename='/どっか/名前/t1mri/default_acquisition/default_analysis/segmentation/mesh/akira_Rwhite.gii';
filename2='/どっかそのへん/名前/surf/akira_Rwhite.gii';
copyfile(filename,filename2);
dsurf1=hns_savefreesurfer(filename1,dsize);
dsurf2=hns_savefreesurfer(filename2,dsize);
% 【4段階】名前_Lwhite_curv.giiと名前_Rwhite_curv.giiからlh.curvとrh.curvを作成
filename='/どっか/名前/t1mri/default_acquisition/default_analysis/segmentation/akira_Lwhite_curv.gii';
filename3='/どっかそのへん/名前/surf/akira_Lwhite_curv.gii';
copyfile(filename,filename3);
filename='/どっか/名前/t1mri/default_acquisition/default_analysis/segmentation/akira_Rwhite_curv.gii';
filename4='/どっかそのへん/名前/surf/akira_Rwhite_curv.gii';
copyfile(filename,filename4);
hns_savefreesurfer(filename3,dsurf1);
hns_savefreesurfer(filename4,dsurf2);
作成したlh/rh.whiteとlh/rh.curvをhns_loadfreesurferで見てみました。
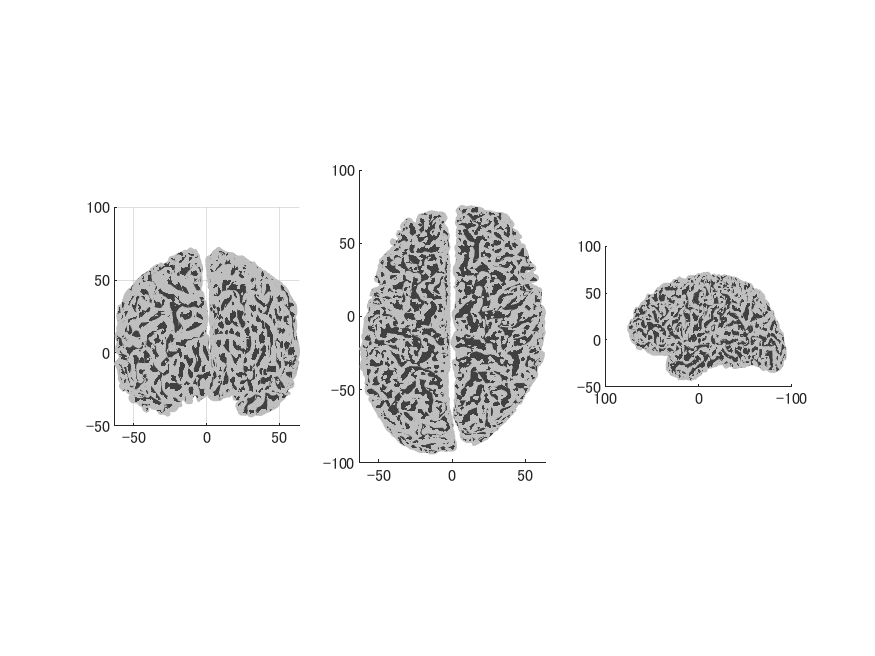
BrainSuiteでは
lh/rh.whiteに相当するのが〜/どっか/名前.left.inner.cortex.dfsと〜/どっか/名前.right.inner.cortex.dfsです。
BrainSuite自体には脳を膨らませたり、メッシュ頂点の曲率を計算する機能は見当たりません。
皮髄境界メッシュである名前.left/right.inner.cortex.dfsだけで電流源推定することは可能ですが、通常MNE-Cで用いられる濃厚部分が灰色の膨張皮質メッシュ画像を作成することはできません。
filename='/どっか/akira.left.inner.cortex.dfs';
F1=hns_loaddfs(filename);
filename='/どっか/akira.right.inner.cortex.dfs';
F2=hns_loaddfs(filename);
figure;
subplot(1,3,1);
h1=patch('vertices',F1.vertices','faces',F1.faces');
hold on;
h2=patch('vertices',F2.vertices','faces',F2.faces');
set([h1;h2],'edgecolor','none','facecolor',[1,1,1]*0.5);
h3=light('position',[0,0,1]);
h4=light('position',[0,0,-1]);
daspect([1,1,1]);
set(gca,'clim',[-1,1]);
view([0,0]);
for n=2:3
ha=subplot(1,3,n);
copyobj([h1;h2;h3;h4],ha);
daspect([1,1,1]);
if n==2
view([0,90]);
elseif n==3
view([-90,0]);
end
end
同じ名前.nii.gzファイルから作成した皮髄境界メッシュですが、BrainVISAと違って、左右・上下・前後が逆になっています。
座標変換してBrainVISAのファイル名に準拠したファイル名で保存します。フォルダの位置は適当です。
addpath('/どっか保存したとこ/gifti-1.6');% A
MATLAB GIfTI libraryにPATHを通す
filename='/どっか/akira.nii.gz';
info=niftiinfo(filename);
m=info.ImageSize'.*info.PixelDimensions'/2;
M=[eye(4,3),[m;1]]*diag([-1,-1,-1,1])*[eye(4,3),[-m;1]];
for n=1:2
if n==1
filename='/どっか/akira.left.inner.cortex.dfs';
savename='/どっか/akira_Lwhite.gii';
else
filename='/どっか/akira.right.inner.cortex.dfs';
savename='/どっか/akira_Rwhite.gii';
end
F=hns_loaddfs(filename);
g=[];
g.faces=F.faces';
P=F.vertices;
P(4,:)=1;
P=M*P;
g.vertices=P(1:3,:)';
g.mat=eye(4);
g=gifti(g);
save(g,savename,'Base64Binary');
end
BrainVISAのMorphologistのInflate cortical Surfaceを選択します。
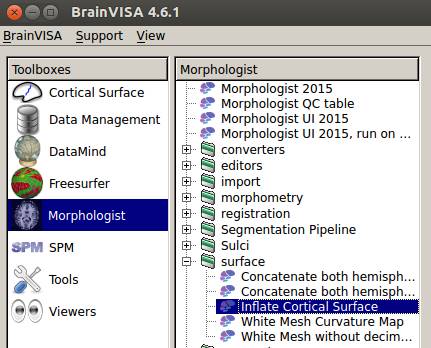
output_meshとcurvature_textureのファイル名はBrainVISAの名前になるよう手入力します。
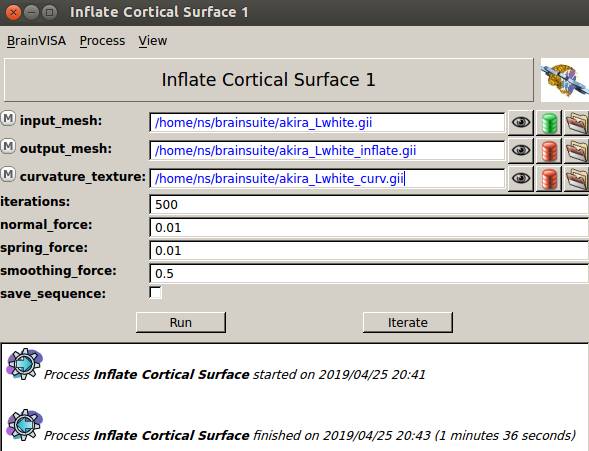
右半球。
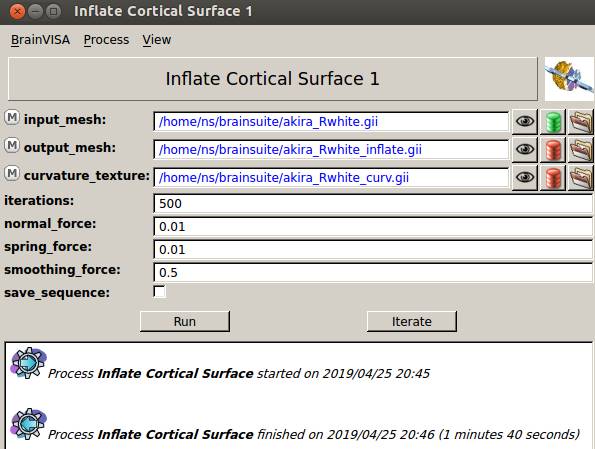
BrainVISAのGIfTIファイルに変換後のファイルです。
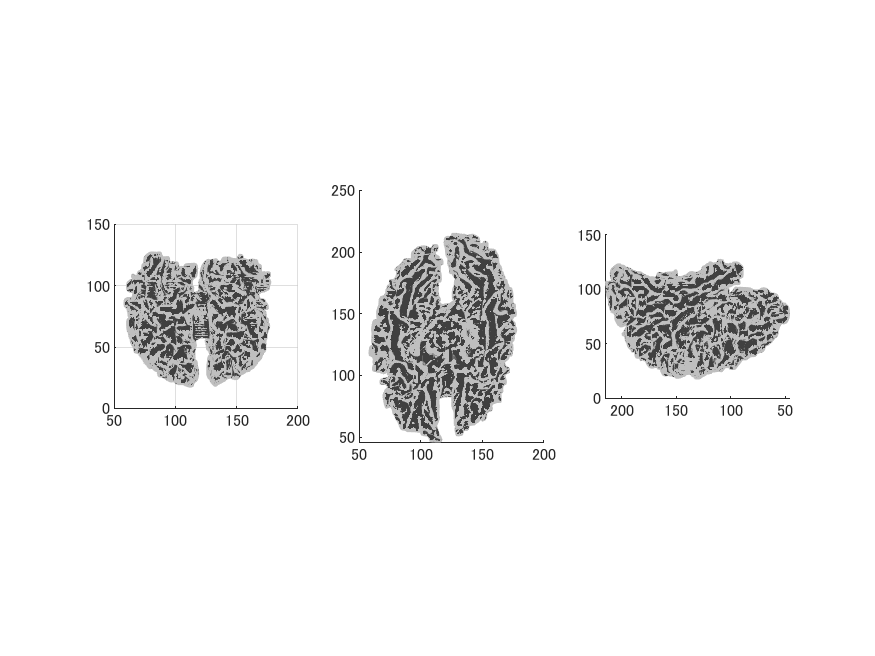
あとは上述のBrainVISAのファイルをFreeSurferのファイルに変換する方法を実行します。
% 【1段階】FreeSurfer用のフォルダ作成
mkdir('/どっかそのへん/名前/mri');
mkdir('/どっかそのへん/名前/surf');
% 【2段階】名前.nii.gzとvoronoi_名前.nii.gzからT1.mgzとbrain.mgzを作成.
filename='/どっか/akira.nii.gz';
filename1='/どっかそのへん/名前/mri/T1.nii.gz';
copyfile(filename,filename1);
filename='/どっか/akira.mask.nii.gz';
filename2='/どっかそのへん/名前/mri/brain.nii.gz';
copyfile(filename,filename2);
dsize=hns_savefreesurfer(filename1,filename2);
% 【3段階】名前_Lwhite.giiと名前_Rwhite.giiからlh.whiteとrh.whiteを作成
filename='/どっか/akira_Lwhite.gii';
filename1='/どっかそのへん/名前/surf/akira_Lwhite.gii';
movefile(filename,filename1);
filename='/どっか/akira_Rwhite.gii';
filename2='/どっかそのへん/名前/surf/akira_Rwhite.gii';
movefile(filename,filename2);
dsurf1=hns_savefreesurfer(filename1,dsize);
dsurf2=hns_savefreesurfer(filename2,dsize);
% 【4段階】名前_Lwhite_curv.giiと名前_Rwhite_curv.giiからlh.curvとrh.curvを作成
filename='/どっか/akira_Lwhite_curv.gii';
filename3='/どっかそのへん/名前/akira_Lwhite_curv.gii';
movefile(filename,filename3);
filename='/どっか/akira_Rwhite_curv.gii';
filename4='/どっかそのへん/名前/akira_Rwhite_curv.gii';
movefile(filename,filename4);
hns_savefreesurfer(filename3,dsurf1);
hns_savefreesurfer(filename4,dsurf2);
FreeSurferの用に変換したlh/rh.whiteとlh/rh.curvをhns_loadfreesurferで見てみました。
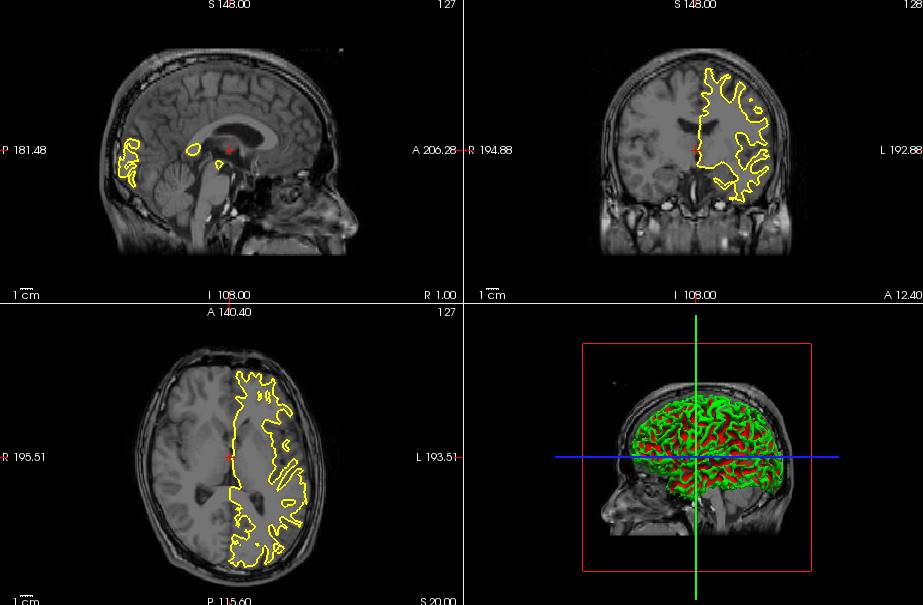
FreeSurferのT1.mgzとlh.whiteをfreeviewで見てみました。
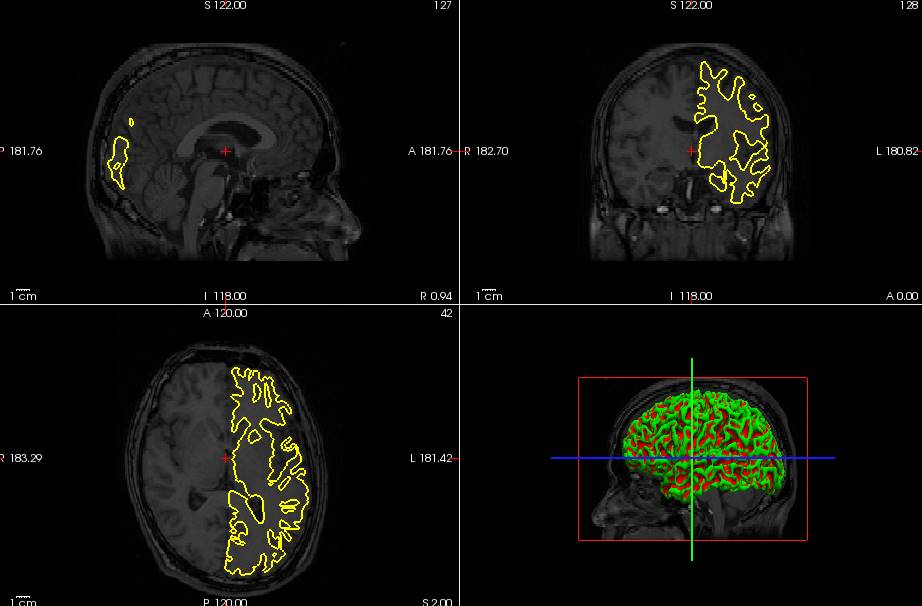
BrainVISAのT1.mgzとlh.whieをfreeviewで見てみました。
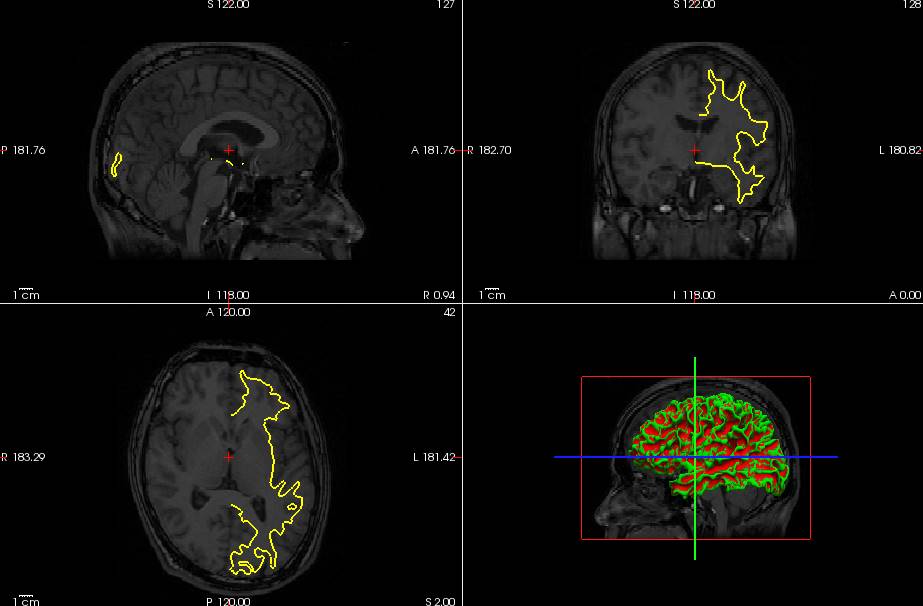
BrainSuiteのT1.mgzとlh.whiteを見てみました。閉曲面になっていません。たぶん、このためmne_analyzeでは開くことができません。
FreeSurferのlh/rh.whiteをmne_analyzeを見たところです。
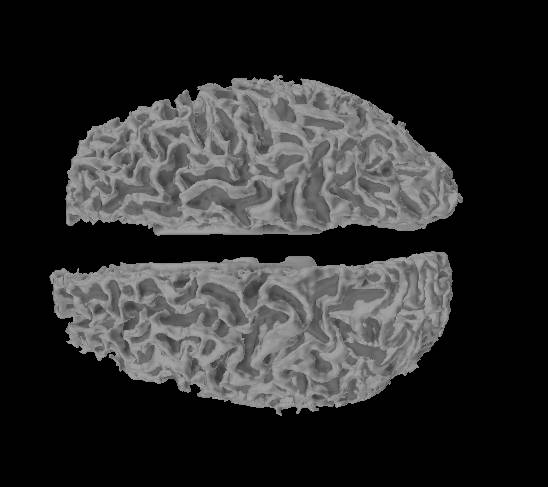
BrainVISAのlh/rh.whiteをmne_analyzeを見たところです。
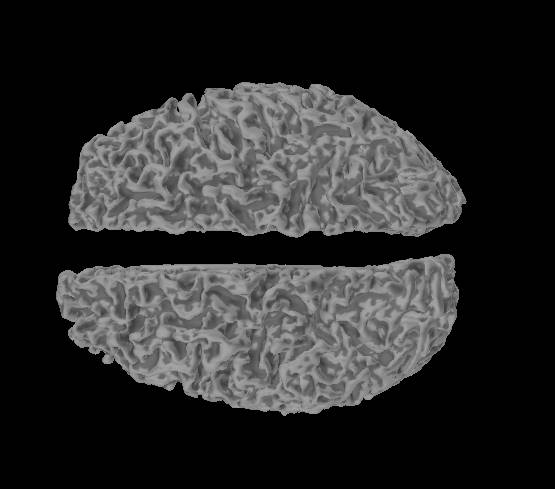
BrainSuiteのlh/rh.whiteをmne_analyzeで見ようとしたら・・・ダメでした。

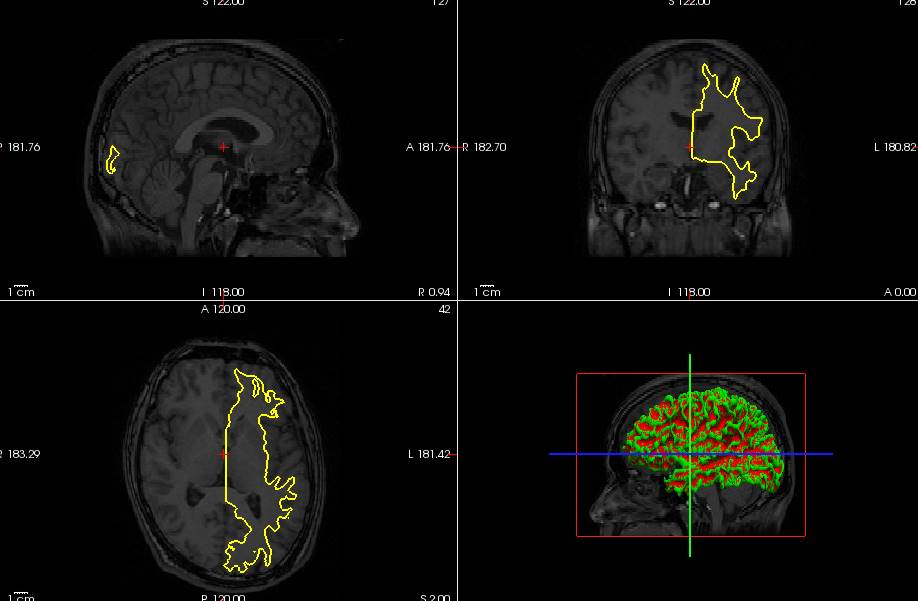
BrainSuiteの皮髄境界抽出画像と左右半球ラベル画像から皮髄境界メッシュを作成
BrainSuiteは左右が連結した状態で皮髄境界メッシュを作成し、左右をぶった切って、左右半球の皮髄境界メッシュファイル、DFSを作成するのがですが、ぶった切ったところは閉じていません。閉曲面になってないので、元画像から左右半球別々に新たに皮髄境界メッシュを作成してみます。
clear;
addpath('/どっか/MATLAB\gifti-1.6');
filename='/そのへん/akira.cortex.dewisp.mask.nii.gz';
F.data=niftiread(filename);
F.info=niftiinfo(filename);
filename='/そのへん/akira.hemi.label.nii.gz';
G.data=niftiread(filename);
G.info=niftiinfo(filename);
figure;
m2=F.info.ImageSize/2;
for n=1:6
subplot(3,3,n);
switch n
case
1;D=F.data(:,:,m2(3));
case
2;D=F.data(:,m2(2),:);
case
3;D=F.data(m2(1),:,:);
case
4;D=G.data(:,:,m2(3));
case
5;D=G.data(:,m2(2),:);
case
6;D=G.data(m2(1),:,:);
end
imagesc(squeeze(D));
colorbar;
end
F.data(F.data>0)=1;
for n=1:2:17
G.data(G.data==n)=1;
end
for n=2:2:16
G.data(G.data==n)=2;
end
F.data=F.data.*G.data;
for n=1:3
subplot(3,3,n+6);
switch n
case
1;D=F.data(:,:,m2(3));
case
2;D=F.data(:,m2(2),:);
case
3;D=F.data(m2(1),:,:);
end
imagesc(squeeze(D));
colorbar;
end
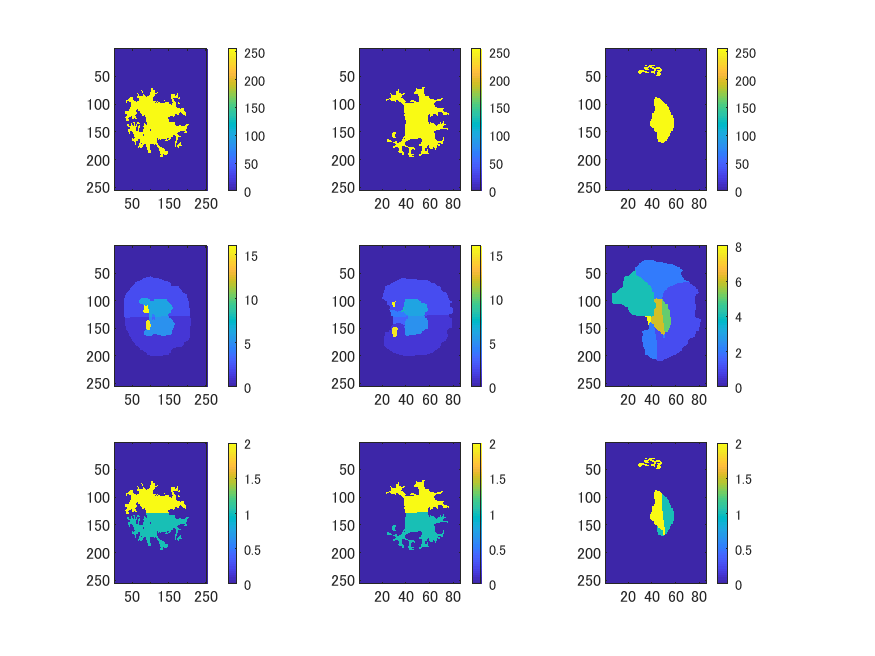
座標をBrainVISAに合わせ、元のNIfTI画像を回転させ、MATLABのsmooth3関数で平滑化し、isosurface関数で曲面パッチを作成し、GIfTIファイル化します。
for n=1:2
if n==1
savename='/そのへん/akira_Rwhite.gii';
else
savename='/そのへん/akira_Lwhite.gii';
end
D=F.data;
D=D(:,:,end:-1:1);
for
nn=1:size(D,3)
D(:,:,nn)=rot90(squeeze(D(:,:,nn))',2);
end
D(D~=n)=0;
D(D>0)=1;
D=double(D);
pix=F.info.ImageSize.*F.info.PixelDimensions;
x=linspace(0,pix(1),F.info.ImageSize(1));
y=linspace(0,pix(2),F.info.ImageSize(2));
z=linspace(0,pix(3),F.info.ImageSize(3));
[X,Y,Z]=meshgrid(x,y,z);
fv=isosurface(X,Y,Z,smooth3(D),0.5);
fv.faces=fv.faces(:,[1,3,2]);
fv.mat=eye(4);
g=gifti(fv);
save(g,savename,'Base64Binary');
end
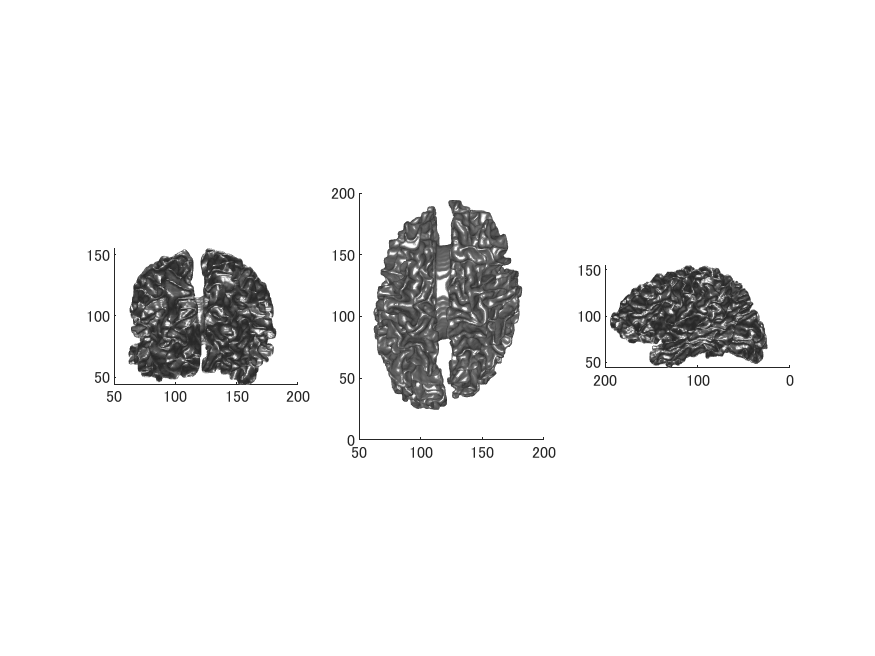
BrainVISAのinflate cortical surface機能を用いて、Lwhite_inflate.gii、Lwhite_curv.gii、Rwhite_inflate.gii、Rwhite_curv.giiを作成します。
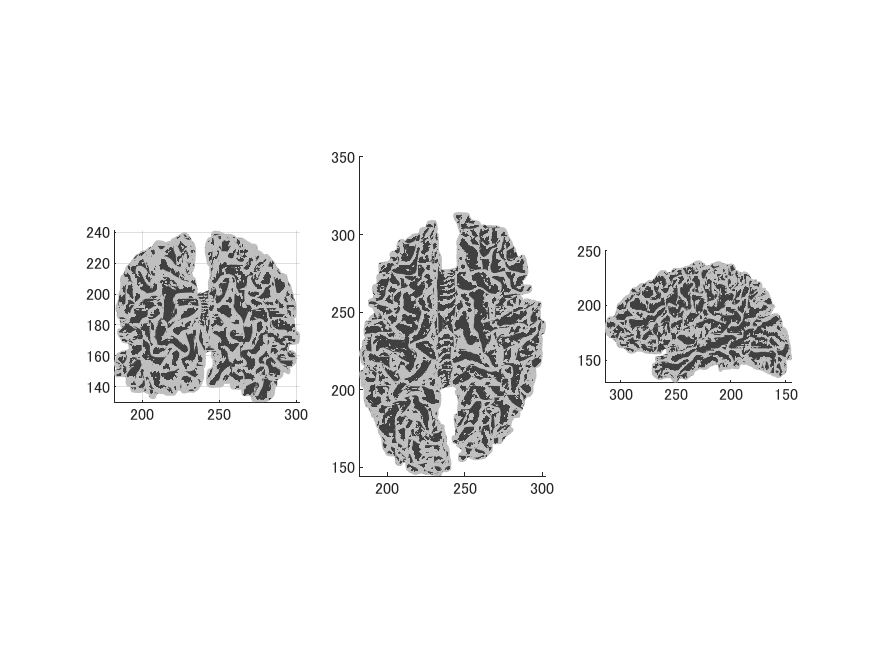
以下のような画像になります。座標系はBrainVISAのものになっています。
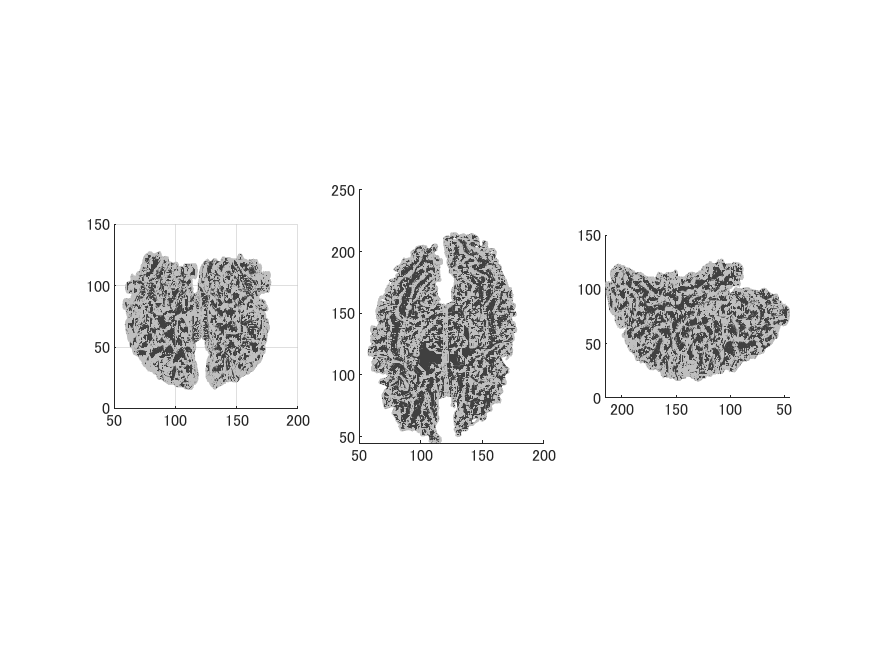
BrainVISA→FreeSurfer変換でmriフォルダにT1.mgz、brain.mgz、surfフォルダにlh/rh.white、lh/rh.inflated、lh/rh.curvを作成します。
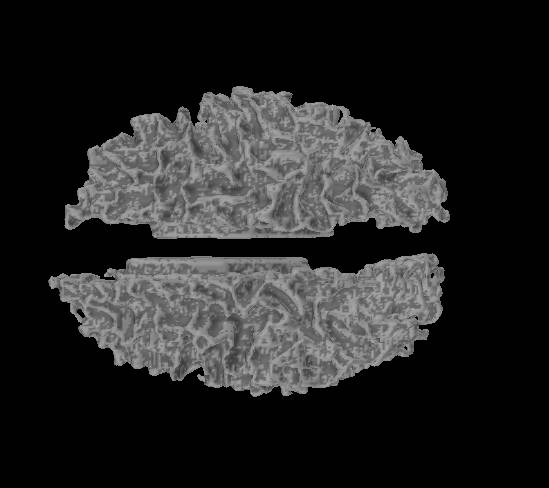
freeviewで見たところです。閉曲面になっています。
mne_analyzeで見てみました。
めでたしめでたしです。やっぱり閉曲面でないとmne_analyzeは表示できないようです。
電流源推定(SEF N20m)の比較
以下を$MNE_ROOTにmne_preparebv2.cshという名前で保存して実行しました。
mne_setup_mri
mne_setup_source_space #--surface whte $ default
mne_watershed_bem --atlas # make 4-layer trigon mesh
cd $SUBJECTS_DIR/$SUBJECT/bem/watershed
mne_convert_surface --surf ${SUBJECT}_inner_skull_surface
--triout ../inner_skull.tri
cd $SUBJECTS_DIR
mne_setup_forward_model --homog --noswap --ico4
座標合わせ後に以下を実行します。なお、別途、分散行列SEF-cov.fifを用意しておきます。
mne_do_forward_solution –-meas SEF.fif –megonly
mne_do_inverse_operator --fwd SEF-7-fwd.fif
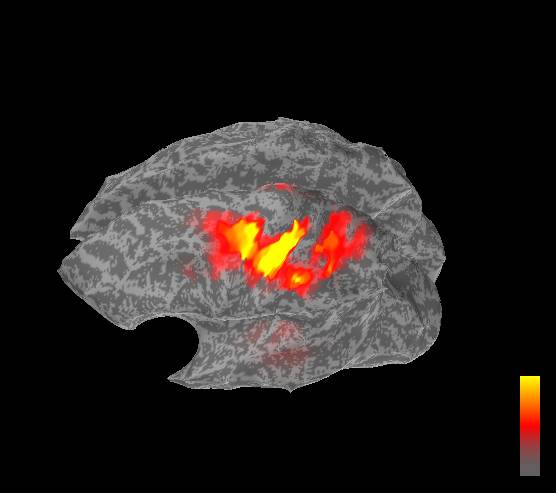
SEFの22msecの電流源推定結果を示します。
FreeSurferです。
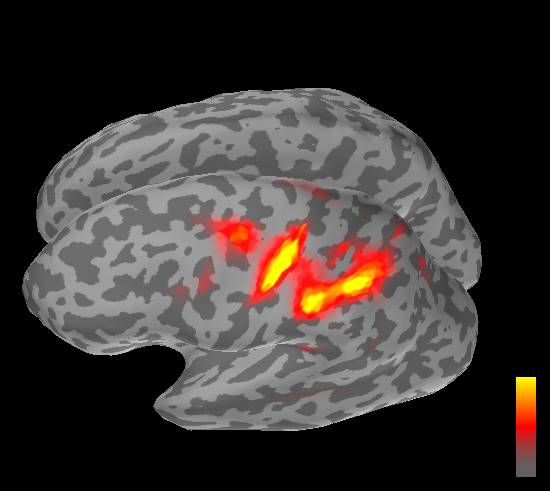
BrainVISAです。
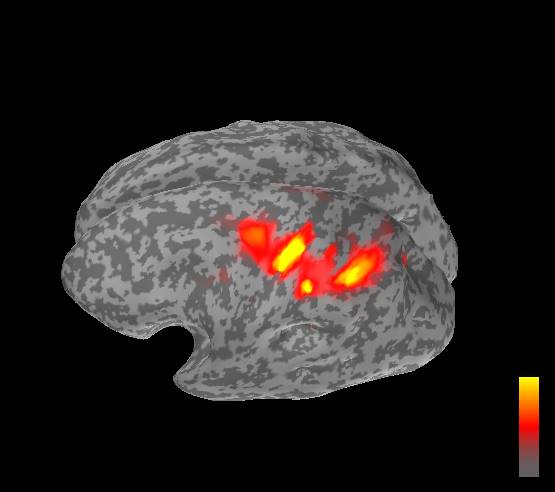
BrainSuiteです。
まとめ
visa_convert2.mという関数Mファイルを作成しました。
BrainVISAの場合は、名前.nii.gzのファイルを指定すればmriとsurfというフォルダを作ってその中にMNE-Cに必要なファイルを吐き出します。
visa_converts2
だけでOKです。
BrainSuiteの場合は
visa_converts2
とやると、brainvisaというフォルダに名前_Lwhite.giiとRwhite.giiというファイルを吐き出します。
これをBrainVISAのinflate cortical surface機能で新たにLwhite_inflated.gii、Rwhite_inflated.gii、Lwhite_curv.gii、Rwhite_curv.giiという4つのファイルを作成し、brainvisaというフォルダに入れ、もう1回
visa_converts2
とやると、mriとsurfというフォルダを作ってその中にMNE-Cに必要なファイルを吐き出します。
作成した関数Mファイル
以下のようなファイルを作成しました。
FreeSurferのMGZやSURFファイルを読む関数Mファイル
function F=hns_loadfreesurfer(filename)
F=[];
if exist(filename,'file')==0;return;end
[~,~,ext]=fileparts(filename);
if ismember(ext,{'.mgz'}) % 基本T1.mgzとbrain.mgz
F=readmgz(filename);
elseif ismember(ext,{'.inflated','.pial','.white'})% lh/rh
pial lh/rh inflated lh/rh white のみ
F=readsurf(filename);
elseif ismember(ext,{'.curv'})
F=readcurv(filename);
end
return;
%%
function F=readcurv(filename)
F=[];
fid=fopen(filename,'rb');
if fid==0;return;end
B=fread(fid,'*uint8');
fclose(fid);
if
all(B(1:3)==uint8([255;255;255]))~=1;return;end
k=3+(1:12);
x=swapbytes(typecast(B(k),'int32'));
F.nvertices=x(1);
F.nfaces=x(2);
F.n_=x(3);
k=3+12+(1:(F.nvertices*4));
F.curv=swapbytes(typecast(B(k),'single'));
return;
%%
function F=readmgz(filename)
F=[];
loadname=gunzip(filename);% T.mgzだとT1となる
loadname=loadname{1};
fid=fopen(loadname,'rb');
if fid==0
delete(loadname);
return
end
B=fread(fid,'*uint8');
fclose(fid);
delete(loadname);
try
k=1:28;
x=swapbytes(typecast(B(k),'int32'));
F.version=x(1);
F.width=x(2);
F.height=x(3);
F.depth=x(4);
F.nframes=x(5);
F.type=x(6);
switch F.type
case 0;fmt='uint8';fbyte=1;
case 1;fmt='int32';fbyte=4;
case 2;fmt='single';fbyte=4;
case 4;fmt='int16';fbyte=2;
end
F.dof=x(7);% degree of freedom
k=29:30;
F.goodRASFlag=swapbytes(typecast(B(k),'int16'));
k=31:90;
x=swapbytes(typecast(B(k),'single'));
F.spacingx=x(1);
F.spacingy=x(2);
F.spacingz=x(3);
F.xr=x(4);%RAS right anterior superior
F.xa=x(5);
F.xs=x(6);
F.yr=x(7);
F.ya=x(8);
F.ys=x(9);
F.zr=x(10);
F.za=x(11);
F.zs=x(12);
F.cr=x(13);
F.ca=x(14);
F.cs=x(15);
k=284+(1:F.width*F.height*F.depth*fbyte);
F.data=swapbytes(typecast(B(k),fmt));
F.data=reshape(F.data,[F.width,F.height,F.depth]);
catch
return;
end
return;
%%
function F=readsurf(filename)
F=[];
if strfind(filename,'area.pial')>0;return;end
if strfind(filename,'curv.pial')>0
F=readcurv(filename);
return;
end
fid=fopen(filename,'rb');
if fid==0;return;end
B=fread(fid,'*uint8');
fclose(fid);
if
all(B(1:3)==uint8([255;255;254]))~=1;return;end
for kk=4:(length(B)-1)
if B(kk)==10
if B(kk+1)==10
break;
end
end
end
kk=kk+1;
k=kk+(1:8);
x=swapbytes(typecast(B(k),'int32'));
F.nvertices=x(1);
F.nfaces=x(2);
k=kk+8+(1:(F.nvertices*4*3));
F.vertices=swapbytes(typecast(B(k),'single'));
F.vertices=reshape(F.vertices,[3,F.nvertices]);
k=kk+8+F.nvertices*4*3+(1:(F.nfaces*4*3));
F.faces=swapbytes(typecast(B(k),'int32'));
F.faces=reshape(F.faces,[3,F.nfaces]);
return;
BrainVISAやBrainSuiteのファイルをFreeSurferのMGZファイルやSURFファイルに変換する関数Mファイル
function
R=hns_savefreesurfer(filename,filename2)
R=[];
if nargin==0;return;end
if nargin==1;filename2='none';end
if strfind(filename,'.nii.gz')>0
R=writemgz(filename,filename2);
elseif strfind(filename,'.gii')>0
% filename2は3次元配列の大きさや頂点数・面数
R=writesurf(filename,filename2);
end
return;
%%
function R=writemgz(filename,filename2)
R=[];
info=niftiinfo(filename);
D=niftiread(filename);
switch class(D)
case 'uint8';ftype=0;
case 'int32';ftype=1;
case 'single';ftype=2;
case 'int16';ftype=4;
end
D2=repmat(D(1,1,1),[info.ImageSize(1),info.ImageSize(3),info.ImageSize(2)]);
for y=1:info.ImageSize(2)
D2(:,:,y)=squeeze(D(:,y,:));
end
D2=D2(:,end:-1:1,:);
savename=[filename(1:end-6),'mgh'];
x=[1,info.ImageSize([1,3,2]),1,ftype,0];
fid=fopen(savename,'wb');
fwrite(fid,x,'int32','ieee-be');%
version, size(x,z,y),nframes,type,dof
fwrite(fid,1,'int16','ieee-be');% good
RAS Flag
fwrite(fid,info.PixelDimensions([1,3,2]),'single','ieee-be');
M=[-1,
0, 0,0;
0, 0, 1,0;
0,-1, 0,0];
fwrite(fid,M(:),'single','ieee-be');
fwrite(fid,zeros(1,284-90),'uint8');
fwrite(fid,D2(:),class(D),'ieee-be');
fclose(fid);
savename2=gzip(savename);
movefile(savename2{1},[savename(1:end-1),'z']);
delete(savename);
R=info.ImageSize.*info.PixelDimensions;
if strcmp(filename2,'none');return;end
info1=niftiinfo(filename2);
if
all(info.ImageSize==info1.ImageSize)~=1;return;end
if
all(info.PixelDimensions==info1.PixelDimensions)~=1;return;end
D1=niftiread(filename2);
D(D1<1)=0;
for y=1:info.ImageSize(2)
D2(:,:,y)=squeeze(D(:,y,:));
end
D2=D2(:,end:-1:1,:);
savename=[filename2(1:end-6),'mgh'];
x=[1,info.ImageSize([1,3,2]),1,ftype,0];
fid=fopen(savename,'wb');
fwrite(fid,x,'int32','ieee-be');%
version, size(x,z,y),nframes,type,dof
fwrite(fid,1,'int16','ieee-be');% good
RAS Flag
fwrite(fid,info.PixelDimensions([1,3,2]),'single','ieee-be');
M=[-1,
0, 0,0;
0, 0, 1,0;
0,-1, 0,0];
fwrite(fid,M(:),'single','ieee-be');
fwrite(fid,zeros(1,284-90),'uint8');
fwrite(fid,D2(:),class(D),'ieee-be');
fclose(fid);
savename2=gzip(savename);
movefile(savename2{1},[savename(1:end-1),'z']);
delete(savename);
return
%%
function R=writesurf(filename,data)
R=[];
if ischar(data);return;end
if ~all(size(data)==[1,3]);return;end
F=gifti(filename);
pathname=fileparts(filename);
if contains(filename,'Lhemi.gii')
savename=[pathname,'\lh.pial'];
dsize=data;
elseif contains(filename,'Rhemi.gii')
savename=[pathname,'\rh.pial'];
dsize=data;
elseif contains(filename,'Lwhite.gii')
savename=[pathname,'\lh.white'];
dsize=data;
elseif contains(filename,'Rwhite.gii')
savename=[pathname,'\rh.white'];
dsize=data;
elseif contains(filename,'Lwhite_inflated.gii')
savename=[pathname,'\lh.inflated'];
dsize=data;
elseif contains(filename,'Rwhite_inflated.gii')
savename=[pathname,'\rh.inflated'];
dsize=data;
elseif contains(filename,'Lwhite_curv.gii')
savename=[pathname,'\lh.curv'];
dsurf=data;
elseif contains(filename,'Rwhite_curv.gii')
savename=[pathname,'\rh.curv'];
dsurf=data;
else
return;
end
if exist('dsize','var')
if ~isfield(F,'vertices');return;end
if ~isfield(F,'faces');return;end
X0=[eye(3,4);-dsize(1)/2,-dsize(2)/2,-dsize(3)/2,1];
X1=diag([-1,-1,-1,0]);
X1=X0*X1*[1,0,0,0;0,1,0,0;0,0,1,0;0,0,0,1];
vertices=F.vertices;
vertices(:,4)=1;
vertices=vertices*X1;
vertices=vertices(:,[1,2,3]);
faces=F.faces(:,[1,3,2])-1;
fid=fopen(savename,'wb');
fwrite(fid,[255,255,254],'uint8');
str=['created by matlab on ',datestr(now,'ddd mmm dd HH:MM:SS yyyy')];
fwrite(fid,str,'uint8');
fwrite(fid,[10,10],'uint8');
fwrite(fid,[size(vertices,1),size(faces,1)],'int32','ieee-be');
fwrite(fid,vertices','single','ieee-be');
fwrite(fid,faces','int32','ieee-be');
fclose(fid);
R=[size(vertices,1),size(faces,1),1];
elseif exist('dsurf','var')
if ~isfield(F,'cdata');return;end
fid=fopen(savename,'wb');
fwrite(fid,[255,255,255],'uint8');
fwrite(fid,dsurf,'int32','ieee-be');%nvertices,nfaces,1
fwrite(fid,-F.cdata,'single','ieee-be');
fclose(fid);
end
return;
BrainSuiteのDFSファイルを読み込む関数Mファイル
function R=hns_loaddfs(loadname)
R=[];
try
fid=fopen(loadname,'rb');
B=fread(fid,'*uint8');
fclose(fid);
catch
return
end
try
ptrimesh=typecast(B(13:16),'int32');
ntri=typecast(B(25:28),'int32');
nvertex=typecast(B(29:32),'int32');
pvertex=ptrimesh+3*ntri*4;
pnormal=typecast(B(33:36),'int32');
puv=typecast(B(37:40),'int32');
prgb=typecast(B(41:44),'int32');
plabel=typecast(B(45:48),'int32');
pattr=typecast(B(49:52),'int32');
catch
return
end
try
address=ptrimesh+(1:(3*ntri*4));
x=typecast(B(address),'int32');
x=reshape(x,[3,ntri]);
R.faces=x+1;
address=pvertex+(1:(3*nvertex*4));
x=typecast(B(address),'single');
R.vertices=reshape(x,[3,nvertex]);
catch
return
end
if pnormal>0
try
address=pnormal+(1:(3*nvertex*4));
x=typecast(B(address),'single');
R.normals=reshape(x,[3,nvertex]);
catch
return
end
end
if puv>0
try
address=puv+(1:(2*nvertex*4));
x=typecast(B(address),'single');
R.uv=reshape(x,[2,nvertex]);
catch
return
end
end
if prgb>0
try
address=prgb+(1:(3*nvertex*4));
x=typecast(B(address),'single');
R.rgb=reshape(x,[3,nvertex]);
catch
return
end
end
if plabel>0
try
address=plabel+(1:(nvertex*2));
x=typecast(B(address),'int16');
R.label=reshape(x,[3,nvertex]);
catch
return
end
end
if pattr>0
try
address=pattr+(1:(nvertex*4));
R.attr=typecast(B(address),'single');
catch
return
end
end
上記のまとめの関数Mファイル
別途hns_savefreesurfer.mが必要です。
function visa_converts2
% released by Akira Hashizume May 2, 2019
giftidir='C:\ToBeInstalled\MATLAB\gifti-1.6';% sample
addpath(giftidir);
[filename,pathname]=uigetfile('*.nii.gz');
if filename==0;return;end
xx=strfind(filename,'.nii.gz');
name=filename(1:(xx-1));
brainvisa=strfind(pathname,'\t1mri');
if brainvisa>0
mridir=[pathname(1:brainvisa),'mri\'];
surfdir=[pathname(1:brainvisa),'surf\'];
filename2=[pathname,'default_analysis\segmentation\brain_',name,'.nii.gz'];
pathname2=[pathname,'default_analysis\segmentation\mesh\'];
pathname3=[pathname,'default_analysis\segmentation\'];
else
pathname0=[pathname,'brainvisa\'];
if ~exist(pathname0,'dir')
mkdir(pathname0);
filename0=[pathname,name,'.cortex.dewisp.mask.nii.gz'];
F.data=niftiread(filename0);
F.info=niftiinfo(filename0);
filename0=[pathname,name,'.hemi.label.nii.gz'];
G.data=niftiread(filename0);
G.info=niftiinfo(filename0);
F.data(F.data>0)=1;
for n=1:2:17
G.data(G.data==n)=1;
end
for n=2:2:16
G.data(G.data==n)=2;
end
F.data=F.data.*G.data;
for n=1:2
if n==1
savename=[pathname0,name,'_Rwhite.gii'];
else
savename=[pathname0,name,'_Lwhite.gii'];
end
D=F.data;
D=D(:,:,end:-1:1);
for nn=1:size(D,3)
D(:,:,nn)=rot90(squeeze(D(:,:,nn))',2);
end
D(D~=n)=0;
D(D>0)=1;
D=double(D);
pix=F.info.ImageSize.*F.info.PixelDimensions;
x=linspace(0,pix(1),F.info.ImageSize(1));
y=linspace(0,pix(2),F.info.ImageSize(2));
z=linspace(0,pix(3),F.info.ImageSize(3));
[X,Y,Z]=meshgrid(x,y,z);
fv=isosurface(X,Y,Z,smooth3(D),0.5);
fv.faces=fv.faces(:,[1,3,2]);
fv.mat=eye(4);
g=gifti(fv);
save(g,savename,'Base64Binary');
end
disp('Create
L/Rwhite_inflated.gii and L/Rwhite_curv.gii with BrainVISA!');
return;
end
mridir=[pathname,'mri\'];
surfdir=[pathname,'surf\'];
filename2=[pathname,name,'.mask.nii.gz'];
pathname2=[pathname,'brainvisa\'];
pathname3=[pathname,'brainvisa\'];
end
if ~exist(mridir,'dir')
mkdir(mridir);
end
if ~exist(surfdir,'dir')
mkdir(surfdir);
end
savename1=[mridir,'T1.nii.gz'];
copyfile([pathname,filename],savename1);
savename2=[mridir,'brain.nii.gz'];
copyfile(filename2,savename2);
dsize=hns_savefreesurfer(savename1,savename2);
delete(savename1);
delete(savename2);
filenames={'Lwhite.gii','Rwhite.gii','Lwhite_inflated.gii','Rwhite_inflated.gii'};% あえてL/Rhemi.giiは含まないことにする
for n=1:length(filenames)
filename3=[pathname2,name,'_',filenames{n}];
if ~exist(filename3,'file')
continue;
end
savename=[surfdir,filenames{n}];
copyfile(filename3,savename);
if contains(savename,'Lwhite.gii')
dsurfL=hns_savefreesurfer(savename,dsize);
elseif contains(savename,'Rwhite.gii')
dsurfR=hns_savefreesurfer(savename,dsize);
else
hns_savefreesurfer(savename,dsize);
end
delete(savename);
end
if exist('dsurfL','var')
savename=[surfdir,'Lwhite_curv.gii'];
filename4=[pathname3,name,'_Lwhite_curv.gii'];
if exist(filename4,'file')
copyfile(filename4,savename);
hns_savefreesurfer(savename,dsurfL);
delete(savename);
end
end
if exist('dsurfR','var')
savename=[surfdir,'Rwhite_curv.gii'];
filename4=[pathname3,name,'_Rwhite_curv.gii'];
if exist(filename4,'file')
copyfile(filename4,savename);
hns_savefreesurfer(savename,dsurfR);
delete(savename);
end
end