BrainVISAとの位置関係
メッシュファイルの比較
BrainSuiteとBrainVISAは同じNIfTIファイルから脳の切り出し、メッシュ化を行いますが、
メッシュファイルの座標は同じではありません。
以下を実行します。GIfTIを認識させるためA GIfTI MATLAB libraryのアドレスにPATHを通します。
hns_loaaddfsはBrainSuiteのDFSファイルで公開した関数Mファイルです。
addpath('C:\ToBeInstalled\MATLAB\gifti-1.6');
filename='akira_Lhemi.gii';
G=gifti(filename);
filename='akira.left.pial.cortex.dfs';
F=hns_loaddfs(filename);
figure;
subplot(221);
h1=patch('faces',G.faces,'vertices',G.vertices);
set(h1,'facecolor',[1,0,0],'edgecolor','none');
hold on;
h2=patch('faces',F.faces','vertices',F.vertices');
set(h2,'facecolor',[0,0,1],'edgecolor','none');
daspect([1,1,1]);
light('position',[0,0,1]);
light('position',[0,0,-1]);
set(gca,'xlim',[0,240],'ylim',[0,240],'zlim',[0,86*2]);
grid on;
view([0,0]);
for n=1:3
ha=subplot(2,2,1+n);
h=copyobj([h1;h2],ha);
daspect([1,1,1]);
light('position',[0,0,1]);
light('position',[0,0,-1]);
set(gca,'xlim',[0,240],'ylim',[0,240],'zlim',[0,86*2]);
grid on;
switch n
case
1:view([90,0])
case
2;view([0,90])
case
3;view([40,40])
end
end
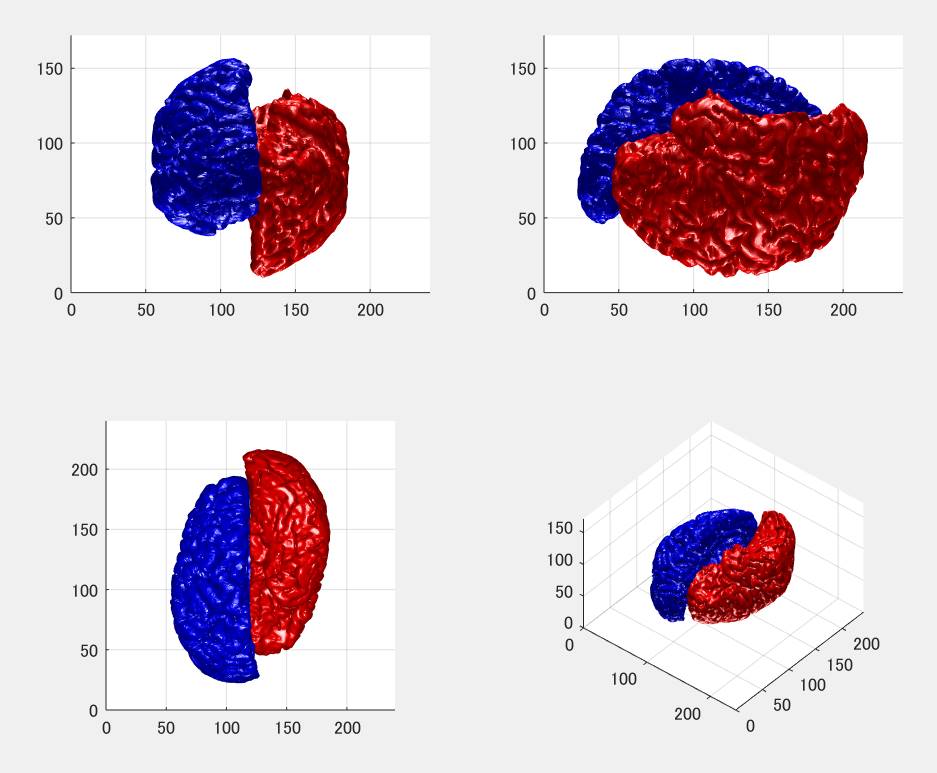
実行結果です。青がBrainSuite、赤がBrainVISAのメッシュです。元のNIfTIファイルのx,y,z座標の中心点で互いに前後左右上下逆になっています。
DFSファイルの座標をを回転
以下を実行しました。
filename='akira_Lhemi.gii';
G=gifti(filename);
figure;
subplot(221);
h1=patch('faces',G.faces,'vertices',G.vertices);
set(h1,'facecolor',[1,0,0],'edgecolor','none','facealpha',0.5);
hold on;
filename='akira.left.pial.cortex.dfs';
F=hns_loaddfs(filename);
filename='akira.nii.gz';
info=niftiinfo(filename);
imagesize=(info.ImageSize).*info.PixelDimensions;
P=F.vertices;
P(4,:)=1;
X=[eye(4,3),[imagesize'/2;1]]*diag([-1,-1,-1,1])*[eye(4,3),[-imagesize'/2;1]]
;
P=X*P;
h2=patch('faces',F.faces','vertices',P(1:3,:)');
set(h2,'facecolor',[0,0,1],'edgecolor','none');
daspect([1,1,1]);
light('position',[0,0,1]);
light('position',[0,0,-1]);
axis tight;
grid on;
view([0,0]);
for n=1:3
ha=subplot(2,2,1+n);
h=copyobj([h1;h2],ha);
daspect([1,1,1]);
light('position',[0,0,1]);
light('position',[0,0,-1]);
axis tight;
grid on;
switch n
case
1:view([90,0])
case
2;view([0,90])
case
3;view([40,40])
end
end
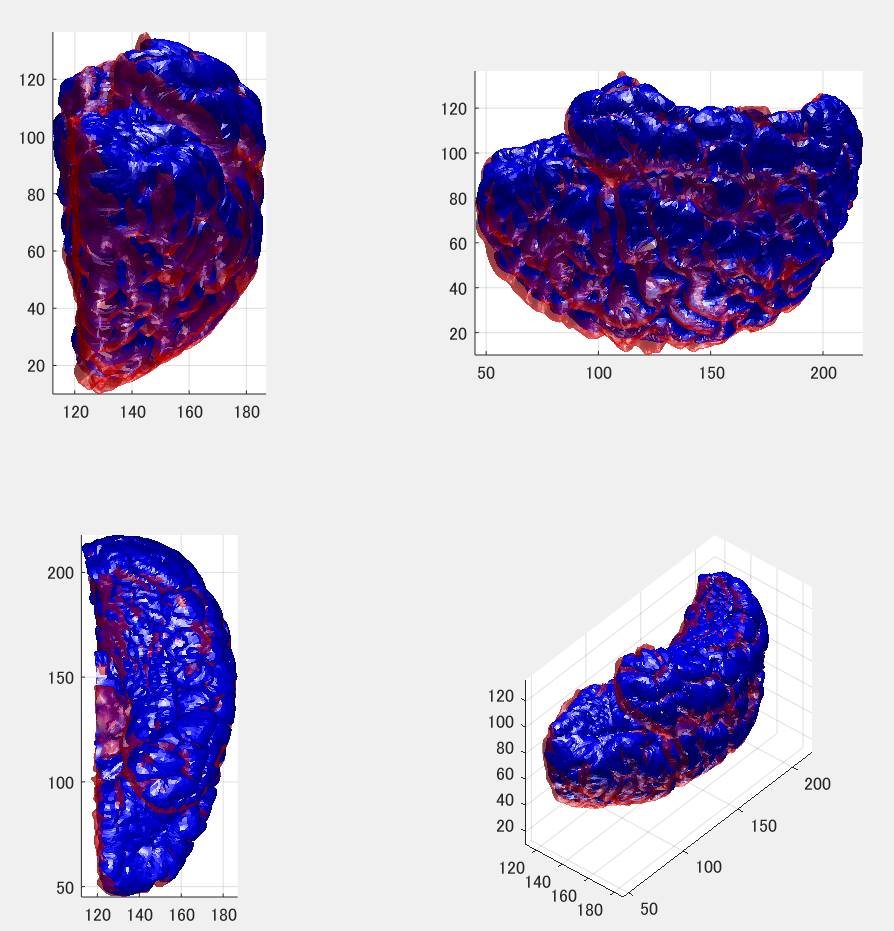
微妙に一致してません。上下方向はずれてるような感じもしますが、実はこれは元の抽出された脳ボクセルの範囲が原因です。
切り出された脳ボクセルの比較
BrainVISAの抽出された脳ボクセルvoronoi_名前.nii.gzをBrainSuiteで見てみます。
BrainVISAでは左半球が1、右半球が2、脳幹が3です。クラスはint16です。
BrainSuiteでは左半球と右半球で255です。クラスはuint8です。
filename='voronoi_akira.nii.gz';
M1=niftiread(filename);
filename='akira.cerebrum.mask.nii.gz';
M2=niftiread(filename);
figure;
for n=1:4
switch n
case
1;D=M1(:,128,:);
case
2;D=M1(128,:,:);
case
3;D=M2(:,128,:);
case
4;D=M2(128,:,:);
end
subplot(2,2,n);
imagesc(rot90(squeeze(D)));
colorbar;
end
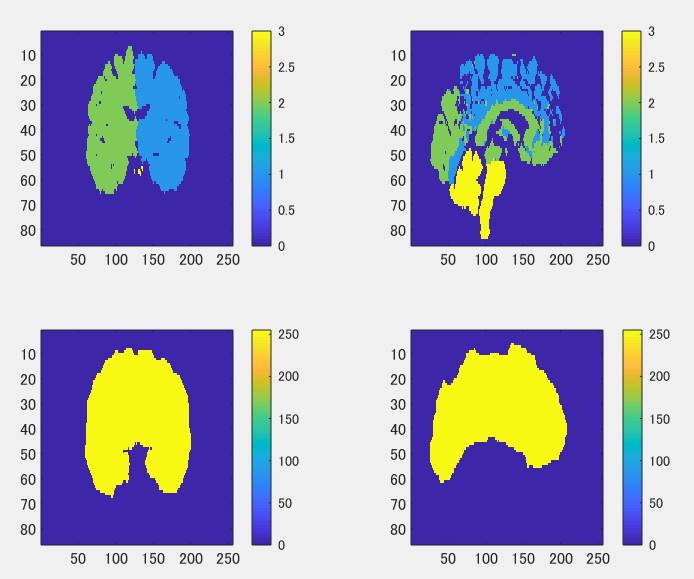
BrainVISAと比較してBrainSuiteの脳抽出が全然ダメダメに見えます。単に脳皮質抽出過程の最終段階か初期段階かの違いでしかありません。
いろいろ調べたんですがBrainSuiteには脳皮質抽出の最終段階を示すNIfTIファイルが見当たりませんでした。
BrainVISAのvoronoi_名前.nii.gzファイルをBrainSuite用の名前.mask.nii.gzに変換してみました。
filename='voronoi_akira.nii.gz';
M1=niftiread(filename);
M1(M1>2)=0;
M1(M1>0)=255;
M1=uint8(M1);
info=niftiinfo(filename);
info.Datatype=class(M1);
info.BitsPerPixel=8;
filename='voronoi_akira.mask.nii';
niftiwrite(uint8(M1),filename,info);
BrainSuiteでマスクとして読み込んでみます。
BrainVISAで作成したメッシュの元となった抽出脳皮質データがBrainSuiteで正確に皮質面を抽出できていることがわかりました。
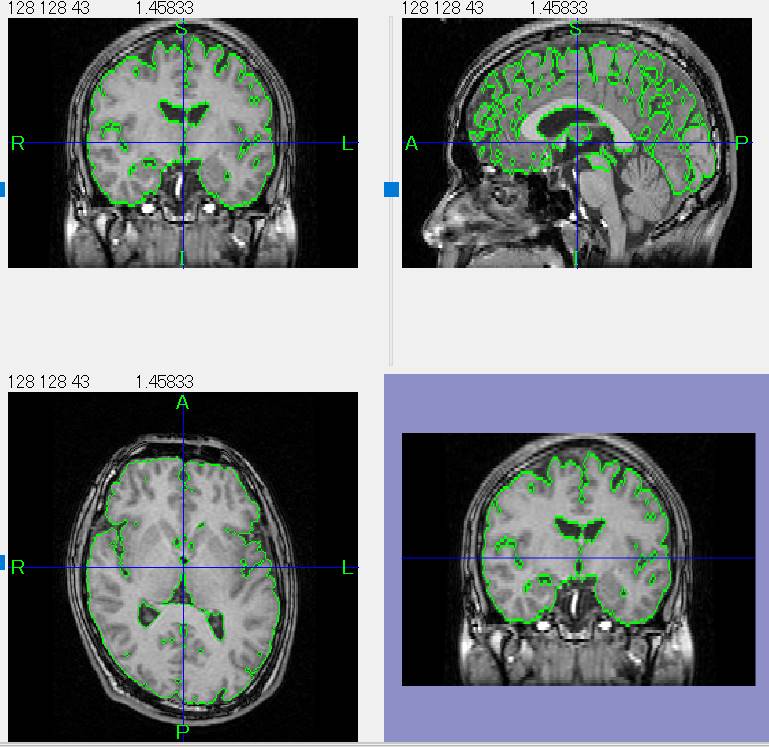
因みにBrainSuiteの名前.cerebrum.mask.nii.gzを読み込んだところです。