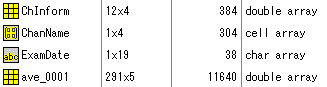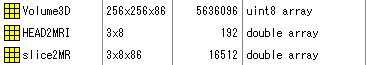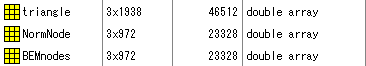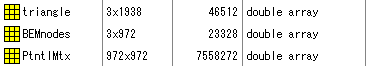Fiff2MatFileの説明
Fiff2MatFileはニューロマグ社のファイルFIFF fileを米国MathWorks社製信号処理ソフトMatlabのMAT fileに変換するソフトです。
Microsoft Windows XPのみで動作確認しています。得られた変数について簡単に説明します。ただし
ソフトを使用して生じたいかなる結果に対しても当方は責任を負いません。自己責任で使用してください。
Fiff2MatFileの活用例は
国立精神・神経センター武蔵病院 金子裕先生のホームページを参照ください。
evoked dataの場合
evoked dataを読むとWorkspaceに
と表示されます。変数の意味は以下のとおりです。

raw dataの場合
raw dataを読むとWorkspaceに

と表示されます。変数の意味は以下のとおりです。
raw dataはファイルサイズがかなり大きくなり、MATLABでは認識できないことがあるので、
FIFF fileで圧縮してあるとおり、係数×正負16bitで保存してあります。
ch10の情報を引き出すには
CalXRnge(10)*double(raw_data(10,:))
とします。
またサンプリング周波数は
CalXRnge(end)=600.6150
として取り出します。

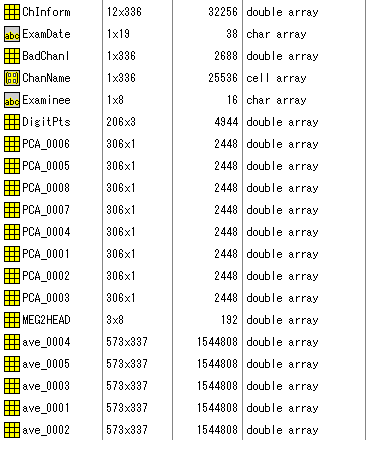
xfit_wavesの場合
Source ModellingのSource waveformsをfiff fileで保存したものをMat化して読み込むと
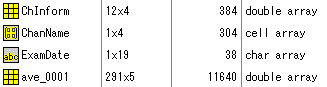
と表示されます。変数の意味は以下のとおりです。
| 変数名 | 意味 |
| ChInform | チャンネル情報 |
| ChanName | チャンネル名 |
| ExamDate | 作成日 |
| ave_0001 | 波形データ |
ChInform(1:3,1:(end-2))はdipoleの位置(m)
ChInform(4:6,1:(end-2))はdipoleの単位ベクトル化された方向
を意味します。
ave_0001(:,1:(end-2))は電流の大きさ(nA)
ave_0001(:,(end-1))はGOF(%)
ave_0001(:,end)は時間(msec)を意味します。

xxx.bdipの場合
xxx.bdipをMat化して、読みこむとWorkspaceに

と表示されます。変数の意味は以下のとおりです。

xxx.meshの場合
SegLabで作成したxxx.meshをMat化して、読みこむとWorkspaceに
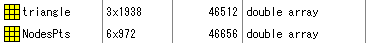
と表示されます。変数の意味は以下のとおりです。
| 変数名 | 意味 |
| triangle | メッシュ点の三角形の組み合わせ |
| NodesPts | メッシュ点の座標と法線方向 |

sets内のfiffの場合
sets内のfiff fileをmat変換後に読むとWorkspaceに

と表示されます。変数の意味は以下のとおりです。
ここではslices内のDICOM fileが86枚あること示しています。

xxx_3D.fifの場合
ここでxxx_3D.fifとはSegLabで脳の切り出しsegmentationを行ったデータを想定しています。
このfileをmat化後に読むとWorkspaceに
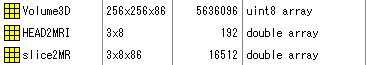
と表示されます。
変数の意味は以下のとおりです。

xxx-bem.fifの場合
ここでxxx_bem.fifとはSource Modellingで作成された境界要素モデルboundary element modelを想定しています。
このfileをMat化し、読み込むとWorkspaceに
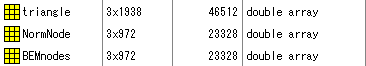
と表示されます。
変数の意味は以下のとおりです。
| 変数名 | 意味 |
| triangle | メッシュ点の三角形の組み合わせ |
| NormNode | メッシュ点上の法線方向 |
| BEMnodes | メッシュ点の位置 |

xxx-bem-sol.fifの場合
ここでxxx_bem-sol.fifとはSource Modellingで作成された境界要素モデルboundary element modelを想定しています。
このfileをMat化し、読み込むとWorkspaceに
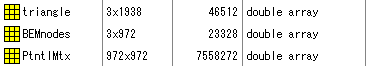
と表示されます。
変数の意味は以下のとおりです。
| 変数名 | 意味 |
| triangle | メッシュ点の三角形の組み合わせ |
| BEMnodes | メッシュ点の位置 |
| PtntlMtx | BEMモデルの電圧計算用行列? |
最後のPtntlMtx = potential solution matrixは何のことだかわかりません。
もっとも境界要素法、有限要素法自体を理解していませんが。

...-bem.trinet.mat
trinetという変数だけですが、実は構造体になっています。
| 変数名 | 意味 |
| tn_id | ? |
| creator | 作者 |
| datafile | 元になったbem.fif |
| original | path付datafile |
| comment | コメント |
| nodes | メッシュ点の座標 |
| triangles | メッシュ点の三角形の組み合わせ |
| ntri | 三角形の数 |
| nnodes | メッシュ点の数 |
| subj_id | ? |

...-bem_pointsetXX.mat
pointsetという変数だけですが、実は構造体になっています。
| 変数名 | 意味 |
| points | 格子点の座標 |
| comment | コメント |
| r0 | 球モデルの中心座標 |
| lattice | 格子点間の距離 |
| minr | 格子点を除去した球の半径 |
| ps_id | pointsetのID |
| tn_id | ? |
| creator | 作者 |
| proj | 格子点の値をメッシュ点に投射する行列 |
| modeltype | 球モデル(BEMモデル) |
| subj_id | ? |
| ori | 格子点の接平面ベクトル |
| originalps | ? |
| trans | ? |
| createtime | 作成日時+時刻 |
MCE version 1.3は球モデルを使っています。
MCE version 1.4は境界要素モデルが使えるという噂。

ave_00XX
誘発データ情報です。誘発データには本来固有の名前がついているのですが、
MATLABの変数として使えない文字があるので便宜的にAVE_00XXとしています。
Neuromag System (306Ch)+脳波21chの場合
- ave_0001(:,1:306)は306Chの脳磁図データ
- ave_0001(:,307:312)は6ChのSTIMデータ
- ave_0001(:,313:315)は3ChのSTIMデータ(HPIコイル入り決め用)
- ave_0001(:,316:336)は21Chの脳波データ
- ave_0001(:,end)は時間データ
を意味します。
binaryDP
binaryで保存されたdipoleの情報です。
- binaryDP(1:2,:) 潜時 msec
- binaryDP(3:5,:) head座標上の位置 m
- binaryDP(6,:) 電流量 nA
- binaryDP(7:9,:) head座標上のx,y,z方向の電流の大きさ nA
- binaryDP(10,:) GOF %
- binaryDP(11,:) spatial conficence volume mm^3
- binaryDP(12,:) χ2値
- binaryDP(13,:) χ2試験で否定される確率
CalXRnge
raw dataにかける係数です。例えば10chのデータを得るときには
CalXRnge(10)*double(raw_data(10,:))
などとします。
ただし、CalXRnge(end)はサンプリング周波数を意味します。
広島大学病院の場合、係数は以下のとおりになっています。
- 平面型グラジオメーター ±9.643554807・・・fT/cm
- マグネトメーター 0.2551269590・・・fT
- STIM 0.000305175・・・ V
- 脳波 0.009068375・・・μV
要するにraw_dataは約9.6fT単位で四捨五入されています。
ChInform
Neuromag System (306Ch)+脳波21Chの場合は以下のとおりです。
- ChInform(1:3,1:306)はdevice座標でのチャンネルの位置
- ChInform(4:6,1:306)はdevice座標でのx方向(差分方向)の単位ベクトル
- ChInform(7:9,1:306)はdevice座標でのy方向の単位ベクトル
- ChInform(10:12,1:306)はdevice座標でのz方向(チャンネル面の法線方向)の単位ベクトル
- ChInform(:,1:3:304)は横方向差分磁場用の平面型グラジオメーター
- ChInform(:,2:3:305)は縦方向差分磁場用の平面型グラジオメーター
- ChInform(:,3:3:306)はマグネトメーター
- ChInform(:,307:312)はSTIMの1-6
- ChInform(:,313:315)はHPIコイル位置推定用のSTIMの7-9
- ChInform(:,317:336)は脳波
を意味します。
センサーの表示は以下のとおりです。
wd=14/1000; % センサー幅の半分
P=ChInform(1:3,1:3:304); % センサーの中心
X=wd*ChInform(4:6,1:3:304); % センサーの横幅の半分
Y=wd*ChInform(7:9,1:3:304); % センサーの縦幅の半分
V=zeros(4,3,102); % あらかじめ領域確保
V(1,:,:)=P+X+Y;V(2,:,:)=P+X-Y;V(3,:,:)=P-X-Y;V(4,:,:)=P-X+Y;% センサーの4隅の座標
fill3(squeeze(V(:,1,:)),squeeze(V(:,2,:)),squeeze(V(:,3,:)),zeros(4,102));
daspect([1,1,1]); % 縦・横・高さ比を1:1:1にする。
rotate3d; % マウスで画像を回転させる
とすれば図のようにセンサーが表示されます。

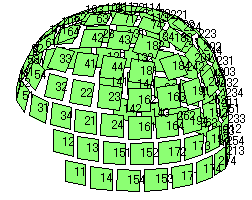
Neuromagはチャンネル名を解剖名と無関係につけているのでチャンネル名がどの位置かわかりません。
そこで
ChanName=ChanName{1:3:304};
ChanName=strrep(ChanName,'MEG ',''); % 文字列'MEG 'を除去
hold on; % 重ねがき許可
for x=1:102;...
Ch=str2num(ChanName{x});... % 文字列を整数に変換
Ch=floor(Ch/10);... % 下一桁を除去
Ch=num2str(Ch);... % 整数を文字列に変換
text(P(1,x),P(2,x),P(3,x),Ch);end;
とすれば下図のようになります。
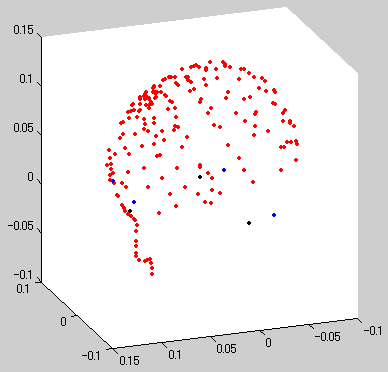
DigitPts
ポルヘムスで認識した点の位置を示します。座標はhead座標です。
HPI coil 4個使用、脳波電極の位置認識なしの時の場合は以下のとおりです。
- DigitPts(1,:)は左耳のhead座標での位置
- DigitPts(2,:)はnasionのhead座標での位置
- DigitPts(3,:)は右耳のhead座標での位置
- DigitPts(4:8,:)はHPIコイルのhead座標での位置
- DigitPts(9,:)は頭皮をプロットした点のhead座標での位置
を意味します。
plot3(DigitPts(1:3,1),DigitPts(1:3,2),DigitPts(1:3,3),'k.'); % 基準点は黒
hold on; % 重ねがき許可
plot3(DigitPts(4:7,1),DigitPts(4:7,2),DigitPts(4:7,3),'b.'); % HPIコイルは青
plot3(DigitPts(8:end,1),DigitPts(8:end,2),DigitPts(8:end,3); % その他の点は赤
view([-110,20]); % 見る角度の指定
daspect([1,1,1]); % 縦・横・高さの比率
とすると下図のように表示されます。
HEAD2MRI
head座標とMRI座標の変換行列とその逆行列を意味します。具体的には
- HEAD2MRI(:,1:3)はhead座標→MRI座標に回転
- HEAD2MRI(:,4)はhead座標→MRI座標に水平移動
- HEAD2MRI(:,5:7)はMRI座標→head座標に回転
- HEAD2MRI(:,8)はMRI座標→head座標に水平移動
となります。
MEG2HEAD
device座標とhead座標の変換行列とその逆行列を意味します。具体的には
- MEG2HEAD(:,1:3)はdevice座標→head座標に回転
- MEG2HEAD(:,4)はdevice座標→head座標に水平移動
- MEG2HEAD(:,5:7)はhead座標→device座標に回転
- MEG2HEAD(:,8)はhead座標→device座標に水平移動
となります。
PCA_00XX
SSPの個々の固有ベクトルを意味します。
SSPの固有ベクトルにはそれぞれfiffで名前がついているのですが、Matlabでは使えない文字があるので、
便宜上PCA_00XXとしています。32bit浮動小数点の情報を64bit浮動小数点にしただけですので精度が悪く、例えば
PCA_0001'*PCA_0002=-4.01020e-0008
とゼロになりません。
Neuromag System (306Ch)の場合
SSPを作るとき、平面型グラジオメーターとマグネトメーターの固有ベクトルは別々に作成されます。
マグネトメーターの固有ベクトルがPCA_0004の時、
sum(PCA_0004(3:3:306).^2)=1.0000
となりますが、
sum(PCA_0004(1:3:304).^2+PCA_004(2:3:305).^2)=0
となります。
不良channelを除外してSSPが作られた場合、
PCA_000Xのサイズが204x1や306x1になりません。
raw_data
raw dataの情報です。正負付16bit整数、すなわち-32768〜32767の整数で保存されています。
係数CalXRngeをかけてデータを復元します。
slice2MR
MRI座標と/slices内DICOMファイル1枚1枚の画素との変換行列とその逆行列を意味します。具体的には
- slice2MR(:,1:3,x)はx番目のDICOM fileの画素→MRI座標に回転
- slice2MR(:,4,x)はx番目のDICOM fileの画素→MRI座標に水平移動
- slice2MR(:,5:7,x)はMRI座標→x番目のDICOM fileの画素に回転
- slice2MR(:,8,x)はMRI座標→x番目のDICOM fileの画素に水平移動
となります。
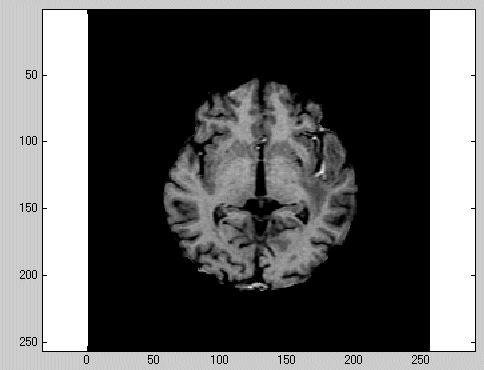
Volume3D
SegLabでxxx_3D.fifとして保存したfileをMat化した場合
元になるDICOMファイルが256x256で86枚あるとすると、Volume3Dは256x256x86の3次元配列となります。
正の8bit、0〜255の範囲で保存されています。
通常MRIはこの範囲で十分ですが、
CTでは-4000〜4000程度必要なことがあり、
この場合DICOM値が正しく保存されません。
V=squeeze(Volume3D(:,:,50)); % スライス選択
imagesc(V,[20,80]); % windowを20〜80とする
colormap(gray); % 白黒スケール
axis equal; % 縦・横比を1:1にする。
とすると以下のように表示されます。
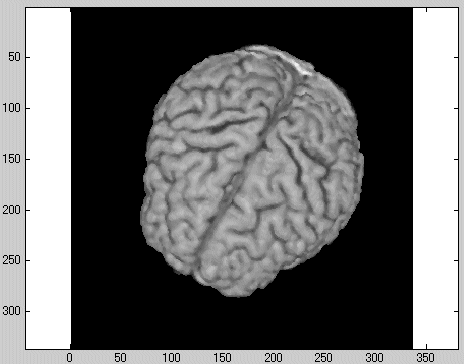
3D脳表画像を作成してxxx_3D.scenes.fifとして保存したfileをMat化した場合
通常は脳表画像60枚が白黒8bitで保存されています。
imagesc(squeeze(Volume3D(:,:,5)),[0,255]); % 画像表示
colormap(gray); % 白黒表示
axis equal; % 縦・横比を1:1にする。
とすると以下のように表示されます。