MEG用MRIをSPMに
標準脳化をめざし、実際に脳磁図用のMRIの三次元データをSPMにのせてみました。
ファイル名の連番化
Rearranger_2.exeを起動し、*.dcmボタンを押します。
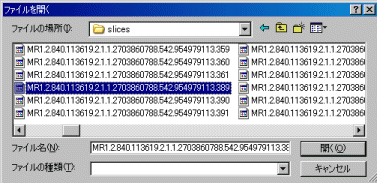
目的とするsequenceの一番若い番号名のDICOMファイルを選びます。
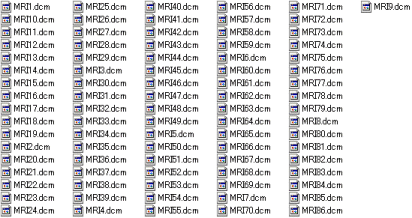
MRという名前で保存することにすると、MRI1.dcm〜MRI86.dcmが作成されました。
これでRearranger_2.exeを終了します。
Analyzeフォーマット化
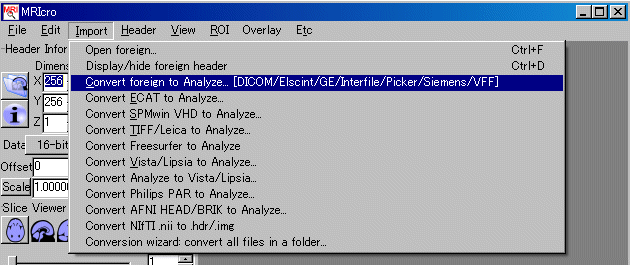
MRIcroを起動し、メニューバーのImport->Convert foreing to Analyzeを選択します。
Numbers of Filesファイル枚数を書き込み、Selectボタンを押します。
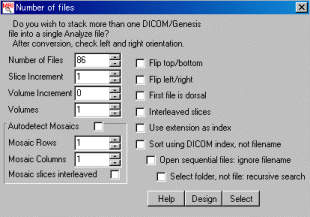
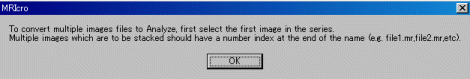
messageがでます。OKボタンを押します。
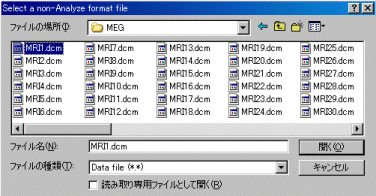
***1.dcmというファイルを選択します。
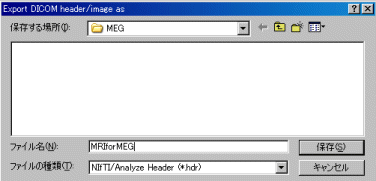
Analyzeフォーマットのファイル名を聞いくるので、任意の名称をつけ、保存ボタンを押します。
***.hdrと***.imgという2つのファイルが作成されました。

これでMRIcro.exeを終了します。
SPMで標準脳化
MATLABを起動し、pathやcdなどの設定を行った後
>>spm
とすれば、SPMが立ち上がります。
ここではSPM2を想定しています。

ボタン、何もなし、説明のwindowが3つ開きます。
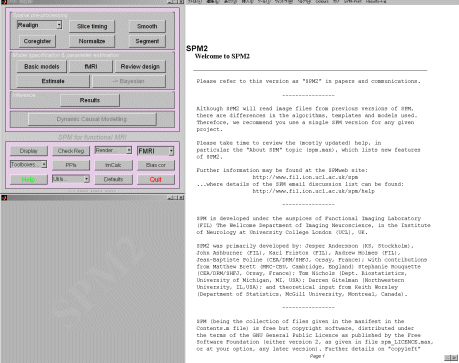
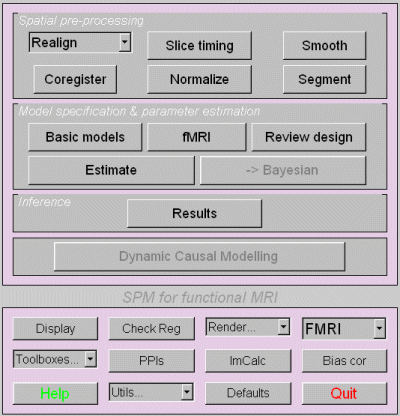
Normalizeボタンを押します。
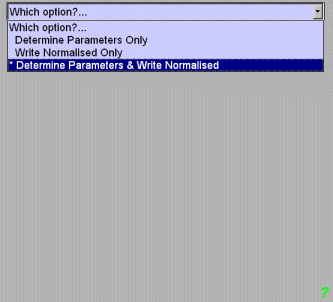
Which option?...と聞いてきます。一番最後のDetermine Parameters &...を選びます。
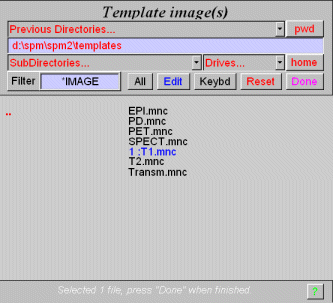
MINC形式で保存された標準脳の読み込み
Template image(s)にT1.mncを選択し、Doneボタンを押します。
被験者1の脳解剖画像の読み込み
Source image,subj1に先ほど作成した***.imgを選択し、Doneボタンを押します。
被験者1の脳機能画像の読み込み
Images to write,subj1はデータがないので無視して、Doneボタンを押します。
Source image,subj2もDoneボタンを押します。
Affine Registrationというwindowが開き、なにやらしています。
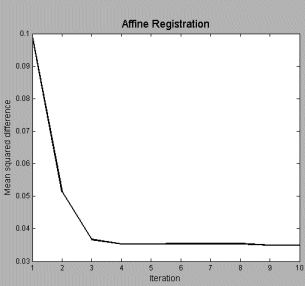
次に**% Completeの縦グラフが出てきます。
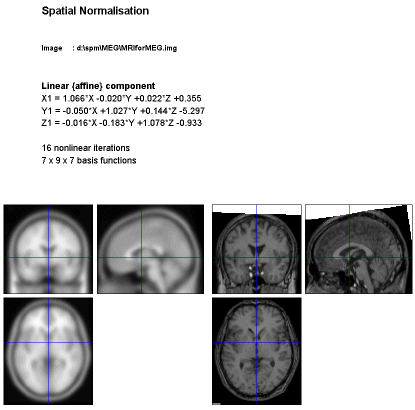
Normaliseが終了し、結果が表示されました。
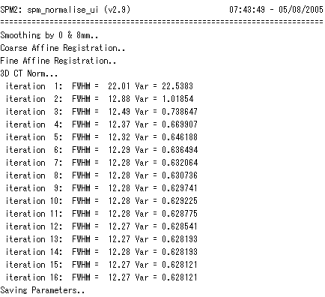
MATLABのCommand windowをみると、以下のように表示されていました。
新たに、***_sn.matというMATLABのデータファイルが作成されていました。






















