spm_sample_vol
三次元配列を線形補間するときに、Matlabはinterp3という関数を用意しています。
interp3は三次元配列が大きくなるとすぐに
??? メモリが足りません.HELP MEMORYとタイプしてオプションを確認してください
と表示され、MRI画像のデータは事実上扱えません(Windows版Matlab)。
この機能に相当するのが
spm_sample_vol.dll...Windows
spm_sample_vol.mexglx...Linux
spm_sample_vol.mexmac...Macintosh
spm_sample_vol.mexsol...Solarix
spm_sample_vol.mexlx...Linux?
などのMEX関数です。interp3.mとspm_sample_vol.dllの互換性を試してみます。
V=rand(50,50,50);
[x,y,z]=meshgrid(1:size(V,1),1:size(V,2),1:size(V,3));
[xi,yi,zi]=meshgrid(1:0.5:size(V,1),1:0.5:size(V,2),1:0.5:size(V,3));
tic;V1=interp3(x,y,z,V,xi,yi,zi);toc
tic;V2=spm_sample_vol(V,yi,xi,zi,1);toc % yiとxiの入れ替え!
max(max(max(abs(V1-reshape(V2,size(V1,1),size(V1,2),size(V1,3))))))
Intel Pentium4 3.2GHzのPCではinterp3が2.4370秒、spm_sample_volが0.4530秒、誤差が6.1062e-015と出ました。
spm_sample_volを使ったほうが高速であることがわかります。
spm_sample_volでxiとyiを入れ替えたのは、spm_sample_volがmeshgridでなく、
同様の機能を持つndgridを利用しているためです。
またMEX関数の制限のためか、三次元配列を出力できないので、
出力結果をreshapeを用いて三次元配列に改める必要があります。
256x256x86、FOV240mm、slice厚2mm、slice間隔0のMRIデータを1mmの立方体からなる三次元配列に変換してみます。
clear all
[filename,pathname]=uigetfile('*.img'); % *.img fileを選択
VF=spm_vol(strcat(pathname,filename)); % 被験者脳のheaderを読む
VF.dat=spm_read_vols(VF); % 被験者脳の三次元配列を読む
VF.dat=int16(VF.dat); % 8byte浮動小数点配列から2byte整数配列へ
x=linspace(1,VF.dim(1),abs(VF.mat(1,1)*VF.dim(1))); % =linspace(1,256,240);
y=linspace(1,VF.dim(2),abs(VF.mat(2,2)*VF.dim(2))); % =linspace(1,256,240);
z=linspace(1,VF.dim(3),abs(VF.mat(3,3)*VF.dim(3))); % =linspace(1,172,86);
[xi,yi,zi]=ndgrid(linspace(1,256,240),linspace(1,256,240),linspace(1,86,86*2));
tic;W=spm_sample_vol(VF.dat,xi,yi,zi,1);toc
W=reshape(int16(W),240,240,86*2);
figure;imagesc(rot90(squeeze(W(120,:,:))),[0,384]);
axis equal;axis tight;colormap(gray);
clear xi yi zi;
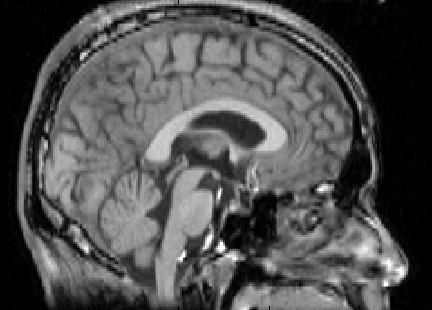
線形補間に要した時間は4.6090秒でした。
1mm voxwlに変換するだけであればMRIcroでできます。
0.5mmの立方体からなる三次元配列に変換してみます。
但し、Windows版では1変数のメモリ上限がやかましいので、少し工夫し四分割します。
clear W;close all;
[xi,yi,zi]=ndgrid(linspace(1,128,240),linspace(1,128,240),linspace(1,86,86*4));
W1=spm_sample_vol(VF.dat(1:128,1:128,:),xi,yi,zi,1);
W1=reshape(int16(W1),240,240,86*4);
W2=spm_sample_vol(V(129:end,1:128,:),xi,yi,zi,1);
W2=reshape(int16(W2),240,240,86*4);
W3=spm_sample_vol(V(1:128,129:end,:),xi,yi,zi,1);
W3=reshape(int16(W3),240,240,86*4);
W4=spm_sample_vol(V(129:end,129:end,:),xi,yi,zi,1);
W4=reshape(int16(W4),240,240,86*4);
clear xi yi zi;
% 128と129の間の補間は無視しています。
MatArray=repmat(int16(0),[480,480,344]);
MatArray(1:240,1:240,:)=W1;
MatArray(241:end,1:240,:)=W2;
MatArray(1:240,241:end,:)=W3;
MatArray(241:end,241:end,:)=W4;
clear W1 W2 W3 W4;
figure;imagesc(rot90(squeeze(MatArray(240,:,:))),[0,384]);
axis equal;axis tight;colormap(gray);
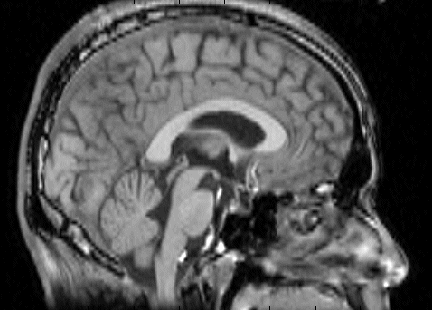
なんとか表示できました。CTの0.5mm voxelにも対応できそうです。

