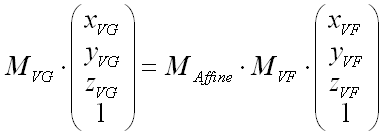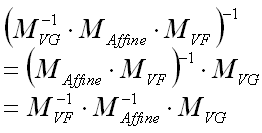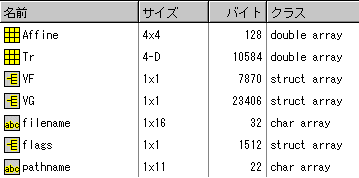
| fname | ファイル名 |
| dim | x,y,zの三次元配列の要素数とデータ型 |
| mat | 三次元配列を実空間座標に変換する行列 |
| pinfo(1,:) | (:,:,z)平面上の信号のscale |
| pinfo(2,:) | (:,:,z)平面上の信号のoffset |
| pinfo(3,:) | (:,:,z)平面へのoffset? |
| n | ? |
| descrip | 撮影時間やファイル形式など |
| private | fnameのheader情報(構造体の入れ子) |
| dat | 実際の三次元配列(削除されている) |
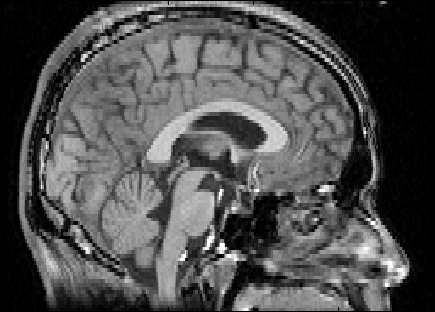
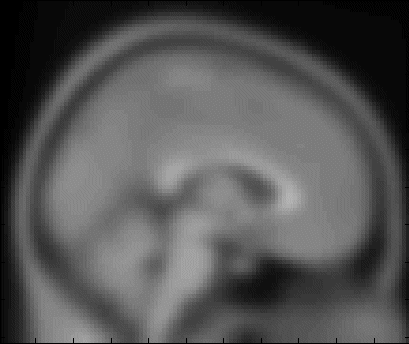
| smosrc | 被験脳の平滑化の半値幅 | 8 |
| smoref | 標準脳の平滑化の半値幅 | 0 |
| regtype | 規則化の方式 'none','rigid','subj','mni'がある | mni |
| weight | 重み付け(病変脳用?) | '' |
| cutoff | 離散cosine変換の切捨て? | 30 |
| nits | 非線形変換の繰り返し回数 | 16 |
| reg | 規則化の変換量? | 0.1 |
| wtsrc | 被験者脳の重み付けの何か? | 0 |
| graphics | spm_normalise_disp.mの実行 | 1 |
| version | version | '@(#)spm_normalise.m 2.8 03/03/04' |
| date | SPM実行日 | Matlabのdate関数 |