標準脳化脳をみる
標準脳化した被験者脳をMRIcroでみてみます。
予め標準脳のvoxelは2mmなので、1mmに変換しておきます。
% 標準脳のvoxelを1mmに変換
[filename,pathname]=uigetfile('*.mnc'); % 標準脳を選択
VG=strcat(pathname,filename);
VG=spm_vol(VG);
VG.dat=spm_read_vols(VG);
x=linspace(1,VG.dim(1),VG.dim(1)*2);
y=linspace(1,VG.dim(2),VG.dim(2)*2);
z=linspace(1,VG.dim(3),VG.dim(3)*2);
[x,y,z]=ndgrid(x,y,z);
VG.dat=spm_sample_vol(VG.dat,x,y,z,1);
clear x y z;
VG.dat=reshape(VG.dat,VG.dim(1:3)*2);
fid=fopen(strcat(pathname,'new_template.img'),'wb');
fwrite(fid,VG.dat*2^8,'uint8');
fclose(fid);
MRIcroのheaderを以下のようにします。
即ち、x,y,zのdimensionを2倍にし、sizeを1mmに、Dataを8-bit intとします。
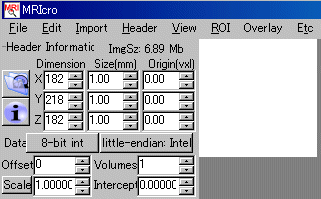
MRIcroのmenu bar→Header->Save header...を選択し、同じfolderにnew_template.hdrという名前で保存します。
VG=strcat(pathname,'new_template.img');
[filename,pathname]=uigetfile('*.img'); % 被験者脳の選択
VF=strcat(pathname,filename);
spm_defaults;
flags=defaults.normalise.estimate;
prm=spm_normalise(VG,VF,'','','',flags); % 標準脳化
% 1mm voxelだと標準脳化処理は時間がかかります。
Vdct=spm_write_sn(VF,prm,flags); % Affine変換+非線形変換
prm.Tr=[]; % Tr=[]で非線形変換は行われなくなる
Vaff=spm_write_sn(VF,prm,flags); % Affine変換のみ
Vaff.fname=strcat('aff_',Vaff.fname); % imgファイルとして保存
spm_write_vol(Vaff,Vaff.dat);
Vdct.fname=strcat('dct_',Vdct.fname); % imgファイルとして保存
spm_write_vol(Vdct,Vdct.dat);
MRIcroのSkullStrip機能を使って脳表を提示してみます。
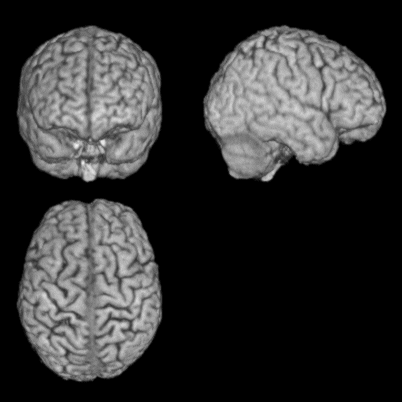
被験者の脳表画像です。
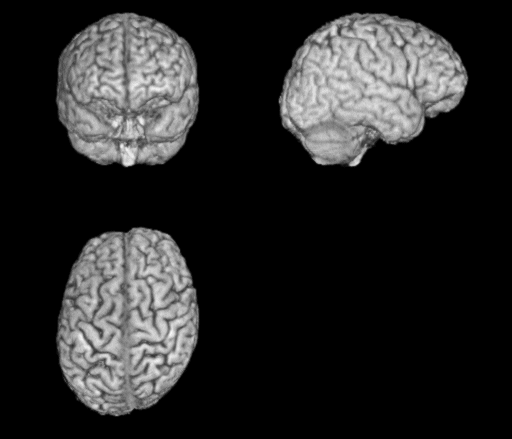
Affine変換後の脳表画像です。MRIcroのSkullStrip機能ではどうも脳底面が削られてしまうようです。
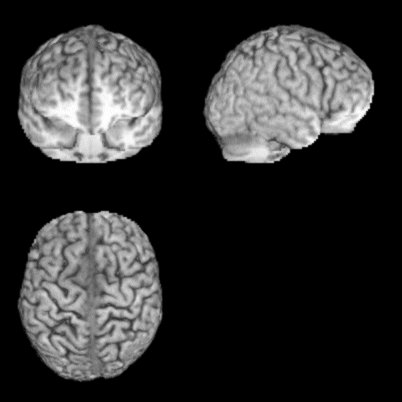
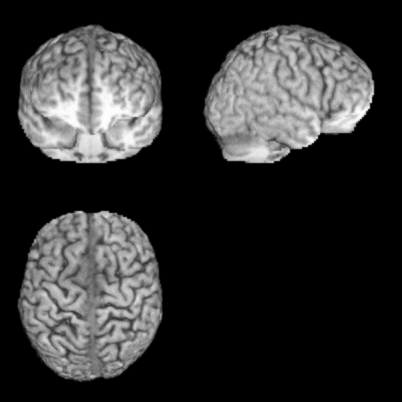
Affine変換+非線形変換後の脳表画像です。
Affine変換だけのときと比較して僅かに変化しています。
個人頭→標準化→脳切り出しとすると脳底面が削られるようです。
そこで、個人頭を標準化したparameterを利用して
個人頭→標準化→個人脳から脳切り出し→標準化としました。
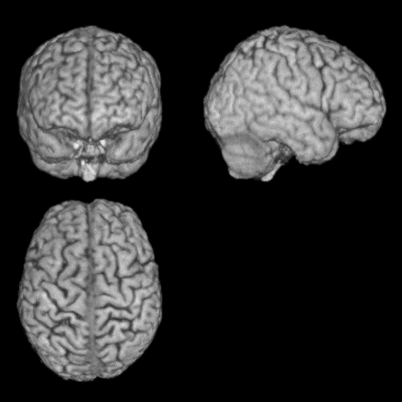
ちなみにAffine変換だけのときは以下の図になります。