MCEの結果の標準脳化
右正中神経電気刺激による体性感覚誘発磁界の電流源を
Minimum Current Estimation (MCE)で求めて得られた結果を標準脳化します。
格子点間隔は10mmです。
[filename,pathname]=uigetfile('*full.mat'); % MCEの結果(全時間)
load(strcat(pathname,filename));
Q=full(q1.^2+q2.^2).^0.5;
time=((1:size(Q,2))-1)/fs+t0; % 時間
T=find(time>0.015 & time<0.025); % 15〜25msecの時間サンプル
q=sum(Q(:,T),2); % 15〜25msecの各格子点における電流の積算
[filename,pathname]=uigetfile('*pointset*.mat'); % 格子点情報
load(strcat(pathname,filename));
P=pointset.points*1000; % mm単位に
L=pointset.lattice*1000; % 格子点間隔 mm以下の端数はないという前提
nx=max(abs(P(:,1)))+L;nx=-nx:L:nx; % 脳実質外のLatticeを含める
ny=max(abs(P(:,2)))+L;ny=-ny:L:ny;
nz=max(abs(P(:,3)))+L;nz=-nz:L:nz;
P=round(P);nx=round(nx);ny=round(ny);nz=round(nz); % 丸め誤差除去
V=zeros(length(nx),length(ny),length(nz));
for n=1:size(P,1);...
V(find(nx==P(n,1)),find(ny==P(n,2)),find(nz==P(n,3)))=q(n);...
end;
[filename,pathname]=uiputfile('*.img');
fid=fopen(strcat(pathname,filename),'wb');
fwrite(fid,V*1e+9,'double'); % 単位はnA 小さすぎるとMRIcroで認識できない?
fclose(fid);
clear;
機能画像の大きさはPのx,y,z座標の最大値×2+3です(ここでは-7〜7、-10〜10、-12〜12cm)。
*.imgファイルとして保存しています。MRIcroでDimension、Size、Dataを指定し、*.hdrとして保存します
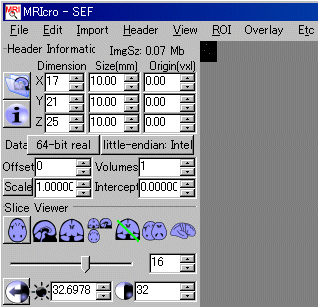
見づらいですが、windowとlevelを調整すれば信号が確認できます。
次に、解剖画像のMRI座標を脳磁図座標に変換し、
先ほどの機能画像の三次元配列座標中心と脳磁図座標上の解剖画像の中心が一致するようにします。
が、Analyze形式のMRIの三次元配列と脳磁図のMRIの三次元配列の配列順序は異なっているので調整が必要です。
脳磁図用のMRIの元画像が水平断であるという仮定して話を進めます。
[filename,pathname]=uigetfile('*'); % 水平断一番下のDICOM fileを選択
d=spm_dicom_headers(strcat(pathname,filename));
scale=[d{1}.PixelSpacing,d{1}.SliceThickness];
translation=d{1}.ImagePositionPatient; % 基準点は右後?
translation(1:2)=scale(1:2).*[d{1}.Rows,d{1}.Columns]+translation(1:2);
Mdicom=eye(4);
Mdicom(1:3,1:3)=diag(scale.*[-1,1,1]); % 左右反転
Mdicom(1,4)=translation(1)+scale(1); % C言語では配列は0から始まるので補正
Mdicom(2,4)=-translation(2)-scale(2); % 経験則です
Mdicom(3,4)=translation(3)-scale(3); % そうとう悩みました
[filename,pathname]=uigetfile('*.mat'); % sets file
load(strcat(pathname,filename));
HEAD2MRI=HEAD2MRI(1:3,1:4);
HEAD2MRI(1:3,4)=HEAD2MRI(1:3,4)*1000;
HEAD2MRI(4,:)=[0,0,0,1];
これでAnaylise形式のMRI三次元配列→MRI座標(Mdicom)の変換行列と、
脳磁図のHPI座標→MRI座標(HEAD2MRI)の変換行列ができました。
spm_slice_volを使って、解剖画像と機能画像の三次元配列の中心を一致させます。
[filename,pathname]=uigetfile('*.img'); % 解剖画像
W=spm_read_vols(spm_vol(strcat(pathname,filename)));
[filename,pathname]=uigetfile('*.img'); % 先ほど保存した機能画像
V=spm_vol(strcat(pathname,filename));
Vsize=(V.dim(1:3)-1)*10+1;
K=eye(4);K(1:2,4)=round(Vsize(1:2)'/2);
newW=zeros(Vsize);
for z=1:Vsize(3);...
K(3,4)=round(Vsize(3)/2)-z;...
mat=inv(K*inv(HEAD2MRI)*Mdicom);...
newW(:,:,z)=spm_slice_vol(W,mat,Vsize(1:2),1);...
end;
newW(newW==NaN)=0;
[filename,pathname]=uiputfile('*.img');
fid=fopen(strcat(pathname,filename),'wb');
fwrite(fid,newW,'int16');fclose(fid);clear;
MRIcroでhdrファイルを作成します。
機能画像(V)および解剖画像(newW)の原点(0,0,0)が、それぞれの三次元配列の中央になるようにしています。
ここではわかりやすくするため脳実質の下方部分のデータも計算していますが、
計算効率を求めるときにはVsizeを切り詰めます。
ではいよいよ標準脳化します。
[filename,pathname]=uigetfile('*.mnc'); % 標準脳templateを選択
VG=strcat(pathname,filename);
[filename,pathname]=uigetfile('*.img'); % 脳磁図HPI座標に変換した解剖画像を選択
VF=strcat(pathname,filename);
spm_defaults; % 初期値設定
prm=spm_normalise(VG,VF,'','','',defaults.normalise.estimate); % 処理に時間がかかります
[filename,pathname]=uigetfile('*.img'); % 機能画像を選択
Vfunc=spm_write_sn(strcat(pathname,filename),prm);
Vanat=spm_write_sn(VF,prm);
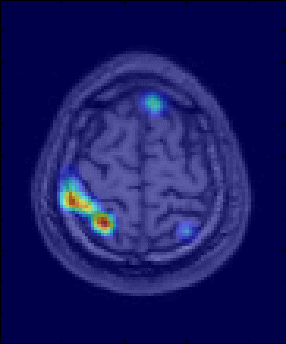
figure;imagesc(rot90(squeeze(Vfunc.dat(:,:,70))));colormap(jet);daspect([1,1,1]);
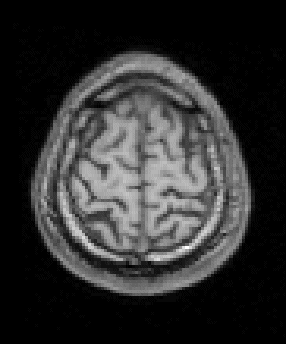
figure;imagesc(rot90(squeeze(Vanat.dat(:,:,70))));colormap(gray);daspect([1,1,1]);
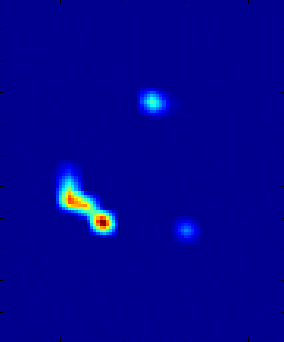
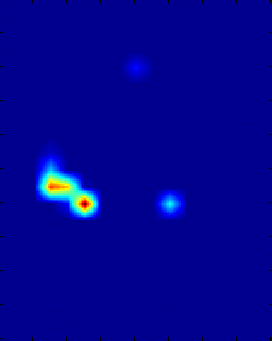
因みに標準脳化前は以下の通りです。
[filename,pathname]=uigetfile('*.img'); % 脳機能画像
VG=spm_vol(strcat(pathname,filename));
VG.dat=spm_read_vols(VG);
Vsize=(VG.dim(1:3)-1)*10+1;
x=linspace(1,VG.dim(1),Vsize(1));
y=linspace(1,VG.dim(2),Vsize(2));
z=linspace(1,VG.dim(3),Vsize(3));
[x,y,z]=ndgrid(x,y,z);
Vfunc=spm_sample_vol(VG.dat,x,y,z,1);
Vfunc=reshape(Vfunc,size(x,1),size(x,2),size(x,3));
clear x y z; % to save memory
[filename,pathname]=uigetfile('*.img'); % 脳磁図HPI座標に変換した解剖画像
Vanat=spm_read_vols(spm_vol(strcat(pathname,filename)));
figure;imagesc(rot90(squeeze(Vfunc(:,:,220))));colormap(jet);daspect([1,1,1]);
figure;imagesc(rot90(squeeze(Vanat(:,:,220))));colormap(gray);daspect([1,1,1]);
動画化しました。まず標準脳化前です。

GIF動画(2.9MB)
MPEG1動画(2.8MB)
次に標準脳化後です。
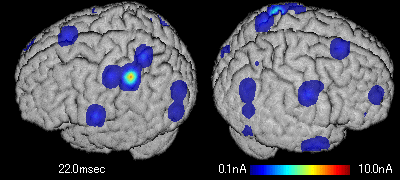
GIF動画(3.3MB)
MPEG1動画(2.9MB)